JACS:化学酶法添加修饰,提升DNA催化活性
【摘要】
南京大学于涵洋课题组与梁勇课题组合作,开发了用化学酶法在DNA上定点修饰官能团的新技术,展示了该方法在提升DNA的催化活性方面的应用。该文章以Chemoenzymatic installation of site-specific chemical groups on DNA enhances catalytic activity为题,发表在J. Am. Chem. Soc期刊上。
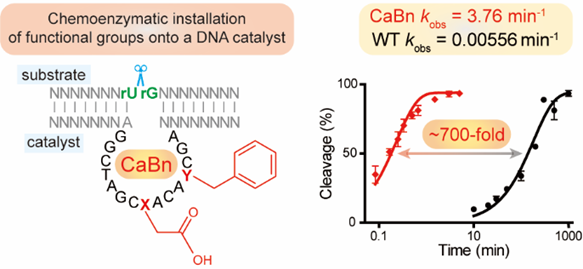
图1 化学酶法加修饰,提升DNA酶活性
【背景】
DNA能够折叠形成具有催化活性的三维结构1-3,在生物传感等领域具有广泛的应用4-7。跟基于蛋白质的酶相比,DNA酶(亦称:脱氧核酶)的催化活性较低,原因之一是DNA的化学官能团种类较少。研究者们通过引入化学修饰的方法,提升脱氧核酶的催化性能8,9;甚至使用体外筛选的技术,直接鉴定具有催化活性的非天然核酶10-12。
在DNA中引入化学修饰通常依赖于固相合成,需要制备含有目标官能团的亚磷酰胺单体。因此,作者尝试开发一种在DNA特定位点引入化学官能团的合成后修饰新方法,并使用该方法寻找具有更高催化活性的DNA酶。
【研究内容】
首先,作者设计了使用化学酶法在DNA特定位点引入化学修饰的新策略:先通过糖苷酶特异性切除非经典碱基,产生缺碱基位点;裸露出的醛基与氧胺化合物发生缩合反应,从而引入多种官能团修饰(图2)。
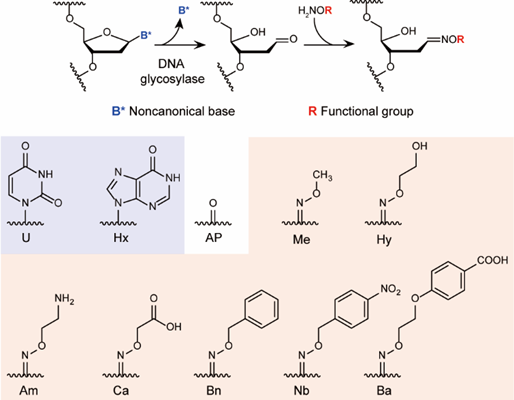
图2 使用化学酶法在DNA中定点引入多种官能团
在成功建立了化学酶法引入修饰的新策略后,作者尝试将上述不同性质的官能团引入到DNA酶10-23催化核心的不同位点,分析官能团修饰对催化活性的影响。结果表明,在8号位引入羧基(T8Ca)、在12号位引入苯环(A12Bn)有效提升了DNA酶的催化活性。作者通过组合使用两种非典型碱基-糖苷酶对,在一个DNA酶分子的8和12号位分别引入羧基和苯环,该双修饰DNA酶(CaBn)的催化活性比野生型提升了近700倍(图3)。
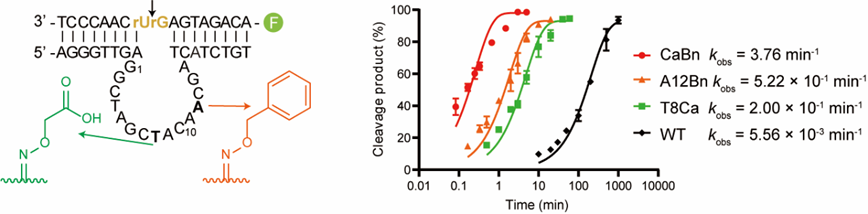
图3 化学修饰提升DNA酶的催化活性。
作者发现DNA酶CaBn对镁离子具有更强的响应性。因此,作者构建了基于CaBn的生物传感器,用于细胞内镁离子成像。在HeLa细胞中,CaBn传感器的平均荧光强度比野生型高约1.6倍。该结果表明,引入官能团修饰有效提升了DNA酶检测金属离子的灵敏度(图4)。
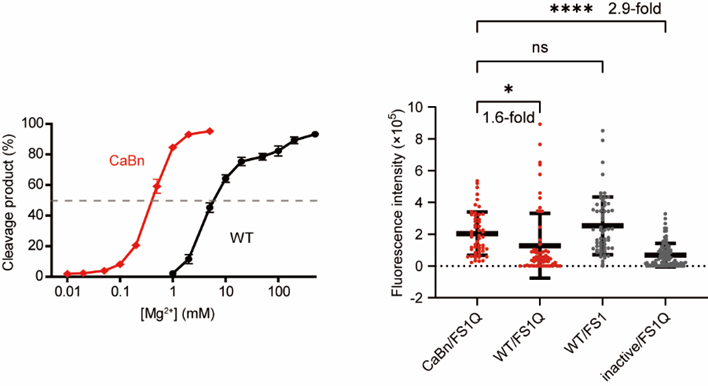
图4 双修饰的DNA酶CaBn响应更低浓度的镁离子,可用于胞内镁离子成像。
随后,作者通过分子动力学模拟,探讨了CaBn响应镁离子的机制。先前的结构解析表明,DNA酶10-23的催化构象中存在三个金属离子结合位点3。作者发现,在引入CaBn修饰后,金属离子结合位点2的镁离子分布概率增加,并且和RNA切割位点的距离变近,暗示了CaBn具有更强的镁离子招募能力,比野生型更容易折叠形成活性的催化构象(图5)。
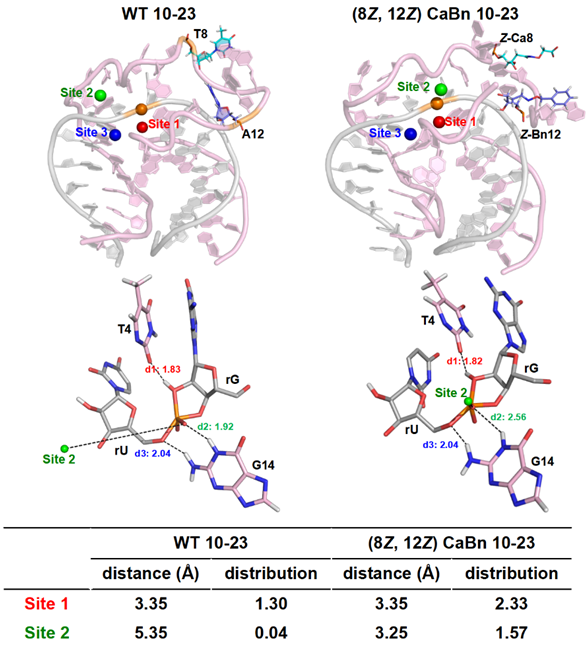
图5 DNA酶CaBn具有更强的招募镁离子的能力。
最后,作者尝试将该方法扩展到其他DNA酶上。作者发现,使用该化学酶法,既能够提升催化RNA切割反应的DNA酶(8-17)的催化活性,又能够提升具有RNA连接酶活性的DNA酶(9DB1)的催化活性(图6)。
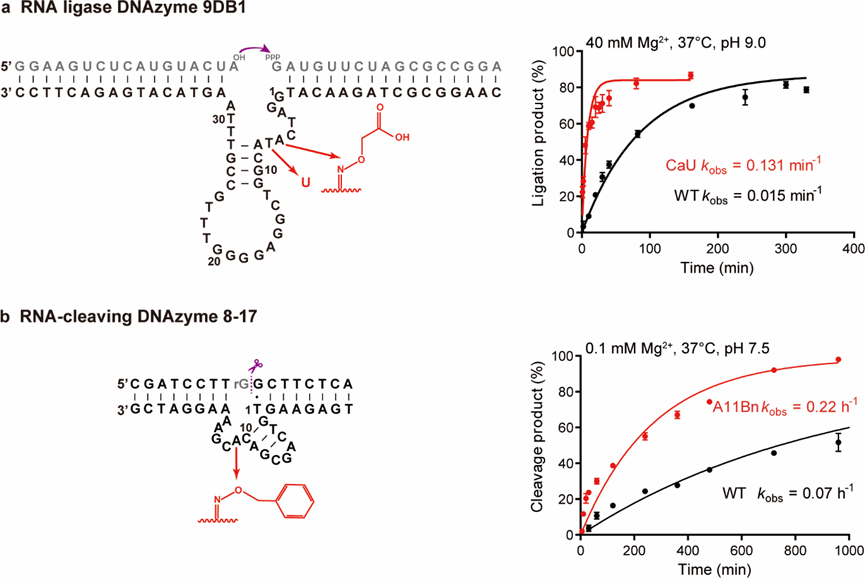
图6 化学酶法提升其他DNA酶的催化活性。
【总结】
总之,该工作开发了在DNA上引入化学修饰的新方法,鉴定了具有更高催化活性的DNA酶。
【作者信息】
文章的第一作者是张泽、魏婉清和陈思琪,文章的通讯作者是于涵洋教授和梁勇教授,杨锦滔、宋东帆、陈颖涵、赵泽润、陈佳雯、王富龙、王家欢和李喆教授对该工作亦有贡献。
【致谢】
这项工作得到了国家重点研发计划、国家自然科学基金委、江苏省自然科学基金委、中央高校基本科研业务费的支持。
【原文链接】
https://doi.org/10.1021/jacs.4c00484
【参考文献】
(1) Ponce-Salvatierra, A.; Wawrzyniak-Turek, K.; Steuerwald, U.; Hobartner, C.; Pena, V. Crystal structure of a DNA catalyst. Nature2016, 529, 231-234.
(2) Liu, H.; Yu, X.; Chen, Y.; Zhang, J.; Wu, B.; et al. Crystal structure of an RNA-cleaving DNAzyme. Nat. Commun.2017, 8, 2006.
(3) Borggrafe, J.; Victor, J.; Rosenbach, H.; Viegas, A.; Gertzen, C. G. W.; et al. Time-resolved structural analysis of an RNA-cleaving DNA catalyst. Nature2022, 601, 144-149.
(4) McGhee, C. E.; Loh, K. Y.; Lu, Y. DNAzyme sensors for detection of metal ions in the environment and imaging them in living cells. Curr. Opin. Biotechnol.2017, 45, 191-201.
(5) Zhou, W.; Saran, R.; Liu, J. Metal Sensing by DNA. Chem. Rev.2017, 117, 8272-8325.
(6) McConnell, E. M.; Cozma, I.; Mou, Q.; Brennan, J. D.; Lu, Y.; et al. Biosensing with DNAzymes. Chem. Soc. Rev.2021, 50, 8954-8994.
(7) Huang, Z.; Wang, X.; Wu, Z.; Jiang, J. H. Recent advances on DNAzyme-based sensing. Chem. Asian J.2022, 17, e202101414.
(8) McKenzie, L. K.; El-Khoury, R.; Thorpe, J. D.; Damha, M. J.; Hollenstein, M. Recent progress in non-native nucleic acid modifications. Chem. Soc. Rev.2021, 50, 5126-5164.
(9) Wang, Y.; Nguyen, K.; Spitale, R. C.; Chaput, J. C. A biologically stable DNAzyme that efficiently silences gene expression in cells. Nat. Chem.2021, 13, 319-326.
(10) Wang, Y.; Wang, Y.; Song, D.; Sun, X.; Li, Z.; et al. An RNA-cleaving threose nucleic acid enzyme capable of single point mutation discrimination. Nat. Chem.2022, 14, 350-359.
(11) Taylor, A. I.; Wan, C. J. K.; Donde, M. J.; Peak-Chew, S. Y.; Holliger, P. A modular XNAzyme cleaves long, structured RNAs under physiological conditions and enables allele-specific gene silencing. Nat. Chem.2022, 14, 1295-1305.
(12) Jerome, C. A.; Hoshika, S.; Bradley, K. M.; Benner, S. A.; Biondi, E. In vitro evolution of ribonucleases from expanded genetic alphabets. Proc. Natl. Acad. Sci. USA2022, 119, e2208261119.
如果篇首注明了授权来源,任何转载需获得来源方的许可!如果篇首未特别注明出处,本文版权属于 X-MOL ( x-mol.com ), 未经许可,谢绝转载!




























 京公网安备 11010802027423号
京公网安备 11010802027423号