单细胞水平观测基因组“暗物质”
人类基因组中,总共含有30亿对碱基,但只有极少部分能够编码成蛋白质,而接近一半的序列由转座元件(TEs)组成,TEs的插入和跳跃可以改变基因组遗传信息,增加遗传信息的多样性,是物种进化的重要原动力,对基因表达起着非常重要的调控作用。近日,生物岛实验室陈捷凯(点击查看介绍)课题组、南方科技大学Andrew Hutchins(点击查看介绍)课题组合作开发了单细胞测序数据中分析定量TEs表达的工具包scTE。
单细胞转录组测序(scRNA-seq)是研究细胞命运状态的绝佳技术,近年来,更多的单细胞测序技术以及相应的生物信息学分析方法也在不断改进,总体的目标是扩展能捕获的信息及挖掘能代表生物学功能的潜在维度。然而,目前从scRNA-seq数据分析的工具包都只定量基因的表达,分析TEs的表达还比较困难,缺乏相关研究需要的生物信息学分析的工具包,因而忽略了基因组中含量最多的TEs来源的遗传信息。
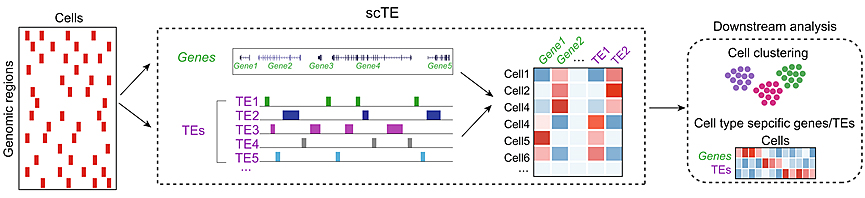
scTE原理示意图
生物岛实验室团队开发的scTE很好的弥补了上述缺憾。由于TEs为多拷贝重复序列,与常规基因不同,同一类TEs通常有成千上万个不同的拷贝遍布在全基因组,并且每个拷贝间的序列高度相似,因此,针对TEs的分析通常难以做到单位点准确定量。为解决这一问题,同时由于常规scRNA-seq只有较短的测序读长的因素,研究团队采用了针对TEs家族层面的定量策略,利用这一策略开发了从scRNA-seq数据中同时定量基因和TEs的表达的生物信息学工具包——scTE,这一策略忽略了基因组位置信息,提高了TEs的定量准确性。研究团队利用scTE,通过分析小鼠胚胎发育和人类疾病scRNA-seq数据,发现了一系列细胞命运、疾病状态特异性高表达的TEs,提示这些TEs的表达可能与胚胎发育或疾病的发生发展有关,也证明了通过scRNA-seq分析TEs的表达很有必要。
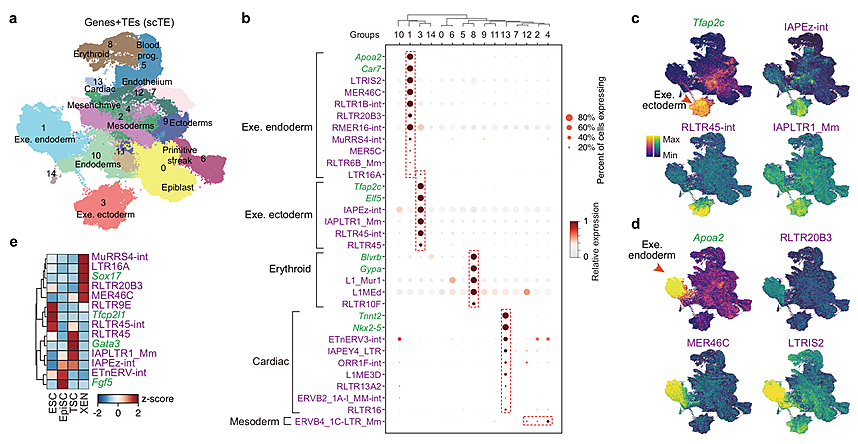
小鼠早期胚胎发育原肠作用过程中TEs细胞类型特异性表达
相对scRNA-seq研究转录组而言,单细胞ATAC-seq(scATAC-seq)等单细胞基因组技术研究的对象是染色质,染色质开放性与表观遗传修饰情况在很大程度上决定了基因表达丰度,以scATAC-seq为代表的单细胞基因组学技术的开发,让获得“高分辨率”的单细胞精度的染色质开放/修饰图谱变为可能,有利于构建从DNA到RNA再到表型的调控网络,寻找与表型强相关的核心调控因子。然而,以scATAC-seq为代表的单细胞基因组测序数据存在几个特点:(一)高维度。每个细胞中可以检测到可能开放的区域可以高达几十万个;(二)数据的稀疏性。由于技术原因,导致大量开放的区域没有检测到信号。由于以上原因,目前对scATAC-seq数据的生物信息学还存在比较大的挑战。研究团队提出由于TEs多拷贝的特点,通过累积TEs信号可以去除数据的稀疏性,降低数据的维度,或许能够有效弥补scATAC-seq数据以上两个挑战。研究团队利用scTE,发现仅利用TEs信号,能够将scATAC-seq数据中主要细胞类型区分开来,提示TEs信息对scATAC-seq等单细胞基因组数据的分析是很好的补充。
通常认为TEs被H3K9me3或DNA甲基化等机制沉默,只有在早期胚胎发育、神经细胞等特定少数细胞类型或细胞受到表观遗传药物干扰时才会被激活。通过scTE的研究,可以发现发育过程中存在大量与细胞命运转化过程高度相关的TEs家族,提示TEs参与发育相关的功能。病理情况下特定细胞TEs的表达也会发生变化,可能具有病理机制或标志物的研究前景。TEs也被发现作为增强子参与胚胎发育和免疫应答,如小鼠中转座元件RLTR13D5作为增强子参与胎盘发育,灵长类特有的转座元件MER41可以作为增强子快速响应干扰素诱导的免疫应答,因此结合scATAC-seq对TEs区域的数据信息进行挖掘是有必要的。综上,scTE能够帮助研究者将研究对象扩展到基因之外的常规研究中大量被忽视的有用的遗传信息。
这一研究成果近期发表在Nature Communications 杂志上,生物岛实验室何江平副研究员为本论文的第一作者,生物岛实验室陈捷凯研究员、南方科技大学Andrew Hutchins研究员为本论文共同通讯作者。
原文(扫描或长按二维码,识别后直达原文页面,或点此查看原文):

Identifying transposable element expression dynamics and heterogeneity during development at the single-cell level with a processing pipeline scTE
Jiangping He, Isaac A. Babarinde, Li Sun, Shuyang Xu, Ruhai Chen, Junjie Shi, Yuanjie Wei, Yuhao Li, Gang Ma, Qiang Zhuang, Andrew P. Hutchins, Jiekai Chen
Nat. Commun., 2021, 12, 1456, DOI: 10.1038/s41467-021-21808-x
导师介绍
Andrew Hutchins
https://www.x-mol.com/university/faculty/242878
陈捷凯
https://www.x-mol.com/groups/ccla_grmh-gdl
scTE地址:
https://github.com/JiekaiLab/scTE
如果篇首注明了授权来源,任何转载需获得来源方的许可!如果篇首未特别注明出处,本文版权属于 X-MOL ( x-mol.com ), 未经许可,谢绝转载!












































 京公网安备 11010802027423号
京公网安备 11010802027423号