Nature:开源!超大!药物发现平台
读者朋友们是不是觉得这个标题风格好熟悉,像极了当年某新闻网站?这次真不是本君故意要搞双叹号标题党,这篇来自美国哈佛大学Christoph Gorgulla等人的Nature 文章,就是这么个标题“An open-source drug discovery platform enables ultra-large virtual screens”。
虚拟筛选可以说得上是众多药物发现技术中备受追捧的明星了。随着蛋白结构井喷式的被解析,虚拟筛选无疑成为众多药物化学工作者手中的利器。在另一方面,合成方法学高速发展,越来越多的有机分子被创造出来,可用于进行虚拟筛选的各种分子库呈几何级数增长。不过,日益增长的数据库同计算方法的发展速度形成了尖锐的矛盾。正如Christoph Gorgulla等人的文章提到,现在一个单核处理器每对接一个配体花费的时间大约15秒,虚筛10亿个分子将耗时475年。现在可用于药物发现的有机分子已经超过1060个,现有的虚拟筛选方法急需迭代升级。Christoph Gorgulla等人开发的VirtualFlow平台解决的就是这个问题。
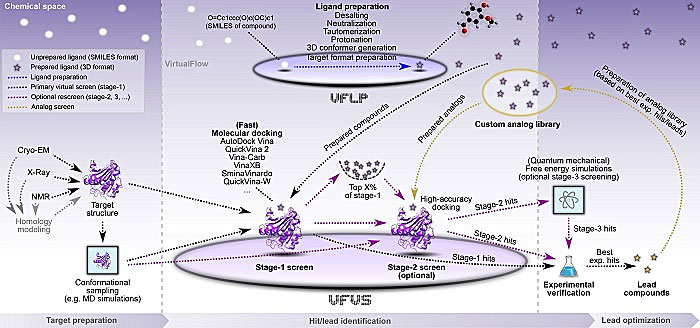
基于VirtualFlow平台的药物发现流程示意图。图片来源:Nature
简单地讲,VirtualFlow平台分为两部分,一部分用于配体准备(VirtualFlow for Ligand Preparation,VFLP),另一部分用于虚拟筛选(VirtualFlow for Virtual Screening,VFVS),VFVS模块兼容市面上大部分免费的分子对接程序,并可进行同时计算交叉验证。VirtualFlow强大的地方在于,它能整合上万个CPU来进行分子对接,还可应用云计算平台。实测结果显示,VirtualFlow使用16000个CPU对接十亿个小分子耗时约15小时。这种超大规模计算的组织架构如下图所示。
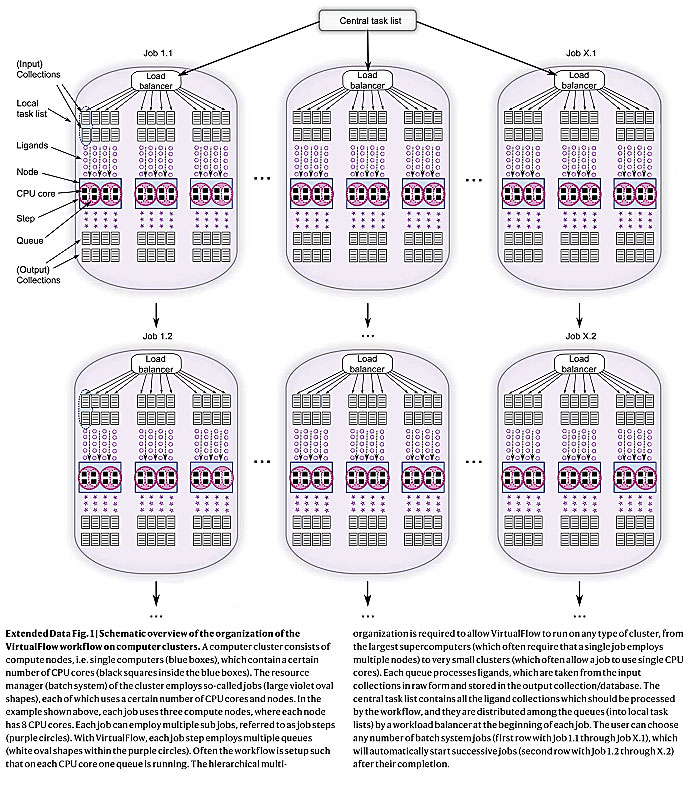
VirtualFlow的计算工作流示意图。图片来源:Nature
对虚拟筛选来说,算得快固然重要,算得准才更具应用价值。VirtualFlow平台算得是否靠谱,那必须得经过实战检验一番。为此,作者选择了肿瘤研究领域热门的KEAP1蛋白作为虚拟筛选靶点,对含有13亿配体的数据库进行了虚拟筛选。在第一阶段,对13亿分子进行刚性对接,选择打分处于前3百万的分子进入第二阶段。在第二阶段,允许小分子对KEAP1和NFR2相互作用的13个氨基酸残基进行柔性对接,筛选出大约1万个打分优秀的分子。
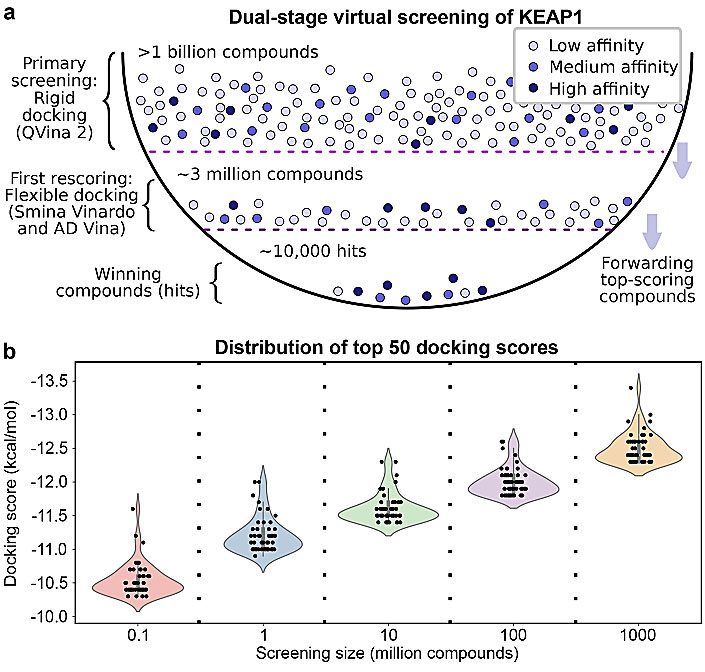
基于VirtualFlow以KEAP1为靶点的虚拟筛选流程。图片来源:Nature
作者随后从成药性、配体效率、化学多样性以及获取难度等方面在这约10000个候选分子中挑选了590个苗头化合物进行活性验证。在活性验证方面,作者选择了4种目前认可度较高的生物物理学方法,表面等离子体共振(surface plasmon resonance,SPR)、核磁共振波谱(nuclear magnetic resonance,NMR)、荧光极化(fluorescence polarization,FP)以及生物层干涉(bio-layer interferometry,BLI)。几种方法的综合应用和化合物的排除标准都在下面这张图中得以体现。
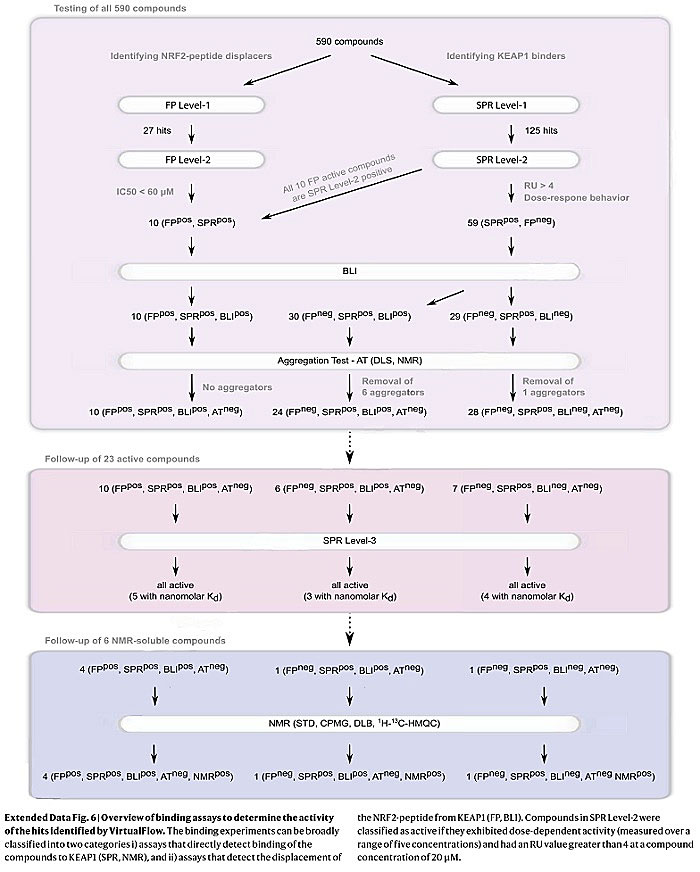
应用4种生物物理手段验证590个苗头化合物与KEAP1的相互作用。图片来源:Nature
最后作者给出了两个活性达到纳摩尔级别的代表性化合物iKEAP 1和iKEAP 2的多种实验结果。
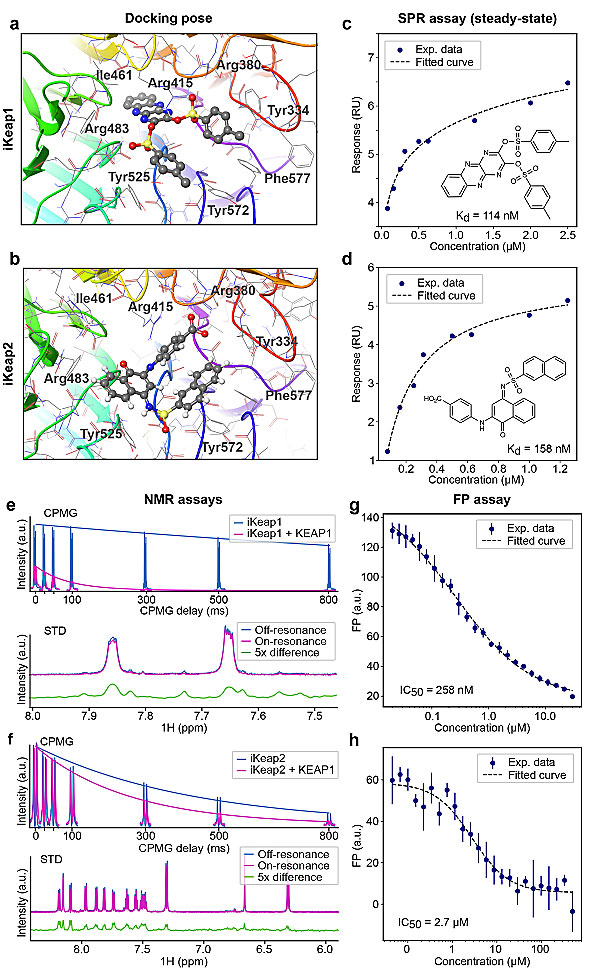
代表性化合物iKEAP 1和iKEAP 2的多种实验结果。图片来源:Nature
本君感觉这篇文章属于初看不足为奇、细思价值连城那种。你说套路吧,分子对接-活性验证的闭环没什么特别之处,高通量虚拟筛选也是很多人在做的工作。可是你细品,人家搞的这个虚拟筛选平台算力惊人,几乎是完全碾压前人,按照他们的算力,“给个靶点算遍天下有机物”指日可待。所以,本君窃以为这篇Nature 文章硬核的地方既不是精妙的科研思路,也不是优秀的实验结果,而是那能调动上万CPU进行计算的代码。虽然这代码开源 [1],但是隔行如隔山,本君同大家一样只能望码兴叹,无法领略代码之间的美感啦。
最后想说的是,更高的命中率,更快的计算速度,更强的迭代能力,虚拟筛选在药物发现的长跑途中从未掉队。
原文(扫描或长按二维码,识别后直达原文页面,或点此查看原文):

An open-source drug discovery platform enables ultra-large virtual screens
Christoph Gorgulla, Andras Boeszoermenyi, Zi-Fu Wang, Patrick D. Fischer, Paul Coote, Krishna M. Padmanabha Das, Yehor S. Malets, Dmytro S. Radchenko, Yurii S. Moroz, David A. Scott, Konstantin Fackeldey, Moritz Hoffmann, Iryna Iavniuk, Gerhard Wagner, Haribabu Arthanari
Nature, 2020, DOI: 10.1038/s41586-020-2117-z
参考资料
1. VirtualFlow platform
(本文由乐只君子供稿)
如果篇首注明了授权来源,任何转载需获得来源方的许可!如果篇首未特别注明出处,本文版权属于 X-MOL ( x-mol.com ), 未经许可,谢绝转载!












































 京公网安备 11010802027423号
京公网安备 11010802027423号