Nature子刊:看蛋白序列,预知化合物结构
提到天然来源化合物的结构预测,科研小伙伴们首先想到的是测个质谱(MS)、做个核磁(NMR)?或者再来个紫外光谱(UV)、红外光谱(IR)?“四大名谱”都凑齐了,还怕推测不出结构?再不行,养个单晶总能看清楚了吧。等一等,要做这些测试,你至少得先有纯度不影响解析结构的化合物,这就要走几轮色谱分离,颇要花费一些时日了。对于做微生物来源天然产物的小伙伴来说,有些“隐士”微生物培养都成问题,更别说取其产物了。如果能预测产物结构,那就能提前决定一株菌有没有深入做下去的价值。“需要一株好菌保平安(毕业)”,这成为微生物药物研究小伙伴的真实心声和美好期待。

这时,你需要“一双慧眼”来看清蕴藏于微生物的化合物宝藏。这双慧眼,就是基于生物序列数据库的生物信息研究工具。本文介绍的就是这样一个通过蛋白质序列信息预测化合物结构的案例。
瑞士苏黎世联邦理工学院(ETH Zürich)微生物研究所的Jörn Piel教授团队和欧洲分子生物实验室-欧洲生物信息学研究所(EMBL-EBI)的Pablo Moreno教授团队在Nature Chemical Biology 上发表论文,研究对象是细菌反式乙酰转移酶聚酮合酶(trans-acyltransferase polyketide synthases, trans-AT PKSs)及其聚酮类代谢产物。Trans-AT PKSs这类酶能产生结构复杂的聚酮类化合物,这些代谢产物具有抗菌、抗癌等多样化的生物活性,但是Trans-AT PKSs往往存在于“隐士”微生物中,研究较少,这影响了对于聚酮类化合物的进一步研究。研究者提出了一个“清奇”的想法——根据Trans-AT PKSs的序列信息来预测它们产物的化合物结构。
“工欲善其事,必先利其器”。为了发展这类聚酮产物的有效预测工具。他们分析了目前已知全部54个已鉴定出产物结构的trans-AT PKSs,发现这些体系有160个不同的模块,并且发现绝大多数模块遵循聚酮类化合物合成的共线性模式(colinear fashion)。他们进一步以系统分类方式分析54个生物合成基因簇(biosynthetic gene clusters, BGCs)的所有655个聚酮合酶(ketosynthase, KS)序列,构建“日晷式”系统进化树(图1)。这一分析揭示出大量多样化的聚酮合酶序列亚分支,涉及到起始单元、中间单元连接、多种单元化学修饰、甚至影响产物手性立体结构或区域结构(例如,L-OH或D-OH;α,β-或β,γ-双键的变化)。这些分析积累了大量预测产物结构的基础信息。

图1. 将已知trans-AT PKSs BGCs中所有KS序列分类构建“日晷式”系统分类发育树(左),对时间进行系统分类的日晷(右)。图片来源:左图来自Nat. Chem. Biol.,右图来自网络
将聚酮合酶分支与产物结构属性进行系统归属后,他们开发出trans-AT PKSs的产物预测工具TransATor。将一条连锁PKS蛋白序列以FASTA格式输入该软件,它可以注释比较每一个KS和PKS细节区域,尤其能探测出trans-AT PKSs中的涉及化学产物的结构信息,如乙酰水解酶(acyl-hydrolase, AH)、分支化(branching, B)、C3-乙酰基转移酶(C3–acyltransferase, FkbH)、羟基化后的绝对构型等信息。整个产物结构预测过程,涉及基于隐马尔采夫模型(Hidden Markov Models, HMMs)的聚酮核心区域注释和聚酮化合物结构预测,使用EMBOSS fuzzpro patterns预测化合物羟基化碳原子的绝对构型,最后用化学开发工具盒(Chemistry Development Kit, CDA)构建聚酮产物结构,其PKS共线性组合化合物的过程,类似于工厂流水化生产装配线(图2)。
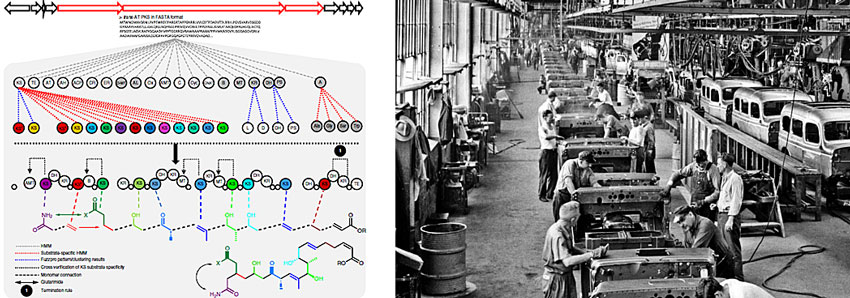
图2. TransATor预测trans-AT PKSs产物化学结构的工作流程(左),工厂生产装配线(右)。图片来源:左图来自Nat. Chem. Biol.,右图来自网络
经过一番网络验证和调试。TransATor大显身手的机会到了。他们选择一株来自海洋苔草植物根际的细菌Gynuella sunhinyii YC6258(内含未鉴定的trans-AT PKSs基因簇),利用TransATor分析其中一个trans-AT PKSs,发现此类系列有极高可能产生具有抗菌活性的tartrolon类聚酮化合物。为了验证所预测的结构,他们对Gynuella sunhinyii YC6258进行培养发酵,分离纯化,最后分离鉴定出3个tartrolon类化合物,其中1个与预测结构完全吻合,另外两个高度相似(图3)。
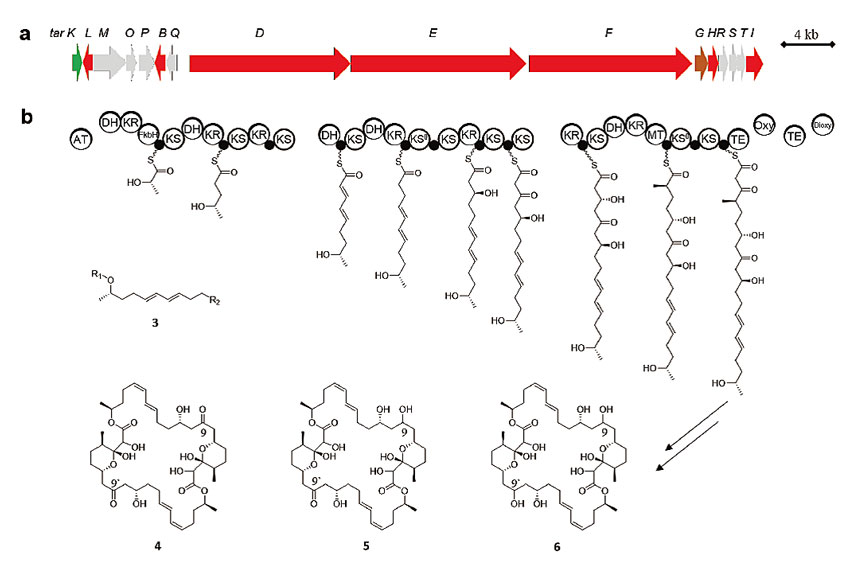
图3. 利用TransATor预测tartrolon结构与实际分离到结构比较。图片来源:Nat. Chem. Biol.
随后,他们选择菌株Leptolyngbya sp. PCC 7375的trans-AT PKSs基因簇,输入TransATor进行分析,预测出其含有的化合物类似于虾毒素phormidolide和oscillariolide,但大环的规模和结构不同,极有可能是一个新聚酮核心结构。为了验证预测结构,他们也对Leptolyngbya sp. PCC 7375培养发酵,分离纯化和结构解析,得到3个化合物分别命名为leptolyngbyalide A,B和C(图4)。由于3个化合物分离出的量有限,暂时不能进行绝对构型验证。但有趣的是,TransATor分析显示这些化合物与phormidolide类绝对构型相反。重新分析phormidolide的数据更支持TransATor预测结果,这使得phormidolide的绝对构型数据可能需要重新分析并加以修正。
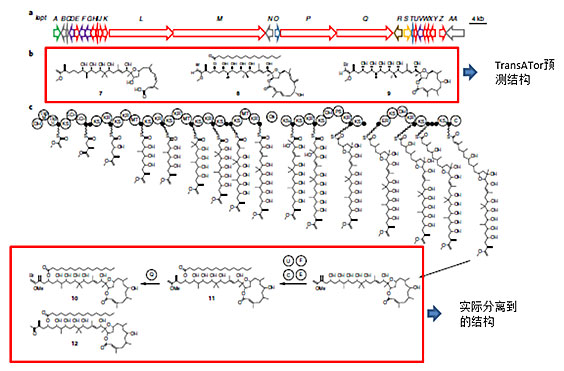
图4. 利用TransATor预测leptolyngbyalide结构与实际分离到结构比较。图片来源:Nat. Chem. Biol.
为验证TransATor能否在从未开发的细菌中发现新骨架化合物,他们选择一株分离自海绵的细菌Aquimarina sp.,将其所含trans-AT PKSs基因簇进行分析,预测出产物具有leinamycin型含硫杂环结构。经培养发酵,分离纯化,得到了两个与预测结构相吻合的化合物,其中一个化合物含硫,且都代表了trans-AT PKSs的一种新骨架类型(图5)。
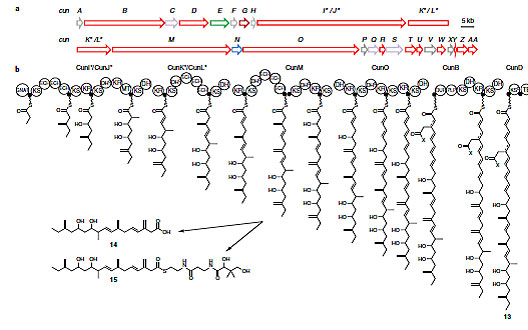
图5. TransATor预测出新骨架化合物。图片来源:Nat. Chem. Biol.
也许一株好菌里面有非常好的化合物资源,但却与我们有着“世界上最遥远的距离”——你就在面前那个小瓶子里,我却不认识你。Jörn Piel教授和Pablo Moreno教授团队基于生物信息学技术和现有大数据,开发出TransATor填补了这类空白,通过酶序列预测代谢产物的结构骨架特征,从而指导后续的靶向分离,以鉴定出结构新颖、具有生物活性的天然来源化合物。这一研究为天然来源化合物开发提供了一个新思路,或许很多人研究中“最遥远的距离”将不再是距离。
原文(扫描或长按二维码,识别后直达原文页面,或点此查看原文):

Automated structure prediction of trans-acyltransferase polyketide synthase products
Eric J. N. Helfrich, Reiko Ueoka, Alon Dolev, Michael Rust, Roy A. Meoded, Agneya Bhushan, Gianmaria Califano, Rodrigo Costa, Muriel Gugger, Christoph Steinbeck, Pablo Moreno, Jörn Piel
Nat. Chem. Biol., 2019, 15, 813–821, DOI: 10.1038/s41589-019-0313-7
导师介绍
Jörn Piel
https://www.x-mol.com/university/faculty/2833
(本文由水村山郭供稿)
如果篇首注明了授权来源,任何转载需获得来源方的许可!如果篇首未特别注明出处,本文版权属于 X-MOL ( x-mol.com ), 未经许可,谢绝转载!












































 京公网安备 11010802027423号
京公网安备 11010802027423号