指示活细胞中错误折叠的蛋白质组的双“模式”荧光探针
蛋白质组的正确折叠是保证细胞正常运作的基础。来自澳洲的科学家近日开发了一种用于指示蛋白质组是否正确折叠的荧光分子探针。蛋白质组的折叠稳态和环境极性的变化将导致该探针的荧光信号和波长位移发生变化,进而可用于标记细胞中蛋白质折叠错误的区域和定量研究。这一重要研究成果发表在Angewandte Chemie 上。
活细胞在受到压力的情况下,蛋白质的合成和折叠都会受到影响。与此同时,错误折叠的蛋白会在细胞中堆积,试图降解这些蛋白的通路会被激活。而更严重地,这些“垃圾”蛋白的碎片和聚集物在细胞质中会形成尺寸更大、毒性更高、更难以分解的颗粒。现有的研究表明,这一过程在一些神经退行性疾病和癌症的发生发展过程中起到了重要作用。来自澳大利亚La Trobe大学的Hong Yuning博士与墨尔本大学的合作者们开发了一种新型荧光“双模式”探针NTPAN-MI用于监测错误折叠蛋白聚集的过程(如图1)。实验研究表明,折叠错误的蛋白会引起其环境的极性的改变,可能是蛋白进一步发生聚集的重要推动力。
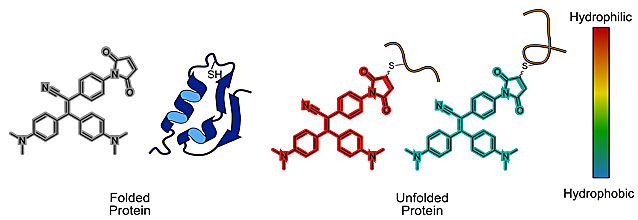
图1. 化合物NTPAN-MI的结构和与蛋白质半胱氨酸结合的作用机理。
在“模式一”下,该探针可用于感受错误折叠蛋白的存在。正确折叠的蛋白,半胱氨酸通常以二硫键形式存在以稳定蛋白结构。游离的半胱氨酸反应性较高,通常被掩盖在蛋白质的疏水核心区域。当蛋白错误折叠时,游离的半胱氨酸会暴露在蛋白质表面,进而与NTPAN-MI特异结合,使其发出荧光(图2A)。
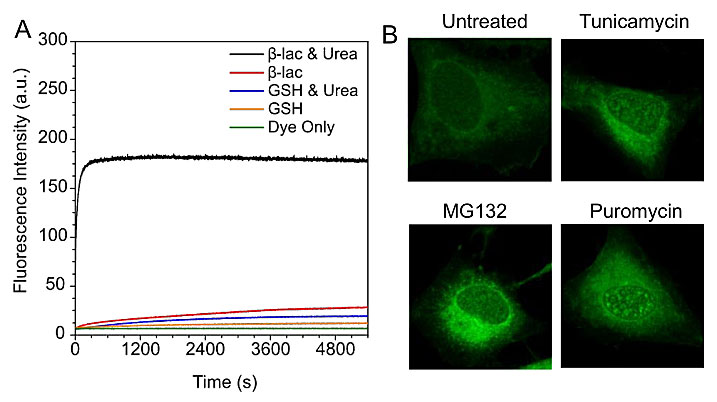
图2.(A)NTPAN-MI与变性β-乳球蛋白的巯基反应而荧光增强,而折叠态β-乳球蛋白或其他小分子生物硫醇如谷胱甘肽(GSH)反应时荧光并无明显增强。(B)不同药物处理下NTPAN-MI细胞内荧光分布。
在“模式二”下,该探针能够检测蛋白质环境的极性变化。研究者在该荧光探针上添加了一个电子对“推-拉”的化学基团(如图1)。通过测量介电常数,他们观察到,在高介电常数的极性条件下,该探针的发射光往长波方向位移。
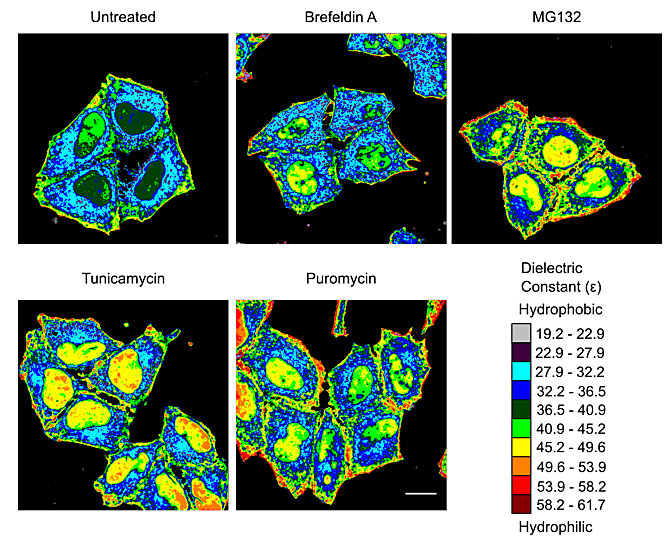
图3. 不同药物处理后细胞内介电常数分布变化。
研究者进一步将该探针用于人源细胞系中。当细胞收到外界压力,例如与药物、毒素孵育或受到病毒感染,蛋白质的合成和折叠受阻时,探针的荧光变强(如图2B)。另一方面,发射波长的位移变化表明了蛋白质的环境的极性变化。在不同的外界刺激下,亚细胞器层面的蛋白质组的极性各不相同(图3)。有趣的是,该研究发现即使是作用于细胞质的刺激源,也可引起细胞核内折叠错误的蛋白含量增加并且使其极性明显增强。
该荧光探针既能“开”“关”,又能变色,对评估蛋白质组的稳态,提供了另一个维度的测定。作者指出,他们的方法能够更加精确地测定不同细胞器中的蛋白质组对于外界压力的反应和变化,在研究错误折叠蛋白相关疾病如神经退行性疾病的机制及其治疗方法方面具有很好的应用前景。
原文(扫描或长按二维码,识别后直达原文页面,或点此查看原文):

A Molecular Chameleon for Mapping Subcellular Polarity in an Unfolded Proteome Environment
Tze Cin Owyong, Pramod Subedi, Jieru Deng, Elizabeth Hinde, Jason J. Paxman, Jonathan M. White, Weisan Chen, Begoña Heras, Wallace W. H. Wong*, Yuning Hong*
Angew. Chem. Int. Ed., 2020, DOI: 10.1002/anie.201914263
如果篇首注明了授权来源,任何转载需获得来源方的许可!如果篇首未特别注明出处,本文版权属于 X-MOL ( x-mol.com ), 未经许可,谢绝转载!












































 京公网安备 11010802027423号
京公网安备 11010802027423号