Science:结构多样的大环化合物如何发现?
环肽尤其是小环肽(含4个及以下的氨基酸残基,也称小型大环化合物)在多肽药物开发中扮演着越来越重要的角色。它们是常见的天然产物结构类型,由于α、β、γ和其他十数种氨基酸主链化学的多样性,小环肽具有丰富多样的结构,也表现出了各种各样的生物活性,例如抗真菌、抗细菌、癌细胞毒性、缓解疼痛等等。尽管近几年批准的环肽药物数量的不断增多,但这些药物基本都源自天然产物,显然当前的药物发现并没有充分利用其结构多样性。目前,使用大规模展示技术(例如mRNA展示技术)对大环化合物的化学空间进行探索也显得有些束手束脚,很大程度上局限于主要由α-氨基酸组成的空间。而多样性导向的合成技术原则上能够使用更多种类的砌块组合合成数十个至数千个规模的大环化合物库,但这些方法受到可用的化学多样性的限制。相比之下,计算方法能够快速全面地探索可能的大环空间,可以极大地促进具有独特生物活性的天然产物类化合物的发现,但现有方法同样受到可用的化学多样性的限制。举个例子,源自大名鼎鼎的AlphaFold和RoseTTAFold的生成模型仅能够设计由20个典型α-氨基酸组成的小肽。
近日,华盛顿大学David Baker教授团队报道了一种计算方法可识别由α、β、γ和其他17种氨基酸主链化学组成的有序大环化合物,并用其预测了由>42,000个单体组合组成的1490万个环肽。他们在实验室合成了18个预测为单一低能态的环肽,并测定了它们的X射线或核磁共振结构,其中15个非常接近设计模型。基于这些环肽,他们还开发了三种蛋白质靶点的选择性抑制剂以证明其药用潜力。这一工作大大开拓了易于合成的类药大环化合物的化学空间,有望加速基于结构的药物设计。相关论文发表于Science 杂志。
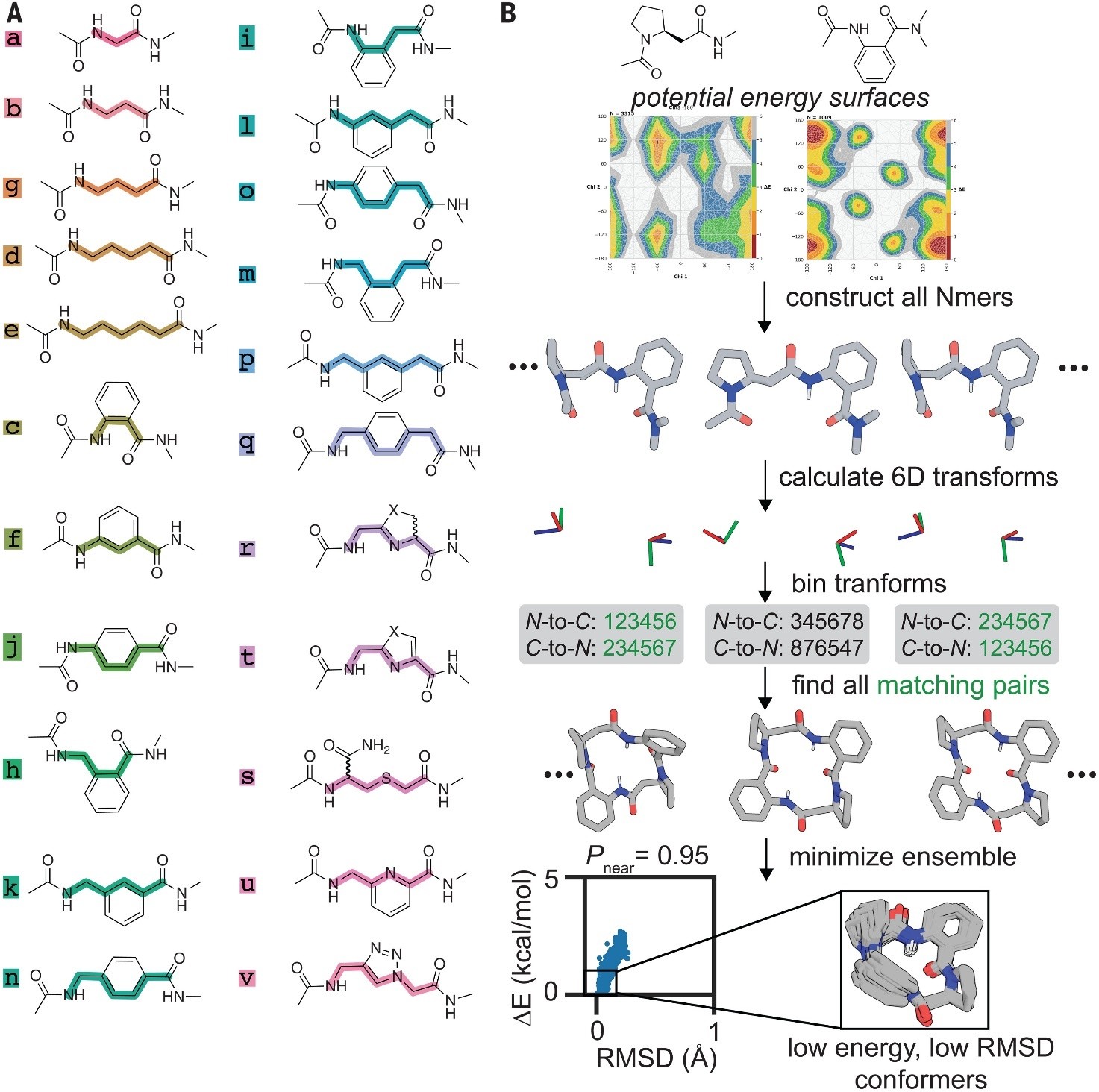
图1. 大环化合物发现方法纵览。图片来源:Science
研究者将组建环肽的氨基酸类型从常规的天然氨基酸扩展到了22种具有不同主链化学的氨基酸砌块(图1a)。如果以此为基础构建二元、三元和四元环肽,可以获得将近六万种组合。如果再在这六万种环肽主链上添加不同的侧链基团,将获得数百万种结构。这个化学空间太大了,无法使用显式构象采样来识别可以闭合成环的那一小部分线性序列。为了在这些浩如烟海的可能结构中精准高效地寻找到潜在的小环肽,研究者基于计算机算法开发了一个三步法进行搜索(图1):第一步,对每个单体氨基酸的AIMNet(Atom-Inspired Machine Learning Network)进行筛选,搜索出低能构象异构体。AIMNet通过深度神经网络模型来学习原子之间的相互作用,并将其用于预测分子结构和属性。AIMNet可以根据输入的分子结构,预测其能量、力场、电荷分布等信息。第二步,对这些单体构象及由这些单体两两组合构成的二肽构象进行刚体变换计算,并将其存储在Hash表中。第三步,通过识别Hash表中刚体变换组合接近零的条目对,以此组合形成环肽,快速而系统地生成二元、三元和四元环肽。
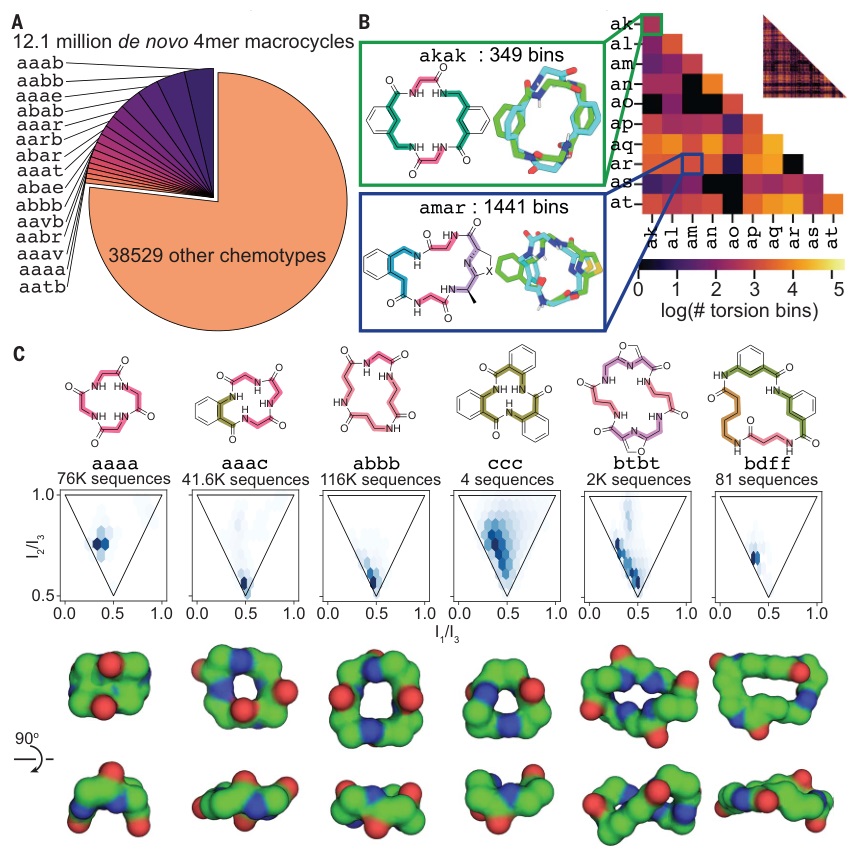
图2. 小环肽的化学和结构多样性。图片来源:Science
研究者筛选了由 130 个单体类型构建的三元和四元环肽,通过以上的三个步骤生成了1490 万个环肽结构,为9元至32元大环,包括3494个三元环肽和38544个四元环肽(图2)。考虑到环肽分子的柔性,研究者为每种分子采样了不同的 3D 结构。为了表征结构多样性,它们主链扭转角分成60°的区间,并用这些区间的字符串来表示每个大环构象。研究人员利用Hash方法来生成一组环肽结构的模型,这些模型通过低能量的单体构象组合而成。如果有足够的算力,他们可以对这些大环结构进行更精确的能量计算,了解这些大环是否有可能存在单一的最稳定结构。他们发现,利用Hash采样和计算方法,他们可以成功地预测小型环肽的结构,并且这些预测与实验结果的误差非常小。然而,对所有生成的大量环肽结构进行完整的能量计算非常耗时,因此他们选择了专注于两种类型的环肽,这些类型更有可能具有单一的最稳定结构:(1)构象可能性很少的环肽。这些环肽的结构受到分子间相互作用的影响,使得它们更可能倾向于采取少数几种特定的闭合构象。(2)存在长距离分子间氢键的环肽。对于第一类多肽,研究者从Hash表中选取了最低能态相差不到1 kcal/mol的单体作为构建环肽的氨基酸,合成了13个这样的大环肽,并使用X射线晶体衍射和核磁共振确定了其中10个的结构。结果显示,NMR结构和X射线晶体学结构都与各自的设计模型预计非常吻合。对于第二类环肽,研究者首先构建了二肽Hash表,并且包含分子间的主链氢键。他们合成了17个含有长距离氢键的环肽,其中7个能够确定高分辨率结构(3个晶体X光衍射、4个NMR)。所有7个实验确定的结构与各自的设计模型基本相符(图3)。
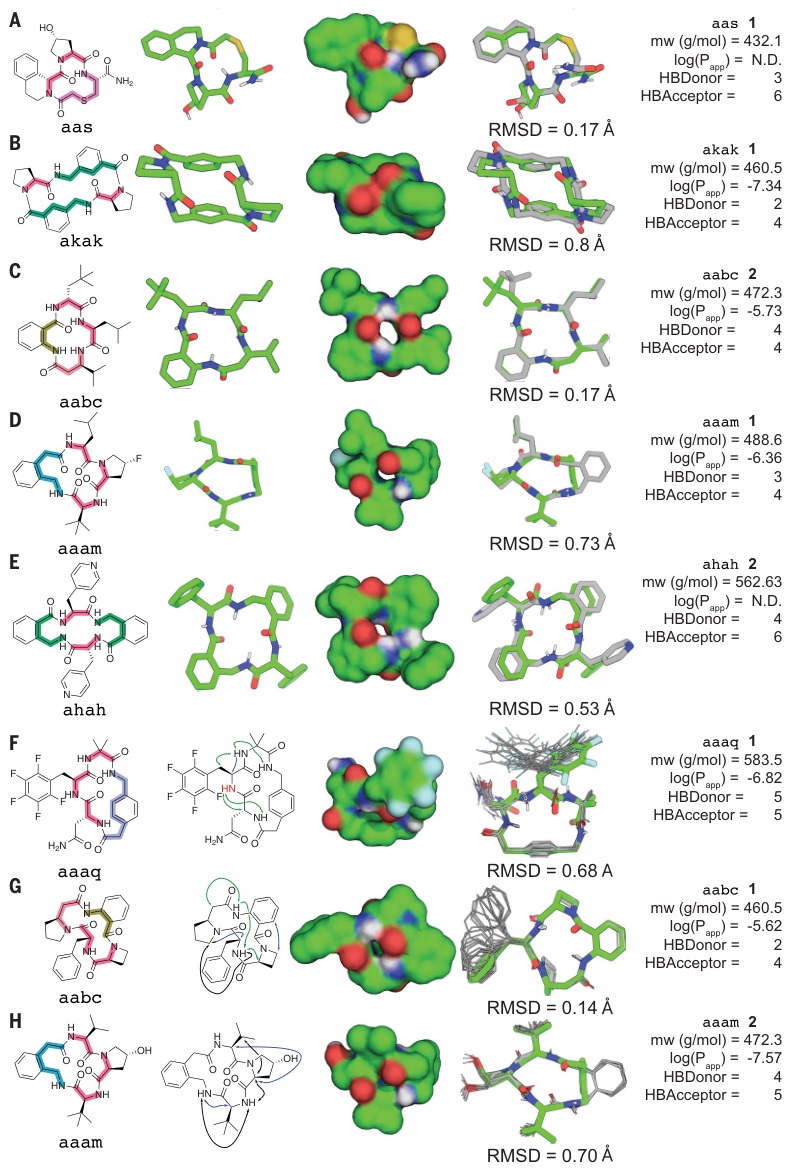
图3. 小环肽的X射线和NMR结构与设计模型非常接近。图片来源:Science
接下来,研究者使用并行人工膜渗透实验(PAMPA)测量了通过上述两种方法设计的所有29个环肽的被动膜扩散。几乎所有的环肽都具有很高的膜渗透性。那些具有暴露于外部极性基团的环肽,特别是侧链羟基和主链酰胺,具有较低的渗透性,而含有长距离分子间氢键的环肽具有更好的渗透性。此外,绝大多数的环肽对血清蛋白酶稳定。随后,为验证这些环肽的药用潜力,研究者设计了几个药物靶点的环肽抑制剂(图4),他们选定的靶标是组蛋白去乙酰化酶6(HDAC6)和SARS-CoV-2病毒的主要蛋白酶(Mpro),并设计了Bak-MCL1相互作用的环肽抑制剂。针对 HDAC6研究者合成了11个环肽抑制剂,其中10个对HDAC6的中位抑制浓度(IC50)值范围从1.5到70 nM,并且对于测试的其他HDAC蛋白质其选择性为100倍到1000倍以上。针对Mpro的6个环肽抑制剂中,只有一个属于aabi化合物类型,得到了16 μM的适中的IC50。在这个环肽的基础上,研究者又合成了27个该环肽的类似物,其中17个对Mpro的IC50值范围为4至0.88 μM。对于抑制Bak-MCL1相互作用,最强的环肽的IC50为200 nM。

图4. De Novo设计的环肽抑制剂抑制酶和蛋白质-蛋白质相互作用。图片来源:Science
结论
David Baker教授团队利用深度神经网络算法生成能量表面,并提取由不同氨基酸主链化学组成的环肽的低能构象。他们利用这些数据识别了近1500万个环肽,并通过实验合成了18个环肽来验证该方法的有效性。此后,该团队还通过开发针对两种酶以及蛋白质-蛋白质相互作用的环肽抑制剂来验证这些大环化合物的药物潜力。可以遇见,这一成果为多肽药物的开发提供了重要的工具,将极大拓展多肽药物的可能性。
原文(扫描或长按二维码,识别后直达原文页面,或点此查看原文):

Expansive discovery of chemically diverse structured macrocyclic oligoamides
Patrick J. Salveson, Adam P. Moyer, Meerit Y. Said, Gizem Gökçe, Xinting Li, Alex Kang, Hannah Nguyen, Asim K. Bera, Paul M. Levine, Gaurav Bhardwaj, David Baker*
Science, 2024, 384, 420-428. DOI: 10.1126/science.adk1687
(本文由北纬55°供稿)
如果篇首注明了授权来源,任何转载需获得来源方的许可!如果篇首未特别注明出处,本文版权属于 X-MOL ( x-mol.com ), 未经许可,谢绝转载!

































 京公网安备 11010802027423号
京公网安备 11010802027423号