Our official English website, www.x-mol.net, welcomes your
feedback! (Note: you will need to create a separate account there.)
How evolution shapes enzyme selectivity - lessons from aminoacyl-tRNA synthetases and other amino acid utilizing enzymes.
The FEBS Journal ( IF 5.5 ) Pub Date : 2020-01-20 , DOI: 10.1111/febs.15199 Dan S Tawfik 1 , Ita Gruic-Sovulj 2
The FEBS Journal ( IF 5.5 ) Pub Date : 2020-01-20 , DOI: 10.1111/febs.15199 Dan S Tawfik 1 , Ita Gruic-Sovulj 2
Affiliation
|
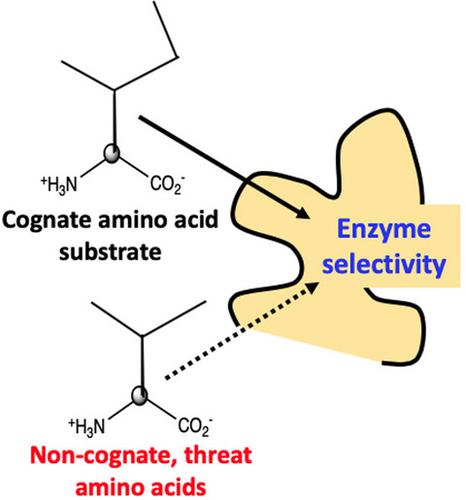
Aminoacyl-tRNA synthetases (AARSs) charge tRNA with their cognate amino acids. Many other enzymes use amino acids as substrates, yet discrimination against noncognate amino acids that threaten the accuracy of protein translation is a hallmark of AARSs. Comparing AARSs to these other enzymes allowed us to recognize patterns in molecular recognition and strategies used by evolution for exercising selectivity. Overall, AARSs are 2-3 orders of magnitude more selective than most other amino acid utilizing enzymes. AARSs also reveal the physicochemical limits of molecular discrimination. For example, amino acids smaller by a single methyl moiety present a discrimination ceiling of ~200, while larger ones can be discriminated by up to 105 -fold. In contrast, substrates larger by a hydroxyl group challenge AARS selectivity, due to promiscuous H-bonding with polar active site groups. This 'hydroxyl paradox' is resolved by editing. Indeed, when the physicochemical discrimination limits are reached, post-transfer editing - hydrolysis of tRNAs charged with noncognate amino acids, evolved. The editing site often selectively recognizes the edited noncognate substrate using the very same feature that the synthetic site could not efficiently discriminate against. Finally, the comparison to other enzymes also reveals that the selectivity of AARSs is an explicitly evolved trait, showing some clear examples of how selection acted not only to optimize catalytic efficiency with the target substrate, but also to abolish activity with noncognate threat substrates ('negative selection').
中文翻译:

进化如何影响酶的选择性-氨酰基-tRNA合成酶和其他利用氨基酸的酶的教训。
氨酰基-tRNA合成酶(AARS)将tRNA及其同源氨基酸带入电荷。许多其他酶都使用氨基酸作为底物,但对威胁蛋白质翻译准确性的非同源氨基酸的区分是AARS的标志。将AARS与这些其他酶进行比较,使我们能够识别分子识别中的模式以及进化过程中用于行使选择性的策略。总体而言,AARS的选择性比大多数其他利用氨基酸的酶高2-3个数量级。AARSs还揭示了分子鉴别的物理化学极限。例如,单个甲基部分较小的氨基酸的鉴别上限约为200,而较大的氨基酸最多可以区分105倍。相比之下,羟基大的底物会挑战ARS的选择性,由于与极性活性位点基团混杂的H键。通过编辑解决了这个“羟基悖论”。确实,当达到理化辨别极限时,就会产生转移后编辑-带有非同源氨基酸的tRNA水解。编辑位点通常使用合成位点无法有效区分的特征来选择性地识别已编辑的非同源底物。最后,与其他酶的比较还表明,AARS的选择性是一个显着进化的性状,显示了一些清楚的例子说明了选择如何不仅可以优化目标底物的催化效率,而且还可以消除非同源威胁底物的活性('否定选择”)。通过编辑解决。确实,当达到理化辨别极限时,就会产生转移后编辑-带有非同源氨基酸的tRNA水解。编辑位点通常使用合成位点无法有效区分的特征来选择性地识别已编辑的非同源底物。最后,与其他酶的比较还表明,AARS的选择性是一个显着进化的性状,显示了一些清楚的例子说明了选择如何不仅可以优化目标底物的催化效率,而且还可以消除非同源威胁底物的活性('否定选择”)。通过编辑解决。确实,当达到理化辨别极限时,就会产生转移后编辑-带有非同源氨基酸的tRNA水解。编辑位点通常使用合成位点无法有效区分的非常相同的特征来选择性地识别已编辑的非同源底物。最后,与其他酶的比较还表明,AARS的选择性是一个显着进化的性状,显示了一些清楚的例子说明了选择如何不仅可以优化目标底物的催化效率,而且还可以消除非同源威胁底物的活性('否定选择”)。编辑位点通常使用合成位点无法有效区分的特征来选择性地识别已编辑的非同源底物。最后,与其他酶的比较还表明,AARS的选择性是一个显着进化的性状,显示了一些清楚的例子说明了选择如何不仅可以优化目标底物的催化效率,而且还可以消除非同源威胁底物的活性('否定选择”)。编辑位点通常使用合成位点无法有效区分的特征来选择性地识别已编辑的非同源底物。最后,与其他酶的比较还表明,AARS的选择性是一个显着进化的性状,显示了一些清楚的例子说明了选择如何不仅可以优化目标底物的催化效率,而且还可以消除非同源威胁底物的活性('否定选择”)。
更新日期:2020-04-06
中文翻译:

进化如何影响酶的选择性-氨酰基-tRNA合成酶和其他利用氨基酸的酶的教训。
氨酰基-tRNA合成酶(AARS)将tRNA及其同源氨基酸带入电荷。许多其他酶都使用氨基酸作为底物,但对威胁蛋白质翻译准确性的非同源氨基酸的区分是AARS的标志。将AARS与这些其他酶进行比较,使我们能够识别分子识别中的模式以及进化过程中用于行使选择性的策略。总体而言,AARS的选择性比大多数其他利用氨基酸的酶高2-3个数量级。AARSs还揭示了分子鉴别的物理化学极限。例如,单个甲基部分较小的氨基酸的鉴别上限约为200,而较大的氨基酸最多可以区分105倍。相比之下,羟基大的底物会挑战ARS的选择性,由于与极性活性位点基团混杂的H键。通过编辑解决了这个“羟基悖论”。确实,当达到理化辨别极限时,就会产生转移后编辑-带有非同源氨基酸的tRNA水解。编辑位点通常使用合成位点无法有效区分的特征来选择性地识别已编辑的非同源底物。最后,与其他酶的比较还表明,AARS的选择性是一个显着进化的性状,显示了一些清楚的例子说明了选择如何不仅可以优化目标底物的催化效率,而且还可以消除非同源威胁底物的活性('否定选择”)。通过编辑解决。确实,当达到理化辨别极限时,就会产生转移后编辑-带有非同源氨基酸的tRNA水解。编辑位点通常使用合成位点无法有效区分的特征来选择性地识别已编辑的非同源底物。最后,与其他酶的比较还表明,AARS的选择性是一个显着进化的性状,显示了一些清楚的例子说明了选择如何不仅可以优化目标底物的催化效率,而且还可以消除非同源威胁底物的活性('否定选择”)。通过编辑解决。确实,当达到理化辨别极限时,就会产生转移后编辑-带有非同源氨基酸的tRNA水解。编辑位点通常使用合成位点无法有效区分的非常相同的特征来选择性地识别已编辑的非同源底物。最后,与其他酶的比较还表明,AARS的选择性是一个显着进化的性状,显示了一些清楚的例子说明了选择如何不仅可以优化目标底物的催化效率,而且还可以消除非同源威胁底物的活性('否定选择”)。编辑位点通常使用合成位点无法有效区分的特征来选择性地识别已编辑的非同源底物。最后,与其他酶的比较还表明,AARS的选择性是一个显着进化的性状,显示了一些清楚的例子说明了选择如何不仅可以优化目标底物的催化效率,而且还可以消除非同源威胁底物的活性('否定选择”)。编辑位点通常使用合成位点无法有效区分的特征来选择性地识别已编辑的非同源底物。最后,与其他酶的比较还表明,AARS的选择性是一个显着进化的性状,显示了一些清楚的例子说明了选择如何不仅可以优化目标底物的催化效率,而且还可以消除非同源威胁底物的活性('否定选择”)。











































 京公网安备 11010802027423号
京公网安备 11010802027423号