当前位置:
X-MOL 学术
›
Mol. Phylogenet. Evol.
›
论文详情
Our official English website, www.x-mol.net, welcomes your
feedback! (Note: you will need to create a separate account there.)
Conservation and innovation: Plastome evolution during rapid radiation of Rhodiola on the Qinghai-Tibetan Plateau.
Molecular Phylogenetics and Evolution ( IF 3.6 ) Pub Date : 2019-12-18 , DOI: 10.1016/j.ympev.2019.106713 Dan-Ni Zhao 1 , Yi Ren 1 , Jian-Qiang Zhang 1
Molecular Phylogenetics and Evolution ( IF 3.6 ) Pub Date : 2019-12-18 , DOI: 10.1016/j.ympev.2019.106713 Dan-Ni Zhao 1 , Yi Ren 1 , Jian-Qiang Zhang 1
Affiliation
|
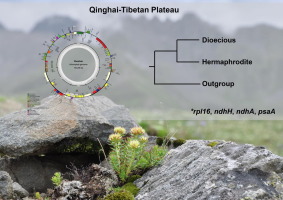
The amount of plastome sequence data available has soared in the last decade, but the nature of plastome evolution during rapid radiations is largely unknown. Moreover, although there is increasing evidence showing that plastomes may have undergone adaptive evolution in order to allow adaptation to various environments, few studies have systematically investigated the role of the plastome in alpine adaptation. To address these questions, we sequenced and analyzed 12 representative species of Rhodiola, a genus which includes ca. 70 perennial herbs mainly growing in alpine habitats in the Qinghai-Tibet Plateau and the Hengduan Mountains. Rapid radiation in this genus was triggered by the uplift of the Qinghai-Tibet Plateau. We also included nine species of Crassulaceae as the outgroups. All plastomes were conserved with respect to size, structure, and gene content and order, with few variations: each contained 134 genes, including 85 protein-coding genes, 37 tRNAs, 8 rRNAs, and 4 potential pseudogenes. Four types of repeat sequence were detected. Slight contraction and expansion of the inverted repeats were also revealed. Both the genome-wide alignment and sequence polymorphism analyses showed that the inverted repeats and coding regions were more conserved than the single-copy regions and the non-coding regions. Positive selection analyses identified three genes containing sites of positive selection (rpl16, ndhA, ndhH), and one gene with a faster than average rate of evolution (psaA). The products of these genes may be involved in the adaptation of Rhodiola to alpine environments such as low CO2 concentration and high-intensity light.
中文翻译:

保护与创新:青藏高原红景天快速辐射过程中的塑性组演化。
在过去的十年中,可塑质体序列数据的数量猛增,但是在快速辐射过程中质体质体进化的本质很大程度上是未知的。此外,尽管越来越多的证据表明质体组可能已经经历了适应性进化,以适应各种环境,但是很少有研究系统地研究了质体组在高山适应中的作用。为了解决这些问题,我们对12个具有代表性的红景天属进行了测序和分析,其中的一个属包括红景天。70种多年生草本植物,主要生长在青藏高原和横断山的高山生境中。该属的快速辐射是由青藏高原的隆升引起的。我们还包括了景天科的九种物种。所有质体在大小,结构,以及基因的内容和顺序,几乎没有变化:每个包含134个基因,包括85个蛋白质编码基因,37个tRNA,8个rRNA和4个潜在的假基因。检测到四种类型的重复序列。还揭示了反向重复序列的轻微收缩和扩增。全基因组比对和序列多态性分析均显示,反向重复和编码区比单拷贝区和非编码区更保守。阳性选择分析确定了三个包含阳性选择位点的基因(rpl16,ndhA,ndhH),以及一个具有比平均进化速率快的基因(psaA)。这些基因的产物可能参与了红景天对高山环境的适应,例如低二氧化碳浓度和高强度光照。每个基因包含134个基因,其中包括85个蛋白质编码基因,37个tRNA,8个rRNA和4个潜在的假基因。检测到四种类型的重复序列。还揭示了反向重复序列的轻微收缩和扩增。全基因组比对和序列多态性分析均显示,反向重复和编码区比单拷贝区和非编码区更保守。阳性选择分析确定了三个包含阳性选择位点的基因(rpl16,ndhA,ndhH),以及一个具有比平均进化速率快的基因(psaA)。这些基因的产物可能参与了红景天对高山环境的适应,例如低二氧化碳浓度和高强度光照。每个基因包含134个基因,其中包括85个蛋白质编码基因,37个tRNA,8个rRNA和4个潜在的假基因。检测到四种类型的重复序列。还揭示了反向重复序列的轻微收缩和扩增。全基因组比对和序列多态性分析均显示,反向重复和编码区比单拷贝区和非编码区更保守。阳性选择分析确定了三个包含阳性选择位点的基因(rpl16,ndhA,ndhH),以及一个具有比平均进化速率快的基因(psaA)。这些基因的产物可能参与了红景天对高山环境的适应,例如低二氧化碳浓度和高强度光照。检测到四种类型的重复序列。还揭示了反向重复序列的轻微收缩和扩增。全基因组比对和序列多态性分析均显示,反向重复和编码区比单拷贝区和非编码区更保守。阳性选择分析确定了三个包含阳性选择位点的基因(rpl16,ndhA,ndhH),以及一个具有比平均进化速率快的基因(psaA)。这些基因的产物可能参与了红景天对高山环境的适应,例如低二氧化碳浓度和高强度光照。检测到四种类型的重复序列。还揭示了反向重复序列的轻微收缩和扩增。全基因组比对和序列多态性分析均显示,反向重复和编码区比单拷贝区和非编码区更保守。阳性选择分析确定了三个包含阳性选择位点的基因(rpl16,ndhA,ndhH),以及一个具有比平均进化速率快的基因(psaA)。这些基因的产物可能参与了红景天对高山环境的适应,例如低二氧化碳浓度和高强度光照。全基因组比对和序列多态性分析均显示,反向重复和编码区比单拷贝区和非编码区更保守。阳性选择分析确定了三个包含阳性选择位点的基因(rpl16,ndhA,ndhH),以及一个具有比平均进化速率快的基因(psaA)。这些基因的产物可能参与了红景天对高山环境的适应,例如低二氧化碳浓度和高强度光照。全基因组比对和序列多态性分析均显示,反向重复和编码区比单拷贝区和非编码区更保守。阳性选择分析确定了三个包含阳性选择位点的基因(rpl16,ndhA,ndhH),以及一个具有比平均进化速率快的基因(psaA)。这些基因的产物可能参与了红景天对高山环境的适应,例如低二氧化碳浓度和高强度光照。
更新日期:2019-12-19
中文翻译:

保护与创新:青藏高原红景天快速辐射过程中的塑性组演化。
在过去的十年中,可塑质体序列数据的数量猛增,但是在快速辐射过程中质体质体进化的本质很大程度上是未知的。此外,尽管越来越多的证据表明质体组可能已经经历了适应性进化,以适应各种环境,但是很少有研究系统地研究了质体组在高山适应中的作用。为了解决这些问题,我们对12个具有代表性的红景天属进行了测序和分析,其中的一个属包括红景天。70种多年生草本植物,主要生长在青藏高原和横断山的高山生境中。该属的快速辐射是由青藏高原的隆升引起的。我们还包括了景天科的九种物种。所有质体在大小,结构,以及基因的内容和顺序,几乎没有变化:每个包含134个基因,包括85个蛋白质编码基因,37个tRNA,8个rRNA和4个潜在的假基因。检测到四种类型的重复序列。还揭示了反向重复序列的轻微收缩和扩增。全基因组比对和序列多态性分析均显示,反向重复和编码区比单拷贝区和非编码区更保守。阳性选择分析确定了三个包含阳性选择位点的基因(rpl16,ndhA,ndhH),以及一个具有比平均进化速率快的基因(psaA)。这些基因的产物可能参与了红景天对高山环境的适应,例如低二氧化碳浓度和高强度光照。每个基因包含134个基因,其中包括85个蛋白质编码基因,37个tRNA,8个rRNA和4个潜在的假基因。检测到四种类型的重复序列。还揭示了反向重复序列的轻微收缩和扩增。全基因组比对和序列多态性分析均显示,反向重复和编码区比单拷贝区和非编码区更保守。阳性选择分析确定了三个包含阳性选择位点的基因(rpl16,ndhA,ndhH),以及一个具有比平均进化速率快的基因(psaA)。这些基因的产物可能参与了红景天对高山环境的适应,例如低二氧化碳浓度和高强度光照。每个基因包含134个基因,其中包括85个蛋白质编码基因,37个tRNA,8个rRNA和4个潜在的假基因。检测到四种类型的重复序列。还揭示了反向重复序列的轻微收缩和扩增。全基因组比对和序列多态性分析均显示,反向重复和编码区比单拷贝区和非编码区更保守。阳性选择分析确定了三个包含阳性选择位点的基因(rpl16,ndhA,ndhH),以及一个具有比平均进化速率快的基因(psaA)。这些基因的产物可能参与了红景天对高山环境的适应,例如低二氧化碳浓度和高强度光照。检测到四种类型的重复序列。还揭示了反向重复序列的轻微收缩和扩增。全基因组比对和序列多态性分析均显示,反向重复和编码区比单拷贝区和非编码区更保守。阳性选择分析确定了三个包含阳性选择位点的基因(rpl16,ndhA,ndhH),以及一个具有比平均进化速率快的基因(psaA)。这些基因的产物可能参与了红景天对高山环境的适应,例如低二氧化碳浓度和高强度光照。检测到四种类型的重复序列。还揭示了反向重复序列的轻微收缩和扩增。全基因组比对和序列多态性分析均显示,反向重复和编码区比单拷贝区和非编码区更保守。阳性选择分析确定了三个包含阳性选择位点的基因(rpl16,ndhA,ndhH),以及一个具有比平均进化速率快的基因(psaA)。这些基因的产物可能参与了红景天对高山环境的适应,例如低二氧化碳浓度和高强度光照。全基因组比对和序列多态性分析均显示,反向重复和编码区比单拷贝区和非编码区更保守。阳性选择分析确定了三个包含阳性选择位点的基因(rpl16,ndhA,ndhH),以及一个具有比平均进化速率快的基因(psaA)。这些基因的产物可能参与了红景天对高山环境的适应,例如低二氧化碳浓度和高强度光照。全基因组比对和序列多态性分析均显示,反向重复和编码区比单拷贝区和非编码区更保守。阳性选择分析确定了三个包含阳性选择位点的基因(rpl16,ndhA,ndhH),以及一个具有比平均进化速率快的基因(psaA)。这些基因的产物可能参与了红景天对高山环境的适应,例如低二氧化碳浓度和高强度光照。











































 京公网安备 11010802027423号
京公网安备 11010802027423号