当前位置:
X-MOL 学术
›
Soft Matter
›
论文详情
Our official English website, www.x-mol.net, welcomes your
feedback! (Note: you will need to create a separate account there.)
A lattice kinetic Monte-Carlo method for simulating chromosomal dynamics and other (non-)equilibrium bio-assemblies.
Soft Matter ( IF 2.9 ) Pub Date : 2019-11-22 , DOI: 10.1039/c9sm01835b Christiaan A Miermans 1 , Chase P Broedersz
Soft Matter ( IF 2.9 ) Pub Date : 2019-11-22 , DOI: 10.1039/c9sm01835b Christiaan A Miermans 1 , Chase P Broedersz
Affiliation
|
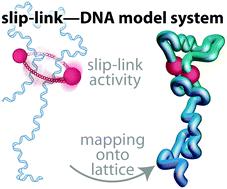
Biological assemblies in living cells such as chromosomes constitute large many-body systems that operate in a fluctuating, out-of-equilibrium environment. Since a brute-force simulation of that many degrees of freedom is currently computationally unfeasible, it is necessary to perform coarse-grained stochastic simulations. Here, we develop all tools necessary to write a lattice kinetic Monte-Carlo (LKMC) algorithm capable of performing such simulations. We discuss the validity and limits of this approach by testing the results of the simulation method in simple settings. Importantly, we illustrate how at large external forces Metropolis–Hastings kinetics violate the fluctuation–dissipation and steady-state fluctuation theorems and discuss better alternatives. Although this simulation framework is rather general, we demonstrate our approach using a DNA polymer with interacting SMC condensin loop-extruding enzymes. Specifically, we show that the scaling behavior of the loop-size distributions that we obtain in our LKMC simulations of this SMC–DNA system is consistent with that reported in other studies using Brownian dynamics simulations and analytic approaches. Moreover, we find that the irreversible dynamics of these enzymes under certain conditions result in frozen, sterically jammed polymer configurations, highlighting a potential pitfall of this approach.
中文翻译:

一种用于模拟染色体动力学和其他(非)平衡生物组装体的晶格动力学蒙特卡洛方法。
诸如染色体之类的活细胞中的生物集合体构成了庞大的多体系统,这些系统在波动的,不平衡的环境中运行。由于目前尚无法在如此大的自由度下进行强力模拟,因此有必要进行粗粒度的随机模拟。在这里,我们开发了所有必要的工具,以编写能够执行此类模拟的晶格动力学蒙特卡洛(LKMC)算法。通过在简单的设置中测试模拟方法的结果,我们讨论了这种方法的有效性和局限性。重要的是,我们说明了在大的外力作用下大都市-哈斯特斯动力学如何违反波动-耗散和稳态波动定理,并讨论了更好的选择。尽管此模拟框架相当笼统,我们展示了使用具有相互作用的SMC凝缩素环挤出酶的DNA聚合物的方法。特别是,我们表明,在我们的SMC-DNA系统的LKMC模拟中获得的环尺寸分布的缩放行为与使用布朗动力学模拟和分析方法进行的其他研究所报道的一致。此外,我们发现这些酶在某些条件下的不可逆动力学会导致冻结,空间上受阻的聚合物构型,从而突出了这种方法的潜在陷阱。我们表明,在我们的SMC-DNA系统的LKMC模拟中获得的环尺寸分布的缩放行为与使用布朗动力学模拟和分析方法进行的其他研究报告的一致。此外,我们发现这些酶在某些条件下的不可逆动力学会导致冻结,空间上受阻的聚合物构型,从而凸显了这种方法的潜在缺陷。我们表明,在我们的SMC-DNA系统的LKMC模拟中获得的环尺寸分布的缩放行为与使用布朗动力学模拟和分析方法进行的其他研究报告的一致。此外,我们发现这些酶在某些条件下的不可逆动力学会导致冻结,空间上受阻的聚合物构型,从而突出了这种方法的潜在陷阱。
更新日期:2019-11-22
中文翻译:

一种用于模拟染色体动力学和其他(非)平衡生物组装体的晶格动力学蒙特卡洛方法。
诸如染色体之类的活细胞中的生物集合体构成了庞大的多体系统,这些系统在波动的,不平衡的环境中运行。由于目前尚无法在如此大的自由度下进行强力模拟,因此有必要进行粗粒度的随机模拟。在这里,我们开发了所有必要的工具,以编写能够执行此类模拟的晶格动力学蒙特卡洛(LKMC)算法。通过在简单的设置中测试模拟方法的结果,我们讨论了这种方法的有效性和局限性。重要的是,我们说明了在大的外力作用下大都市-哈斯特斯动力学如何违反波动-耗散和稳态波动定理,并讨论了更好的选择。尽管此模拟框架相当笼统,我们展示了使用具有相互作用的SMC凝缩素环挤出酶的DNA聚合物的方法。特别是,我们表明,在我们的SMC-DNA系统的LKMC模拟中获得的环尺寸分布的缩放行为与使用布朗动力学模拟和分析方法进行的其他研究所报道的一致。此外,我们发现这些酶在某些条件下的不可逆动力学会导致冻结,空间上受阻的聚合物构型,从而突出了这种方法的潜在陷阱。我们表明,在我们的SMC-DNA系统的LKMC模拟中获得的环尺寸分布的缩放行为与使用布朗动力学模拟和分析方法进行的其他研究报告的一致。此外,我们发现这些酶在某些条件下的不可逆动力学会导致冻结,空间上受阻的聚合物构型,从而凸显了这种方法的潜在缺陷。我们表明,在我们的SMC-DNA系统的LKMC模拟中获得的环尺寸分布的缩放行为与使用布朗动力学模拟和分析方法进行的其他研究报告的一致。此外,我们发现这些酶在某些条件下的不可逆动力学会导致冻结,空间上受阻的聚合物构型,从而突出了这种方法的潜在陷阱。











































 京公网安备 11010802027423号
京公网安备 11010802027423号