Our official English website, www.x-mol.net, welcomes your
feedback! (Note: you will need to create a separate account there.)
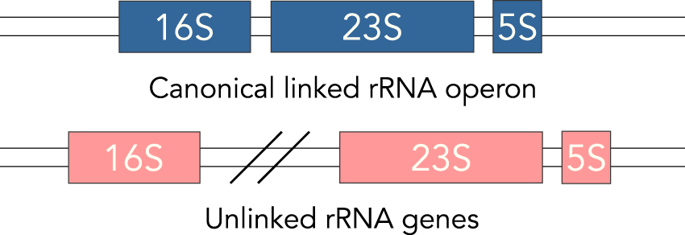
Unlinked rRNA genes are widespread among bacteria and archaea.
The ISME Journal ( IF 10.8 ) Pub Date : 2019-11-11 , DOI: 10.1038/s41396-019-0552-3 Tess E Brewer 1, 2 , Mads Albertsen 3 , Arwyn Edwards 4 , Rasmus H Kirkegaard 3 , Eduardo P C Rocha 5 , Noah Fierer 1, 6
The ISME Journal ( IF 10.8 ) Pub Date : 2019-11-11 , DOI: 10.1038/s41396-019-0552-3 Tess E Brewer 1, 2 , Mads Albertsen 3 , Arwyn Edwards 4 , Rasmus H Kirkegaard 3 , Eduardo P C Rocha 5 , Noah Fierer 1, 6
Affiliation
|
Ribosomes are essential to cellular life and the genes for their RNA components are the most conserved and transcribed genes in bacteria and archaea. Ribosomal RNA genes are typically organized into a single operon, an arrangement thought to facilitate gene regulation. In reality, some bacteria and archaea do not share this canonical rRNA arrangement-their 16S and 23S rRNA genes are separated across the genome and referred to as "unlinked". This rearrangement has previously been treated as an anomaly or a byproduct of genome degradation in intracellular bacteria. Here, we leverage complete genome and long-read metagenomic data to show that unlinked 16S and 23S rRNA genes are more common than previously thought. Unlinked rRNA genes occur in many phyla, most significantly within Deinococcus-Thermus, Chloroflexi, and Planctomycetes, and occur in differential frequencies across natural environments. We found that up to 41% of rRNA genes in soil were unlinked, in contrast to the human gut, where all sequenced rRNA genes were linked. The frequency of unlinked rRNA genes may reflect meaningful life history traits, as they tend to be associated with a mix of slow-growing free-living species and intracellular species. We speculate that unlinked rRNA genes may confer selective advantages in some environments, though the specific nature of these advantages remains undetermined and worthy of further investigation. More generally, the prevalence of unlinked rRNA genes in poorly-studied taxa serves as a reminder that paradigms derived from model organisms do not necessarily extend to the broader diversity of bacteria and archaea.
中文翻译:

未连接的 rRNA 基因广泛存在于细菌和古细菌中。
核糖体对细胞生命至关重要,其 RNA 成分的基因是细菌和古细菌中最保守和转录的基因。核糖体 RNA 基因通常被组织成单个操纵子,这种排列被认为有助于基因调控。实际上,一些细菌和古生菌并不共享这种典型的 rRNA 排列——它们的 16S 和 23S rRNA 基因在基因组中是分开的,被称为“未链接”。这种重排以前被视为细胞内细菌基因组降解的异常或副产品。在这里,我们利用完整的基因组和长读长的宏基因组数据来证明未链接的 16S 和 23S rRNA 基因比以前认为的更常见。未连接的 rRNA 基因存在于许多门中,最显着的是在 Deinococcus-Thermus、Chloroflexi 和 Planctomycetes 中,并且在自然环境中以不同的频率发生。我们发现土壤中高达 41% 的 rRNA 基因是未连接的,而人类肠道中所有测序的 rRNA 基因都是连接的。未连接的 rRNA 基因的频率可能反映了有意义的生活史特征,因为它们往往与生长缓慢的自由生活物种和细胞内物种的混合有关。我们推测未连接的 rRNA 基因可能在某些环境中赋予选择性优势,尽管这些优势的具体性质仍未确定,值得进一步研究。更一般地说,未链接 rRNA 基因在研究不足的分类群中的流行提醒人们,源自模式生物的范式不一定延伸到更广泛的细菌和古生菌多样性。我们发现土壤中高达 41% 的 rRNA 基因是未连接的,而人类肠道中所有测序的 rRNA 基因都是连接的。未连接的 rRNA 基因的频率可能反映了有意义的生活史特征,因为它们往往与生长缓慢的自由生活物种和细胞内物种的混合有关。我们推测未连接的 rRNA 基因可能在某些环境中赋予选择性优势,尽管这些优势的具体性质仍未确定,值得进一步研究。更一般地说,未链接 rRNA 基因在研究不足的分类群中的流行提醒人们,源自模式生物的范式不一定延伸到更广泛的细菌和古生菌多样性。我们发现土壤中高达 41% 的 rRNA 基因是未连接的,而人类肠道中所有测序的 rRNA 基因都是连接的。未连接的 rRNA 基因的频率可能反映了有意义的生活史特征,因为它们往往与生长缓慢的自由生活物种和细胞内物种的混合有关。我们推测未连接的 rRNA 基因可能在某些环境中赋予选择性优势,尽管这些优势的具体性质仍未确定,值得进一步研究。更一般地说,未链接 rRNA 基因在研究不足的分类群中的流行提醒人们,源自模式生物的范式不一定延伸到更广泛的细菌和古生菌多样性。其中所有测序的 rRNA 基因都连接在一起。未连接的 rRNA 基因的频率可能反映了有意义的生活史特征,因为它们往往与生长缓慢的自由生活物种和细胞内物种的混合有关。我们推测未连接的 rRNA 基因可能在某些环境中赋予选择性优势,尽管这些优势的具体性质仍未确定,值得进一步研究。更一般地说,未链接 rRNA 基因在研究不足的分类群中的流行提醒人们,源自模式生物的范式不一定延伸到更广泛的细菌和古生菌多样性。其中所有测序的 rRNA 基因都连接在一起。未连接的 rRNA 基因的频率可能反映了有意义的生活史特征,因为它们往往与生长缓慢的自由生活物种和细胞内物种的混合有关。我们推测未连接的 rRNA 基因可能在某些环境中赋予选择性优势,尽管这些优势的具体性质仍未确定,值得进一步研究。更一般地说,未链接 rRNA 基因在研究不足的分类群中的流行提醒人们,源自模式生物的范式不一定延伸到更广泛的细菌和古生菌多样性。我们推测未连接的 rRNA 基因可能在某些环境中赋予选择性优势,尽管这些优势的具体性质仍未确定,值得进一步研究。更一般地说,未链接 rRNA 基因在研究不足的分类群中的流行提醒人们,源自模式生物的范式不一定延伸到更广泛的细菌和古生菌多样性。我们推测未连接的 rRNA 基因可能在某些环境中赋予选择性优势,尽管这些优势的具体性质仍未确定,值得进一步研究。更一般地说,未链接 rRNA 基因在研究不足的分类群中的流行提醒人们,源自模式生物的范式不一定延伸到更广泛的细菌和古生菌多样性。
更新日期:2020-01-17
中文翻译:

未连接的 rRNA 基因广泛存在于细菌和古细菌中。
核糖体对细胞生命至关重要,其 RNA 成分的基因是细菌和古细菌中最保守和转录的基因。核糖体 RNA 基因通常被组织成单个操纵子,这种排列被认为有助于基因调控。实际上,一些细菌和古生菌并不共享这种典型的 rRNA 排列——它们的 16S 和 23S rRNA 基因在基因组中是分开的,被称为“未链接”。这种重排以前被视为细胞内细菌基因组降解的异常或副产品。在这里,我们利用完整的基因组和长读长的宏基因组数据来证明未链接的 16S 和 23S rRNA 基因比以前认为的更常见。未连接的 rRNA 基因存在于许多门中,最显着的是在 Deinococcus-Thermus、Chloroflexi 和 Planctomycetes 中,并且在自然环境中以不同的频率发生。我们发现土壤中高达 41% 的 rRNA 基因是未连接的,而人类肠道中所有测序的 rRNA 基因都是连接的。未连接的 rRNA 基因的频率可能反映了有意义的生活史特征,因为它们往往与生长缓慢的自由生活物种和细胞内物种的混合有关。我们推测未连接的 rRNA 基因可能在某些环境中赋予选择性优势,尽管这些优势的具体性质仍未确定,值得进一步研究。更一般地说,未链接 rRNA 基因在研究不足的分类群中的流行提醒人们,源自模式生物的范式不一定延伸到更广泛的细菌和古生菌多样性。我们发现土壤中高达 41% 的 rRNA 基因是未连接的,而人类肠道中所有测序的 rRNA 基因都是连接的。未连接的 rRNA 基因的频率可能反映了有意义的生活史特征,因为它们往往与生长缓慢的自由生活物种和细胞内物种的混合有关。我们推测未连接的 rRNA 基因可能在某些环境中赋予选择性优势,尽管这些优势的具体性质仍未确定,值得进一步研究。更一般地说,未链接 rRNA 基因在研究不足的分类群中的流行提醒人们,源自模式生物的范式不一定延伸到更广泛的细菌和古生菌多样性。我们发现土壤中高达 41% 的 rRNA 基因是未连接的,而人类肠道中所有测序的 rRNA 基因都是连接的。未连接的 rRNA 基因的频率可能反映了有意义的生活史特征,因为它们往往与生长缓慢的自由生活物种和细胞内物种的混合有关。我们推测未连接的 rRNA 基因可能在某些环境中赋予选择性优势,尽管这些优势的具体性质仍未确定,值得进一步研究。更一般地说,未链接 rRNA 基因在研究不足的分类群中的流行提醒人们,源自模式生物的范式不一定延伸到更广泛的细菌和古生菌多样性。其中所有测序的 rRNA 基因都连接在一起。未连接的 rRNA 基因的频率可能反映了有意义的生活史特征,因为它们往往与生长缓慢的自由生活物种和细胞内物种的混合有关。我们推测未连接的 rRNA 基因可能在某些环境中赋予选择性优势,尽管这些优势的具体性质仍未确定,值得进一步研究。更一般地说,未链接 rRNA 基因在研究不足的分类群中的流行提醒人们,源自模式生物的范式不一定延伸到更广泛的细菌和古生菌多样性。其中所有测序的 rRNA 基因都连接在一起。未连接的 rRNA 基因的频率可能反映了有意义的生活史特征,因为它们往往与生长缓慢的自由生活物种和细胞内物种的混合有关。我们推测未连接的 rRNA 基因可能在某些环境中赋予选择性优势,尽管这些优势的具体性质仍未确定,值得进一步研究。更一般地说,未链接 rRNA 基因在研究不足的分类群中的流行提醒人们,源自模式生物的范式不一定延伸到更广泛的细菌和古生菌多样性。我们推测未连接的 rRNA 基因可能在某些环境中赋予选择性优势,尽管这些优势的具体性质仍未确定,值得进一步研究。更一般地说,未链接 rRNA 基因在研究不足的分类群中的流行提醒人们,源自模式生物的范式不一定延伸到更广泛的细菌和古生菌多样性。我们推测未连接的 rRNA 基因可能在某些环境中赋予选择性优势,尽管这些优势的具体性质仍未确定,值得进一步研究。更一般地说,未链接 rRNA 基因在研究不足的分类群中的流行提醒人们,源自模式生物的范式不一定延伸到更广泛的细菌和古生菌多样性。











































 京公网安备 11010802027423号
京公网安备 11010802027423号