当前位置:
X-MOL 学术
›
J. Chem. Inf. Model.
›
论文详情
Our official English website, www.x-mol.net, welcomes your
feedback! (Note: you will need to create a separate account there.)
Leveraging Protein Dynamics to Identify Functional Phosphorylation Sites using Deep Learning Models
Journal of Chemical Information and Modeling ( IF 5.6 ) Pub Date : 2022-07-11 , DOI: 10.1021/acs.jcim.2c00484 Fei Zhu 1, 2 , Sijie Yang 2 , Fanwang Meng 3 , Yuxiang Zheng 1 , Xin Ku 4 , Cheng Luo 5 , Guang Hu 1 , Zhongjie Liang 1, 4, 5
Journal of Chemical Information and Modeling ( IF 5.6 ) Pub Date : 2022-07-11 , DOI: 10.1021/acs.jcim.2c00484 Fei Zhu 1, 2 , Sijie Yang 2 , Fanwang Meng 3 , Yuxiang Zheng 1 , Xin Ku 4 , Cheng Luo 5 , Guang Hu 1 , Zhongjie Liang 1, 4, 5
Affiliation
|
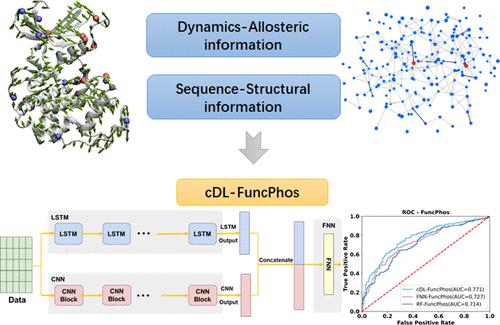
Accurate prediction of post-translational modifications (PTMs) is of great significance in understanding cellular processes, by modulating protein structure and dynamics. Nowadays, with the rapid growth of protein data at different “omics” levels, machine learning models largely enriched the prediction of PTMs. However, most machine learning models only rely on protein sequence and little structural information. The lack of the systematic dynamics analysis underlying PTMs largely limits the PTM functional predictions. In this research, we present two dynamics-centric deep learning models, namely, cDL-PAU and cDL-FuncPhos, by incorporating sequence, structure, and dynamics-based features to elucidate the molecular basis and underlying functional landscape of PTMs. cDL-PAU achieved satisfactory area under the curve (AUC) scores of 0.804–0.888 for predicting phosphorylation, acetylation, and ubiquitination (PAU) sites, while cDL-FuncPhos achieved an AUC value of 0.771 for predicting functional phosphorylation (FuncPhos) sites, displaying reliable improvements. Through a feature selection, the dynamics-based coupling and commute ability show large contributions in discovering PAU sites and FuncPhos sites, suggesting the allosteric propensity for important PTMs. The application of cDL-FuncPhos in three oncoproteins not only corroborates its strong performance in FuncPhos prioritization but also gains insight into the physical basis for the functions. The source code and data set of cDL-PAU and cDL-FuncPhos are available at https://github.com/ComputeSuda/PTM_ML.
中文翻译:

利用蛋白质动力学使用深度学习模型识别功能性磷酸化位点
通过调节蛋白质结构和动力学,准确预测翻译后修饰 (PTM) 对于理解细胞过程具有重要意义。如今,随着不同“组学”水平的蛋白质数据的快速增长,机器学习模型在很大程度上丰富了 PTM 的预测。然而,大多数机器学习模型只依赖于蛋白质序列和很少的结构信息。PTM 缺乏系统动力学分析在很大程度上限制了 PTM 的功能预测。在这项研究中,我们提出了两个以动力学为中心的深度学习模型,即 cDL-PAU 和 cDL-FuncPhos,通过结合基于序列、结构和动力学的特征来阐明 PTM 的分子基础和潜在的功能景观。cDL-PAU 的曲线下面积 (AUC) 得分为 0.804-0,令人满意。888 用于预测磷酸化、乙酰化和泛素化 (PAU) 位点,而 cDL-FuncPhos 在预测功能性磷酸化 (FuncPhos) 位点时的 AUC 值为 0.771,显示出可靠的改进。通过特征选择,基于动力学的耦合和通勤能力在发现 PAU 位点和 FuncPhos 位点方面表现出很大的贡献,表明重要 PTM 的变构倾向。cDL-FuncPhos 在三种癌蛋白中的应用不仅证实了其在 FuncPhos 优先排序方面的强大性能,而且还深入了解了这些功能的物理基础。cDL-PAU 和 cDL-FuncPhos 的源代码和数据集可在 https://github.com/ComputeSuda/PTM_ML 获得。771 用于预测功能性磷酸化 (FuncPhos) 位点,显示出可靠的改进。通过特征选择,基于动力学的耦合和通勤能力在发现 PAU 位点和 FuncPhos 位点方面表现出很大的贡献,表明重要 PTM 的变构倾向。cDL-FuncPhos 在三种癌蛋白中的应用不仅证实了其在 FuncPhos 优先排序方面的强大性能,而且还深入了解了这些功能的物理基础。cDL-PAU 和 cDL-FuncPhos 的源代码和数据集可在 https://github.com/ComputeSuda/PTM_ML 获得。771 用于预测功能性磷酸化 (FuncPhos) 位点,显示出可靠的改进。通过特征选择,基于动力学的耦合和通勤能力在发现 PAU 位点和 FuncPhos 位点方面表现出很大的贡献,表明重要 PTM 的变构倾向。cDL-FuncPhos 在三种癌蛋白中的应用不仅证实了其在 FuncPhos 优先排序方面的强大性能,而且还深入了解了这些功能的物理基础。cDL-PAU 和 cDL-FuncPhos 的源代码和数据集可在 https://github.com/ComputeSuda/PTM_ML 获得。表明重要 PTM 的变构倾向。cDL-FuncPhos 在三种癌蛋白中的应用不仅证实了其在 FuncPhos 优先排序方面的强大性能,而且还深入了解了这些功能的物理基础。cDL-PAU 和 cDL-FuncPhos 的源代码和数据集可在 https://github.com/ComputeSuda/PTM_ML 获得。表明重要 PTM 的变构倾向。cDL-FuncPhos 在三种癌蛋白中的应用不仅证实了其在 FuncPhos 优先排序方面的强大性能,而且还深入了解了这些功能的物理基础。cDL-PAU 和 cDL-FuncPhos 的源代码和数据集可在 https://github.com/ComputeSuda/PTM_ML 获得。
更新日期:2022-07-11
中文翻译:

利用蛋白质动力学使用深度学习模型识别功能性磷酸化位点
通过调节蛋白质结构和动力学,准确预测翻译后修饰 (PTM) 对于理解细胞过程具有重要意义。如今,随着不同“组学”水平的蛋白质数据的快速增长,机器学习模型在很大程度上丰富了 PTM 的预测。然而,大多数机器学习模型只依赖于蛋白质序列和很少的结构信息。PTM 缺乏系统动力学分析在很大程度上限制了 PTM 的功能预测。在这项研究中,我们提出了两个以动力学为中心的深度学习模型,即 cDL-PAU 和 cDL-FuncPhos,通过结合基于序列、结构和动力学的特征来阐明 PTM 的分子基础和潜在的功能景观。cDL-PAU 的曲线下面积 (AUC) 得分为 0.804-0,令人满意。888 用于预测磷酸化、乙酰化和泛素化 (PAU) 位点,而 cDL-FuncPhos 在预测功能性磷酸化 (FuncPhos) 位点时的 AUC 值为 0.771,显示出可靠的改进。通过特征选择,基于动力学的耦合和通勤能力在发现 PAU 位点和 FuncPhos 位点方面表现出很大的贡献,表明重要 PTM 的变构倾向。cDL-FuncPhos 在三种癌蛋白中的应用不仅证实了其在 FuncPhos 优先排序方面的强大性能,而且还深入了解了这些功能的物理基础。cDL-PAU 和 cDL-FuncPhos 的源代码和数据集可在 https://github.com/ComputeSuda/PTM_ML 获得。771 用于预测功能性磷酸化 (FuncPhos) 位点,显示出可靠的改进。通过特征选择,基于动力学的耦合和通勤能力在发现 PAU 位点和 FuncPhos 位点方面表现出很大的贡献,表明重要 PTM 的变构倾向。cDL-FuncPhos 在三种癌蛋白中的应用不仅证实了其在 FuncPhos 优先排序方面的强大性能,而且还深入了解了这些功能的物理基础。cDL-PAU 和 cDL-FuncPhos 的源代码和数据集可在 https://github.com/ComputeSuda/PTM_ML 获得。771 用于预测功能性磷酸化 (FuncPhos) 位点,显示出可靠的改进。通过特征选择,基于动力学的耦合和通勤能力在发现 PAU 位点和 FuncPhos 位点方面表现出很大的贡献,表明重要 PTM 的变构倾向。cDL-FuncPhos 在三种癌蛋白中的应用不仅证实了其在 FuncPhos 优先排序方面的强大性能,而且还深入了解了这些功能的物理基础。cDL-PAU 和 cDL-FuncPhos 的源代码和数据集可在 https://github.com/ComputeSuda/PTM_ML 获得。表明重要 PTM 的变构倾向。cDL-FuncPhos 在三种癌蛋白中的应用不仅证实了其在 FuncPhos 优先排序方面的强大性能,而且还深入了解了这些功能的物理基础。cDL-PAU 和 cDL-FuncPhos 的源代码和数据集可在 https://github.com/ComputeSuda/PTM_ML 获得。表明重要 PTM 的变构倾向。cDL-FuncPhos 在三种癌蛋白中的应用不仅证实了其在 FuncPhos 优先排序方面的强大性能,而且还深入了解了这些功能的物理基础。cDL-PAU 和 cDL-FuncPhos 的源代码和数据集可在 https://github.com/ComputeSuda/PTM_ML 获得。











































 京公网安备 11010802027423号
京公网安备 11010802027423号