当前位置:
X-MOL 学术
›
J. Chem. Inf. Model.
›
论文详情
Our official English website, www.x-mol.net, welcomes your
feedback! (Note: you will need to create a separate account there.)
DNA Deformation Exerted by Regulatory DNA-Binding Motifs in Human Alkyladenine DNA Glycosylase Promotes Base Flipping
Journal of Chemical Information and Modeling ( IF 5.6 ) Pub Date : 2022-06-16 , DOI: 10.1021/acs.jcim.2c00091 Lingyan Wang 1 , Kun Xi 2 , Lizhe Zhu 2 , Lin-Tai Da 1
Journal of Chemical Information and Modeling ( IF 5.6 ) Pub Date : 2022-06-16 , DOI: 10.1021/acs.jcim.2c00091 Lingyan Wang 1 , Kun Xi 2 , Lizhe Zhu 2 , Lin-Tai Da 1
Affiliation
|
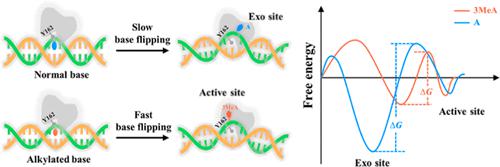
Human alkyladenine DNA glycosylase (AAG) is a key enzyme that corrects a broad range of alkylated and deaminated nucleobases to maintain genomic integrity. When encountering the lesions, AAG adopts a base-flipping strategy to extrude the target base from the DNA duplex to its active site, thereby cleaving the glycosidic bond. Despite its functional importance, the detailed mechanism of such base extrusion and how AAG distinguishes the lesions from an excess of normal bases both remain elusive. Here, through the Markov state model constructed on extensive all-atom molecular dynamics simulations, we find that the alkylated nucleobase (N3-methyladenine, 3MeA) everts through the DNA major groove. Two key AAG motifs, the intercalation and E131-N146 motifs, play active roles in bending/pressing the DNA backbone and widening the DNA minor groove during 3MeA eversion. In particular, the intercalated residue Y162 is involved in buckling the target site at the early stage of 3MeA eversion. Our traveling-salesman based automated path searching algorithm further revealed that a non-target normal adenine tends to be trapped in an exo site near the active site, which however barely exists for a target base 3MeA. Collectively, these results suggest that the Markov state model combined with traveling-salesman based automated path searching acts as a promising approach for studying complex conformational changes of biomolecules and dissecting the elaborate mechanism of target recognition by this unique enzyme.
中文翻译:

人烷基腺嘌呤 DNA 糖基化酶中调节性 DNA 结合基序引起的 DNA 变形促进碱基翻转
人类烷基腺嘌呤 DNA 糖基化酶 (AAG) 是一种关键酶,可纠正广泛的烷基化和脱氨基核碱基以维持基因组完整性。当遇到病灶时,AAG 采用碱基翻转策略,将目标碱基从 DNA 双链体挤出到其活性位点,从而裂解糖苷键。尽管具有功能重要性,但这种碱基挤出的详细机制以及 AAG 如何区分病变与过多的正常碱基仍然难以捉摸。在这里,通过基于广泛的全原子分子动力学模拟构建的马尔可夫状态模型,我们发现烷基化核碱基(N3-甲基腺嘌呤,3MeA)通过 DNA 大沟外翻。两个关键的 AAG 基序,嵌入和 E131-N146 基序,在 3MeA 外翻过程中,在弯曲/挤压 DNA 骨架和扩大 DNA 小沟方面发挥积极作用。特别是,插入的残基 Y162 在 3MeA 外翻的早期参与了目标位点的屈曲。我们基于旅行商的自动路径搜索算法进一步揭示了非目标正常腺嘌呤倾向于被困在靠近活性位点的外位点中,但对于目标碱基 3MeA 来说几乎不存在。总的来说,这些结果表明,马尔可夫状态模型与基于旅行商的自动路径搜索相结合,是研究生物分子复杂构象变化和剖析这种独特酶识别目标的复杂机制的一种很有前途的方法。插入的残基 Y162 在 3MeA 外翻的早期参与了目标部位的屈曲。我们基于旅行商的自动路径搜索算法进一步揭示了非目标正常腺嘌呤倾向于被困在靠近活性位点的外位点中,但对于目标碱基 3MeA 来说几乎不存在。总的来说,这些结果表明,马尔可夫状态模型与基于旅行商的自动路径搜索相结合,是研究生物分子复杂构象变化和剖析这种独特酶识别目标的复杂机制的一种很有前途的方法。插入的残基 Y162 在 3MeA 外翻的早期参与了目标部位的屈曲。我们基于旅行商的自动路径搜索算法进一步揭示了非目标正常腺嘌呤倾向于被困在靠近活性位点的外位点中,但对于目标碱基 3MeA 来说几乎不存在。总的来说,这些结果表明,马尔可夫状态模型与基于旅行商的自动路径搜索相结合,是研究生物分子复杂构象变化和剖析这种独特酶识别目标的复杂机制的一种很有前途的方法。然而,对于目标碱基 3MeA,它几乎不存在。总的来说,这些结果表明,马尔可夫状态模型与基于旅行商的自动路径搜索相结合,是研究生物分子复杂构象变化和剖析这种独特酶识别目标的复杂机制的一种很有前途的方法。然而,对于目标碱基 3MeA,它几乎不存在。总的来说,这些结果表明,马尔可夫状态模型与基于旅行商的自动路径搜索相结合,是研究生物分子复杂构象变化和剖析这种独特酶识别目标的复杂机制的一种很有前途的方法。
更新日期:2022-06-16
中文翻译:

人烷基腺嘌呤 DNA 糖基化酶中调节性 DNA 结合基序引起的 DNA 变形促进碱基翻转
人类烷基腺嘌呤 DNA 糖基化酶 (AAG) 是一种关键酶,可纠正广泛的烷基化和脱氨基核碱基以维持基因组完整性。当遇到病灶时,AAG 采用碱基翻转策略,将目标碱基从 DNA 双链体挤出到其活性位点,从而裂解糖苷键。尽管具有功能重要性,但这种碱基挤出的详细机制以及 AAG 如何区分病变与过多的正常碱基仍然难以捉摸。在这里,通过基于广泛的全原子分子动力学模拟构建的马尔可夫状态模型,我们发现烷基化核碱基(N3-甲基腺嘌呤,3MeA)通过 DNA 大沟外翻。两个关键的 AAG 基序,嵌入和 E131-N146 基序,在 3MeA 外翻过程中,在弯曲/挤压 DNA 骨架和扩大 DNA 小沟方面发挥积极作用。特别是,插入的残基 Y162 在 3MeA 外翻的早期参与了目标位点的屈曲。我们基于旅行商的自动路径搜索算法进一步揭示了非目标正常腺嘌呤倾向于被困在靠近活性位点的外位点中,但对于目标碱基 3MeA 来说几乎不存在。总的来说,这些结果表明,马尔可夫状态模型与基于旅行商的自动路径搜索相结合,是研究生物分子复杂构象变化和剖析这种独特酶识别目标的复杂机制的一种很有前途的方法。插入的残基 Y162 在 3MeA 外翻的早期参与了目标部位的屈曲。我们基于旅行商的自动路径搜索算法进一步揭示了非目标正常腺嘌呤倾向于被困在靠近活性位点的外位点中,但对于目标碱基 3MeA 来说几乎不存在。总的来说,这些结果表明,马尔可夫状态模型与基于旅行商的自动路径搜索相结合,是研究生物分子复杂构象变化和剖析这种独特酶识别目标的复杂机制的一种很有前途的方法。插入的残基 Y162 在 3MeA 外翻的早期参与了目标部位的屈曲。我们基于旅行商的自动路径搜索算法进一步揭示了非目标正常腺嘌呤倾向于被困在靠近活性位点的外位点中,但对于目标碱基 3MeA 来说几乎不存在。总的来说,这些结果表明,马尔可夫状态模型与基于旅行商的自动路径搜索相结合,是研究生物分子复杂构象变化和剖析这种独特酶识别目标的复杂机制的一种很有前途的方法。然而,对于目标碱基 3MeA,它几乎不存在。总的来说,这些结果表明,马尔可夫状态模型与基于旅行商的自动路径搜索相结合,是研究生物分子复杂构象变化和剖析这种独特酶识别目标的复杂机制的一种很有前途的方法。然而,对于目标碱基 3MeA,它几乎不存在。总的来说,这些结果表明,马尔可夫状态模型与基于旅行商的自动路径搜索相结合,是研究生物分子复杂构象变化和剖析这种独特酶识别目标的复杂机制的一种很有前途的方法。











































 京公网安备 11010802027423号
京公网安备 11010802027423号