当前位置:
X-MOL 学术
›
J. Chem. Inf. Model.
›
论文详情
Our official English website, www.x-mol.net, welcomes your
feedback! (Note: you will need to create a separate account there.)
IDL-PPBopt: A Strategy for Prediction and Optimization of Human Plasma Protein Binding of Compounds via an Interpretable Deep Learning Method
Journal of Chemical Information and Modeling ( IF 5.6 ) Pub Date : 2022-05-24 , DOI: 10.1021/acs.jcim.2c00297 Chaofeng Lou 1 , Hongbin Yang 1 , Jiye Wang 1 , Mengting Huang 1 , Weihua Li 1 , Guixia Liu 1 , Philip W Lee 1 , Yun Tang 1
Journal of Chemical Information and Modeling ( IF 5.6 ) Pub Date : 2022-05-24 , DOI: 10.1021/acs.jcim.2c00297 Chaofeng Lou 1 , Hongbin Yang 1 , Jiye Wang 1 , Mengting Huang 1 , Weihua Li 1 , Guixia Liu 1 , Philip W Lee 1 , Yun Tang 1
Affiliation
|
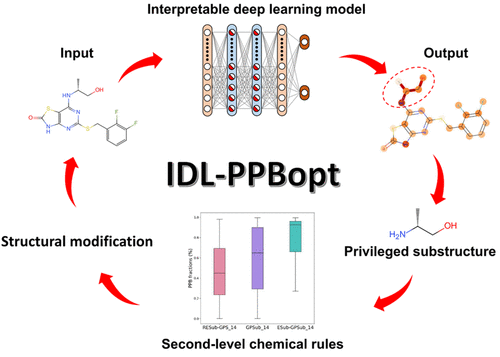
The prediction and optimization of pharmacokinetic properties are essential in lead optimization. Traditional strategies mainly depend on the empirical chemical rules from medicinal chemists. However, with the rising amount of data, it is getting more difficult to manually extract useful medicinal chemistry knowledge. To this end, we introduced IDL-PPBopt, a computational strategy for predicting and optimizing the plasma protein binding (PPB) property based on an interpretable deep learning method. At first, a curated PPB data set was used to construct an interpretable deep learning model, which showed excellent predictive performance with a root mean squared error of 0.112 for the entire test set. Then, we designed a detection protocol based on the model and Wilcoxon test to identify the PPB-related substructures (named privileged substructures, PSubs) for each molecule. In total, 22 general privileged substructures (GPSubs) were identified, which shared some common features such as nitrogen-containing groups, diamines with two carbon units, and azetidine. Furthermore, a series of second-level chemical rules for each GPSub were derived through a statistical test and then summarized into substructure pairs. We demonstrated that these substructure pairs were equally applicable outside the training set and accordingly customized the structural modification schemes for each GPSub, which provided alternatives for the optimization of the PPB property. Therefore, IDL-PPBopt provides a promising scheme for the prediction and optimization of the PPB property and would be helpful for lead optimization of other pharmacokinetic properties.
中文翻译:

IDL-PPBopt:通过可解释的深度学习方法预测和优化化合物与人血浆蛋白结合的策略
药代动力学特性的预测和优化对于先导优化至关重要。传统策略主要依赖于药物化学家的经验化学规则。然而,随着数据量的增加,人工提取有用的药物化学知识变得越来越困难。为此,我们介绍了 IDL-PPBopt,这是一种基于可解释的深度学习方法预测和优化血浆蛋白结合 (PPB) 特性的计算策略。起初,使用精选的 PPB 数据集构建可解释的深度学习模型,该模型显示出出色的预测性能,整个测试集的均方根误差为 0.112。然后,我们设计了一个基于模型和 Wilcoxon 检验的检测协议来识别 PPB 相关的子结构(命名为特权子结构,PSubs) 用于每个分子。总共确定了 22 个通用特权子结构 (GPSub),它们具有一些共同特征,例如含氮基团、具有两个碳单元的二胺和氮杂环丁烷。此外,通过统计检验得出每个 GPSub 的一系列二级化学规则,然后汇总成子结构对。我们证明了这些子结构对在训练集之外同样适用,并相应地为每个 GPSub 定制了结构修改方案,这为 PPB 属性的优化提供了替代方案。因此,IDL-PPBopt 为 PPB 特性的预测和优化提供了一个有前景的方案,并将有助于其他药代动力学特性的先导优化。确定了 22 个通用特权子结构 (GPSub),它们具有一些共同特征,例如含氮基团、具有两个碳单元的二胺和氮杂环丁烷。此外,通过统计检验得出每个 GPSub 的一系列二级化学规则,然后汇总成子结构对。我们证明了这些子结构对在训练集之外同样适用,并相应地为每个 GPSub 定制了结构修改方案,这为 PPB 属性的优化提供了替代方案。因此,IDL-PPBopt 为 PPB 特性的预测和优化提供了一个有前景的方案,并将有助于其他药代动力学特性的先导优化。确定了 22 个通用特权子结构 (GPSub),它们具有一些共同特征,例如含氮基团、具有两个碳单元的二胺和氮杂环丁烷。此外,通过统计检验得出每个 GPSub 的一系列二级化学规则,然后汇总成子结构对。我们证明了这些子结构对在训练集之外同样适用,并相应地为每个 GPSub 定制了结构修改方案,这为 PPB 属性的优化提供了替代方案。因此,IDL-PPBopt 为 PPB 特性的预测和优化提供了一个有前景的方案,并将有助于其他药代动力学特性的先导优化。它们具有一些共同的特征,例如含氮基团、具有两个碳单元的二胺和氮杂环丁烷。此外,通过统计检验得出每个 GPSub 的一系列二级化学规则,然后汇总成子结构对。我们证明了这些子结构对在训练集之外同样适用,并相应地为每个 GPSub 定制了结构修改方案,这为 PPB 属性的优化提供了替代方案。因此,IDL-PPBopt 为 PPB 特性的预测和优化提供了一个有前景的方案,并将有助于其他药代动力学特性的先导优化。它们具有一些共同的特征,例如含氮基团、具有两个碳单元的二胺和氮杂环丁烷。此外,通过统计检验得出每个 GPSub 的一系列二级化学规则,然后汇总成子结构对。我们证明了这些子结构对在训练集之外同样适用,并相应地为每个 GPSub 定制了结构修改方案,这为 PPB 属性的优化提供了替代方案。因此,IDL-PPBopt 为 PPB 特性的预测和优化提供了一个有前景的方案,并将有助于其他药代动力学特性的先导优化。通过统计检验得出每个 GPSub 的一系列二级化学规则,然后汇总成子结构对。我们证明了这些子结构对在训练集之外同样适用,并相应地为每个 GPSub 定制了结构修改方案,这为 PPB 属性的优化提供了替代方案。因此,IDL-PPBopt 为 PPB 特性的预测和优化提供了一个有前景的方案,并将有助于其他药代动力学特性的先导优化。通过统计检验得出每个 GPSub 的一系列二级化学规则,然后汇总成子结构对。我们证明了这些子结构对在训练集之外同样适用,并相应地为每个 GPSub 定制了结构修改方案,这为 PPB 属性的优化提供了替代方案。因此,IDL-PPBopt 为 PPB 特性的预测和优化提供了一个有前景的方案,并将有助于其他药代动力学特性的先导优化。
更新日期:2022-05-24
中文翻译:

IDL-PPBopt:通过可解释的深度学习方法预测和优化化合物与人血浆蛋白结合的策略
药代动力学特性的预测和优化对于先导优化至关重要。传统策略主要依赖于药物化学家的经验化学规则。然而,随着数据量的增加,人工提取有用的药物化学知识变得越来越困难。为此,我们介绍了 IDL-PPBopt,这是一种基于可解释的深度学习方法预测和优化血浆蛋白结合 (PPB) 特性的计算策略。起初,使用精选的 PPB 数据集构建可解释的深度学习模型,该模型显示出出色的预测性能,整个测试集的均方根误差为 0.112。然后,我们设计了一个基于模型和 Wilcoxon 检验的检测协议来识别 PPB 相关的子结构(命名为特权子结构,PSubs) 用于每个分子。总共确定了 22 个通用特权子结构 (GPSub),它们具有一些共同特征,例如含氮基团、具有两个碳单元的二胺和氮杂环丁烷。此外,通过统计检验得出每个 GPSub 的一系列二级化学规则,然后汇总成子结构对。我们证明了这些子结构对在训练集之外同样适用,并相应地为每个 GPSub 定制了结构修改方案,这为 PPB 属性的优化提供了替代方案。因此,IDL-PPBopt 为 PPB 特性的预测和优化提供了一个有前景的方案,并将有助于其他药代动力学特性的先导优化。确定了 22 个通用特权子结构 (GPSub),它们具有一些共同特征,例如含氮基团、具有两个碳单元的二胺和氮杂环丁烷。此外,通过统计检验得出每个 GPSub 的一系列二级化学规则,然后汇总成子结构对。我们证明了这些子结构对在训练集之外同样适用,并相应地为每个 GPSub 定制了结构修改方案,这为 PPB 属性的优化提供了替代方案。因此,IDL-PPBopt 为 PPB 特性的预测和优化提供了一个有前景的方案,并将有助于其他药代动力学特性的先导优化。确定了 22 个通用特权子结构 (GPSub),它们具有一些共同特征,例如含氮基团、具有两个碳单元的二胺和氮杂环丁烷。此外,通过统计检验得出每个 GPSub 的一系列二级化学规则,然后汇总成子结构对。我们证明了这些子结构对在训练集之外同样适用,并相应地为每个 GPSub 定制了结构修改方案,这为 PPB 属性的优化提供了替代方案。因此,IDL-PPBopt 为 PPB 特性的预测和优化提供了一个有前景的方案,并将有助于其他药代动力学特性的先导优化。它们具有一些共同的特征,例如含氮基团、具有两个碳单元的二胺和氮杂环丁烷。此外,通过统计检验得出每个 GPSub 的一系列二级化学规则,然后汇总成子结构对。我们证明了这些子结构对在训练集之外同样适用,并相应地为每个 GPSub 定制了结构修改方案,这为 PPB 属性的优化提供了替代方案。因此,IDL-PPBopt 为 PPB 特性的预测和优化提供了一个有前景的方案,并将有助于其他药代动力学特性的先导优化。它们具有一些共同的特征,例如含氮基团、具有两个碳单元的二胺和氮杂环丁烷。此外,通过统计检验得出每个 GPSub 的一系列二级化学规则,然后汇总成子结构对。我们证明了这些子结构对在训练集之外同样适用,并相应地为每个 GPSub 定制了结构修改方案,这为 PPB 属性的优化提供了替代方案。因此,IDL-PPBopt 为 PPB 特性的预测和优化提供了一个有前景的方案,并将有助于其他药代动力学特性的先导优化。通过统计检验得出每个 GPSub 的一系列二级化学规则,然后汇总成子结构对。我们证明了这些子结构对在训练集之外同样适用,并相应地为每个 GPSub 定制了结构修改方案,这为 PPB 属性的优化提供了替代方案。因此,IDL-PPBopt 为 PPB 特性的预测和优化提供了一个有前景的方案,并将有助于其他药代动力学特性的先导优化。通过统计检验得出每个 GPSub 的一系列二级化学规则,然后汇总成子结构对。我们证明了这些子结构对在训练集之外同样适用,并相应地为每个 GPSub 定制了结构修改方案,这为 PPB 属性的优化提供了替代方案。因此,IDL-PPBopt 为 PPB 特性的预测和优化提供了一个有前景的方案,并将有助于其他药代动力学特性的先导优化。











































 京公网安备 11010802027423号
京公网安备 11010802027423号