Journal of Molecular Graphics and Modelling ( IF 2.7 ) Pub Date : 2021-09-15 , DOI: 10.1016/j.jmgm.2021.108023 Rodrigo Ochoa 1 , Karen Palacio-Rodriguez 1 , Camila M Clemente 2 , Natalia S Adler 3
|
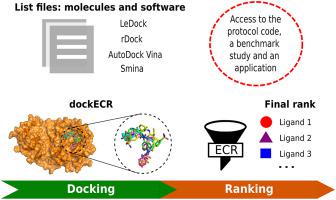
The development of open computational pipelines to accelerate the discovery of treatments for emerging diseases allows finding novel solutions in shorter periods of time. Consensus molecular docking is one of these approaches, and its main purpose is to increase the detection of real actives within virtual screening campaigns. Here we present dockECR, an open consensus docking and ranking protocol that implements the exponential consensus ranking method to prioritize molecular candidates. The protocol uses four open source molecular docking programs: AutoDock Vina, Smina, LeDock and rDock, to rank the molecules. In addition, we introduce a scoring strategy based on the average RMSD obtained from comparing the best poses from each single program to complement the consensus ranking with information about the predicted poses. The protocol was benchmarked using 15 relevant protein targets with known actives and decoys, and applied using the main protease of the SARS-CoV-2 virus. For the application, different crystal structures of the protease, and frames obtained from molecular dynamics simulations were used to dock a library of 79 molecules derived from previously co-crystallized fragments. The ranking obtained with dockECR was used to prioritize eight candidates, which were evaluated in terms of the interactions generated with key residues from the protease. The protocol can be implemented in any virtual screening campaign involving proteins as molecular targets. The dockECR code is publicly available at: https://github.com/rochoa85/dockECR.
中文翻译:

dockECR:用于小分子虚拟筛选的开放共识对接和排序协议
开发开放式计算管道以加速发现新兴疾病的治疗方法,可以在更短的时间内找到新的解决方案。共识分子对接是其中一种方法,其主要目的是在虚拟筛选活动中增加对真实活性物质的检测。在这里,我们介绍了 dockECR,这是一种开放的共识对接和排名协议,它实施指数共识排名方法来确定候选分子的优先级。该协议使用四个开源分子对接程序:AutoDock Vina、Smina、LeDock 和 rDock,对分子进行排序。此外,我们引入了一种基于平均 RMSD 的评分策略,该平均 RMSD 通过比较每个单个程序的最佳姿势获得,以使用有关预测姿势的信息补充共识排名。该协议使用 15 个具有已知活性和诱饵的相关蛋白质靶标进行基准测试,并使用 SARS-CoV-2 病毒的主要蛋白酶进行应用。对于该应用,蛋白酶的不同晶体结构和从分子动力学模拟中获得的框架被用于对接一个由先前共结晶片段衍生的 79 个分子的库。使用 dockECR 获得的排名用于确定八个候选者的优先级,这些候选者根据与蛋白酶关键残基产生的相互作用进行评估。该协议可以在任何涉及蛋白质作为分子目标的虚拟筛选活动中实施。dockECR 代码公开于:https://github.com/rochoa85/dockECR。并使用 SARS-CoV-2 病毒的主要蛋白酶进行应用。对于该应用,蛋白酶的不同晶体结构和从分子动力学模拟中获得的框架被用于对接一个由先前共结晶片段衍生的 79 个分子的库。使用 dockECR 获得的排名用于确定八个候选者的优先级,这些候选者根据与蛋白酶关键残基产生的相互作用进行评估。该协议可以在任何涉及蛋白质作为分子目标的虚拟筛选活动中实施。dockECR 代码公开于:https://github.com/rochoa85/dockECR。并使用 SARS-CoV-2 病毒的主要蛋白酶进行应用。对于该应用,蛋白酶的不同晶体结构和从分子动力学模拟中获得的框架被用于对接一个由先前共结晶片段衍生的 79 个分子的库。使用 dockECR 获得的排名用于确定八个候选者的优先级,这些候选者根据与蛋白酶关键残基产生的相互作用进行评估。该协议可以在任何涉及蛋白质作为分子目标的虚拟筛选活动中实施。dockECR 代码公开于:https://github.com/rochoa85/dockECR。使用 dockECR 获得的排名用于确定八个候选者的优先级,这些候选者根据与蛋白酶关键残基产生的相互作用进行评估。该协议可以在任何涉及蛋白质作为分子目标的虚拟筛选活动中实施。dockECR 代码公开于:https://github.com/rochoa85/dockECR。使用 dockECR 获得的排名用于确定八个候选者的优先级,这些候选者根据与蛋白酶关键残基产生的相互作用进行评估。该协议可以在任何涉及蛋白质作为分子目标的虚拟筛选活动中实施。dockECR 代码公开于:https://github.com/rochoa85/dockECR。











































 京公网安备 11010802027423号
京公网安备 11010802027423号