当前位置:
X-MOL 学术
›
Biochemistry
›
论文详情
Our official English website, www.x-mol.net, welcomes your
feedback! (Note: you will need to create a separate account there.)
Helical and β-Turn Conformations in the Peptide Recognition Regions of the VIM1 PHD Finger Abrogate H3K4 Peptide Recognition
Biochemistry ( IF 2.9 ) Pub Date : 2021-08-18 , DOI: 10.1021/acs.biochem.1c00191 Suman Abhishek 1 , Waghela Deeksha 1 , Eerappa Rajakumara 1
Biochemistry ( IF 2.9 ) Pub Date : 2021-08-18 , DOI: 10.1021/acs.biochem.1c00191 Suman Abhishek 1 , Waghela Deeksha 1 , Eerappa Rajakumara 1
Affiliation
|
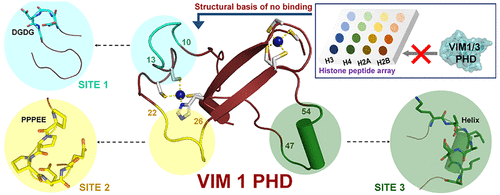
The PHD finger-containing VARIANT IN METHYLATION/ORTHRUS (VIM/ORTH) family of proteins in Arabidopsis consists of functional homologues of mammalian UHRF1 and is required for the maintenance of DNA methylation. Comparison of the sequence with those of other PHD fingers implied that VIM1 and VIM3 PHD could recognize lysine 4 of histone H3 (H3K4) through interactions mediated by a conserved aspartic acid. However, our calorimetric and modified histone peptide array binding studies suggested that neither H3K4 nor other histone marks are recognized by VIM1 and VIM3 PHD fingers. Here, we report a 2.6 Å resolution crystal structure of the VIM1 PHD finger and demonstrate significant structural changes in the putative H3 recognition segments in contrast to canonical H3K4 binding PHD fingers. These changes include (i) the H3A1 binding region, (ii) strand β1 that forms an intermolecular β-sheet with the H3 peptide, and (iii) an aspartate-containing motif involved in salt bridge interaction with H3K4, which together appear to abrogate recognition of H3K4 by the VIM1 PHD finger. To understand the significance of the altered structural features in the VIM1 PHD that might prevent histone H3 recognition, we modeled a chimeric VIM1 PHD (chmVIM1 PHD) by grafting the peptide binding structural features of the BHC80 PHD onto the VIM1 PHD. Molecular dynamics simulation and metadynamics analyses revealed that the chmVIM1 PHD-H3 complex is stable and also showed a network of intermolecular interactions similar to those of the BHC80 PHD-H3 complex. Collectively, this study reveals that subtle structural changes in the peptide binding region of the VIM1 PHD abrogate histone H3 recognition.
中文翻译:

VIM1 PHD Finger Abrogate H3K4 Peptide Recognition的肽识别区域中的螺旋和β-转构象
拟南芥中甲基化/ORTHRUS (VIM/ORTH) 蛋白家族中含有PHD 指的变异体由哺乳动物 UHRF1 的功能同源物组成,是维持 DNA 甲基化所必需的。将序列与其他 PHD 手指的序列进行比较表明,VIM1 和 VIM3 PHD 可以通过保守的天冬氨酸介导的相互作用识别组蛋白 H3 (H3K4) 的赖氨酸 4。然而,我们的量热法和修饰的组蛋白肽阵列结合研究表明,VIM1 和 VIM3 PHD 手指均无法识别 H3K4 和其他组蛋白标记。在这里,我们报告了 VIM1 PHD 手指的 2.6 Å 分辨率晶体结构,并证明了与规范的 H3K4 结合 PHD 手指相比,假定的 H3 识别片段的显着结构变化。这些变化包括(i)H3A1结合区,(ii)与H3肽形成分子间β-折叠的β1链,(iii) 与 H3K4 的盐桥相互作用所涉及的含天冬氨酸基序,它们一起似乎取消了 VIM1 PHD 手指对 H3K4 的识别。为了了解 VIM1 PHD 中可能阻止组蛋白 H3 识别的结构特征改变的重要性,我们通过将 BHC80 PHD 的肽结合结构特征移植到 VIM1 PHD 上来模拟嵌合 VIM1 PHD(chmVIM1 PHD)。分子动力学模拟和元动力学分析表明 chmVIM1 PHD-H3 复合物是稳定的,并且还显示出类似于 BHC80 PHD-H3 复合物的分子间相互作用网络。总的来说,这项研究揭示了 VIM1 PHD 肽结合区的细微结构变化取消了组蛋白 H3 识别。它们一起似乎废除了 VIM1 PHD 手指对 H3K4 的识别。为了了解 VIM1 PHD 中可能阻止组蛋白 H3 识别的结构特征改变的重要性,我们通过将 BHC80 PHD 的肽结合结构特征移植到 VIM1 PHD 上来模拟嵌合 VIM1 PHD(chmVIM1 PHD)。分子动力学模拟和元动力学分析表明 chmVIM1 PHD-H3 复合物是稳定的,并且还显示出类似于 BHC80 PHD-H3 复合物的分子间相互作用网络。总的来说,这项研究揭示了 VIM1 PHD 肽结合区的细微结构变化取消了组蛋白 H3 识别。它们一起似乎废除了 VIM1 PHD 手指对 H3K4 的识别。为了了解 VIM1 PHD 中可能阻止组蛋白 H3 识别的结构特征改变的重要性,我们通过将 BHC80 PHD 的肽结合结构特征移植到 VIM1 PHD 上来模拟嵌合 VIM1 PHD(chmVIM1 PHD)。分子动力学模拟和元动力学分析表明 chmVIM1 PHD-H3 复合物是稳定的,并且还显示出类似于 BHC80 PHD-H3 复合物的分子间相互作用网络。总的来说,这项研究揭示了 VIM1 PHD 肽结合区的细微结构变化取消了组蛋白 H3 识别。我们通过将 BHC80 PHD 的肽结合结构特征移植到 VIM1 PHD 上来模拟嵌合 VIM1 PHD (chmVIM1 PHD)。分子动力学模拟和元动力学分析表明 chmVIM1 PHD-H3 复合物是稳定的,并且还显示出类似于 BHC80 PHD-H3 复合物的分子间相互作用网络。总的来说,这项研究揭示了 VIM1 PHD 肽结合区的细微结构变化取消了组蛋白 H3 识别。我们通过将 BHC80 PHD 的肽结合结构特征移植到 VIM1 PHD 上来模拟嵌合 VIM1 PHD (chmVIM1 PHD)。分子动力学模拟和元动力学分析表明 chmVIM1 PHD-H3 复合物是稳定的,并且还显示出类似于 BHC80 PHD-H3 复合物的分子间相互作用网络。总的来说,这项研究揭示了 VIM1 PHD 肽结合区的细微结构变化取消了组蛋白 H3 识别。
更新日期:2021-09-07
中文翻译:

VIM1 PHD Finger Abrogate H3K4 Peptide Recognition的肽识别区域中的螺旋和β-转构象
拟南芥中甲基化/ORTHRUS (VIM/ORTH) 蛋白家族中含有PHD 指的变异体由哺乳动物 UHRF1 的功能同源物组成,是维持 DNA 甲基化所必需的。将序列与其他 PHD 手指的序列进行比较表明,VIM1 和 VIM3 PHD 可以通过保守的天冬氨酸介导的相互作用识别组蛋白 H3 (H3K4) 的赖氨酸 4。然而,我们的量热法和修饰的组蛋白肽阵列结合研究表明,VIM1 和 VIM3 PHD 手指均无法识别 H3K4 和其他组蛋白标记。在这里,我们报告了 VIM1 PHD 手指的 2.6 Å 分辨率晶体结构,并证明了与规范的 H3K4 结合 PHD 手指相比,假定的 H3 识别片段的显着结构变化。这些变化包括(i)H3A1结合区,(ii)与H3肽形成分子间β-折叠的β1链,(iii) 与 H3K4 的盐桥相互作用所涉及的含天冬氨酸基序,它们一起似乎取消了 VIM1 PHD 手指对 H3K4 的识别。为了了解 VIM1 PHD 中可能阻止组蛋白 H3 识别的结构特征改变的重要性,我们通过将 BHC80 PHD 的肽结合结构特征移植到 VIM1 PHD 上来模拟嵌合 VIM1 PHD(chmVIM1 PHD)。分子动力学模拟和元动力学分析表明 chmVIM1 PHD-H3 复合物是稳定的,并且还显示出类似于 BHC80 PHD-H3 复合物的分子间相互作用网络。总的来说,这项研究揭示了 VIM1 PHD 肽结合区的细微结构变化取消了组蛋白 H3 识别。它们一起似乎废除了 VIM1 PHD 手指对 H3K4 的识别。为了了解 VIM1 PHD 中可能阻止组蛋白 H3 识别的结构特征改变的重要性,我们通过将 BHC80 PHD 的肽结合结构特征移植到 VIM1 PHD 上来模拟嵌合 VIM1 PHD(chmVIM1 PHD)。分子动力学模拟和元动力学分析表明 chmVIM1 PHD-H3 复合物是稳定的,并且还显示出类似于 BHC80 PHD-H3 复合物的分子间相互作用网络。总的来说,这项研究揭示了 VIM1 PHD 肽结合区的细微结构变化取消了组蛋白 H3 识别。它们一起似乎废除了 VIM1 PHD 手指对 H3K4 的识别。为了了解 VIM1 PHD 中可能阻止组蛋白 H3 识别的结构特征改变的重要性,我们通过将 BHC80 PHD 的肽结合结构特征移植到 VIM1 PHD 上来模拟嵌合 VIM1 PHD(chmVIM1 PHD)。分子动力学模拟和元动力学分析表明 chmVIM1 PHD-H3 复合物是稳定的,并且还显示出类似于 BHC80 PHD-H3 复合物的分子间相互作用网络。总的来说,这项研究揭示了 VIM1 PHD 肽结合区的细微结构变化取消了组蛋白 H3 识别。我们通过将 BHC80 PHD 的肽结合结构特征移植到 VIM1 PHD 上来模拟嵌合 VIM1 PHD (chmVIM1 PHD)。分子动力学模拟和元动力学分析表明 chmVIM1 PHD-H3 复合物是稳定的,并且还显示出类似于 BHC80 PHD-H3 复合物的分子间相互作用网络。总的来说,这项研究揭示了 VIM1 PHD 肽结合区的细微结构变化取消了组蛋白 H3 识别。我们通过将 BHC80 PHD 的肽结合结构特征移植到 VIM1 PHD 上来模拟嵌合 VIM1 PHD (chmVIM1 PHD)。分子动力学模拟和元动力学分析表明 chmVIM1 PHD-H3 复合物是稳定的,并且还显示出类似于 BHC80 PHD-H3 复合物的分子间相互作用网络。总的来说,这项研究揭示了 VIM1 PHD 肽结合区的细微结构变化取消了组蛋白 H3 识别。











































 京公网安备 11010802027423号
京公网安备 11010802027423号