当前位置:
X-MOL 学术
›
J. Biomed. Mater. Res. Part A
›
论文详情
Our official English website, www.x-mol.net, welcomes your
feedback! (Note: you will need to create a separate account there.)
Platelet microparticles load a repertory of miRNAs programmed to drive osteogenic phenotype
Journal of Biomedical Materials Research Part A ( IF 3.9 ) Pub Date : 2020-12-01 , DOI: 10.1002/jbm.a.37140 Marcel Rodrigues Ferreira 1 , Willian Fernando Zambuzzi 1
Journal of Biomedical Materials Research Part A ( IF 3.9 ) Pub Date : 2020-12-01 , DOI: 10.1002/jbm.a.37140 Marcel Rodrigues Ferreira 1 , Willian Fernando Zambuzzi 1
Affiliation
|
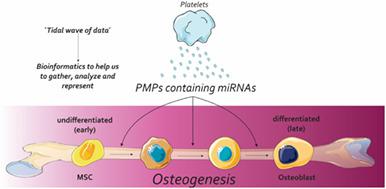
Autologous platelet-rich plasma accelerates bone healing by releasing biomolecules during their degranulation process, which are transported by vesicle-like structures called platelet microparticles (PMPs). However, the underlying mechanisms regulating the osteogenic differentiation by PMP-released miRs remain poorly understood and this prompted us to better address this issue. Thus, miRNAseq expression profiles (E-GEOD-76789) were downloaded from ArrayExpress database. GEO2R was performed to evaluate the differential expression, and mirnatap R package was used to find targets for differentially expressed miRNAs. An extend protein–protein (ePPI) network for osteogenic marker proteins was generated using String, and DAVID tools were used to perform gene ontology and KEGG pathway analysis from ePPI and miRNAs targets. Our data show that ePPI network was composed by 232 nodes and 2,175 edges, with a clustering coefficient of 0.546. MCODE was able to identify seven clusters contained in the ePPI network, and the two that presented a score above 10 were used in further analysis. Conversely, 15,944 different targets were found as down-expressed while 5,715 different targets were up-expressed. Among the downregulated 75 miRNAs, 70 have predicted targets present in the ePPI network, while the 21 upregulated miRNAs have 19 predicted targets in the ePPI network. Our study provides a registry of miRNAs that play a central role in regulating osteogenic phenotype, which might have potential therapeutic applications in bone regeneration and bone tissue engineering.
中文翻译:

血小板微粒加载一系列被编程以驱动成骨表型的 miRNA
自体富含血小板的血浆通过在脱粒过程中释放生物分子来加速骨愈合,这些生物分子由称为血小板微粒 (PMP) 的囊泡状结构运输。然而,PMP释放的miR调节成骨分化的潜在机制仍然知之甚少,这促使我们更好地解决这个问题。因此,从 ArrayExpress 数据库下载了 miRNAseq 表达谱 (E-GEOD-76789)。GEO2R 用于评估差异表达,mirnatap R 包用于寻找差异表达的 miRNA 的目标。使用 String 生成用于成骨标记蛋白的扩展蛋白-蛋白 (ePPI) 网络,并使用 DAVID 工具对 ePPI 和 miRNA 靶标进行基因本体和 KEGG 通路分析。我们的数据显示,ePPI网络由232个节点和2175条边组成,聚类系数为0.546。MCODE 能够识别 ePPI 网络中包含的 7 个集群,并且将得分高于 10 的两个集群用于进一步分析。相反,发现 15,944 个不同的目标被下调,而 5,715 个不同的目标被上调。在下调的 75 个 miRNA 中,有 70 个预测目标存在于 ePPI 网络中,而 21 个上调的 miRNA 在 ePPI 网络中有 19 个预测目标。我们的研究提供了一个在调节成骨表型中起核心作用的 miRNA 登记表,这可能在骨再生和骨组织工程中具有潜在的治疗应用。MCODE 能够识别 ePPI 网络中包含的 7 个集群,并且将得分高于 10 的两个集群用于进一步分析。相反,发现 15,944 个不同的目标被下调,而 5,715 个不同的目标被上调。在下调的 75 个 miRNA 中,有 70 个预测目标存在于 ePPI 网络中,而 21 个上调的 miRNA 在 ePPI 网络中有 19 个预测目标。我们的研究提供了一个在调节成骨表型中起核心作用的 miRNA 登记表,这可能在骨再生和骨组织工程中具有潜在的治疗应用。MCODE 能够识别 ePPI 网络中包含的 7 个集群,并且将得分高于 10 的两个集群用于进一步分析。相反,发现 15,944 个不同的目标被下调,而 5,715 个不同的目标被上调。在下调的 75 个 miRNA 中,有 70 个预测目标存在于 ePPI 网络中,而 21 个上调的 miRNA 在 ePPI 网络中有 19 个预测目标。我们的研究提供了一个在调节成骨表型中起核心作用的 miRNA 登记表,这可能在骨再生和骨组织工程中具有潜在的治疗应用。上表达了 715 个不同的目标。在下调的 75 个 miRNA 中,有 70 个预测目标存在于 ePPI 网络中,而 21 个上调的 miRNA 在 ePPI 网络中有 19 个预测目标。我们的研究提供了一个在调节成骨表型中起核心作用的 miRNA 登记表,这可能在骨再生和骨组织工程中具有潜在的治疗应用。上表达了 715 个不同的目标。在下调的 75 个 miRNA 中,有 70 个预测目标存在于 ePPI 网络中,而 21 个上调的 miRNA 在 ePPI 网络中有 19 个预测目标。我们的研究提供了一个在调节成骨表型中起核心作用的 miRNA 登记表,这可能在骨再生和骨组织工程中具有潜在的治疗应用。
更新日期:2020-12-01
中文翻译:

血小板微粒加载一系列被编程以驱动成骨表型的 miRNA
自体富含血小板的血浆通过在脱粒过程中释放生物分子来加速骨愈合,这些生物分子由称为血小板微粒 (PMP) 的囊泡状结构运输。然而,PMP释放的miR调节成骨分化的潜在机制仍然知之甚少,这促使我们更好地解决这个问题。因此,从 ArrayExpress 数据库下载了 miRNAseq 表达谱 (E-GEOD-76789)。GEO2R 用于评估差异表达,mirnatap R 包用于寻找差异表达的 miRNA 的目标。使用 String 生成用于成骨标记蛋白的扩展蛋白-蛋白 (ePPI) 网络,并使用 DAVID 工具对 ePPI 和 miRNA 靶标进行基因本体和 KEGG 通路分析。我们的数据显示,ePPI网络由232个节点和2175条边组成,聚类系数为0.546。MCODE 能够识别 ePPI 网络中包含的 7 个集群,并且将得分高于 10 的两个集群用于进一步分析。相反,发现 15,944 个不同的目标被下调,而 5,715 个不同的目标被上调。在下调的 75 个 miRNA 中,有 70 个预测目标存在于 ePPI 网络中,而 21 个上调的 miRNA 在 ePPI 网络中有 19 个预测目标。我们的研究提供了一个在调节成骨表型中起核心作用的 miRNA 登记表,这可能在骨再生和骨组织工程中具有潜在的治疗应用。MCODE 能够识别 ePPI 网络中包含的 7 个集群,并且将得分高于 10 的两个集群用于进一步分析。相反,发现 15,944 个不同的目标被下调,而 5,715 个不同的目标被上调。在下调的 75 个 miRNA 中,有 70 个预测目标存在于 ePPI 网络中,而 21 个上调的 miRNA 在 ePPI 网络中有 19 个预测目标。我们的研究提供了一个在调节成骨表型中起核心作用的 miRNA 登记表,这可能在骨再生和骨组织工程中具有潜在的治疗应用。MCODE 能够识别 ePPI 网络中包含的 7 个集群,并且将得分高于 10 的两个集群用于进一步分析。相反,发现 15,944 个不同的目标被下调,而 5,715 个不同的目标被上调。在下调的 75 个 miRNA 中,有 70 个预测目标存在于 ePPI 网络中,而 21 个上调的 miRNA 在 ePPI 网络中有 19 个预测目标。我们的研究提供了一个在调节成骨表型中起核心作用的 miRNA 登记表,这可能在骨再生和骨组织工程中具有潜在的治疗应用。上表达了 715 个不同的目标。在下调的 75 个 miRNA 中,有 70 个预测目标存在于 ePPI 网络中,而 21 个上调的 miRNA 在 ePPI 网络中有 19 个预测目标。我们的研究提供了一个在调节成骨表型中起核心作用的 miRNA 登记表,这可能在骨再生和骨组织工程中具有潜在的治疗应用。上表达了 715 个不同的目标。在下调的 75 个 miRNA 中,有 70 个预测目标存在于 ePPI 网络中,而 21 个上调的 miRNA 在 ePPI 网络中有 19 个预测目标。我们的研究提供了一个在调节成骨表型中起核心作用的 miRNA 登记表,这可能在骨再生和骨组织工程中具有潜在的治疗应用。











































 京公网安备 11010802027423号
京公网安备 11010802027423号