当前位置:
X-MOL 学术
›
Biopolymers
›
论文详情
Our official English website, www.x-mol.net, welcomes your
feedback! (Note: you will need to create a separate account there.)
6‐phenylpyrrolocytosine as a fluorescent probe to examine nucleotide flipping catalyzed by a DNA repair protein
Biopolymers ( IF 3.2 ) Pub Date : 2020-10-24 , DOI: 10.1002/bip.23405 Delshanee Kotandeniya 1 , Melanie S Rogers 2 , Jenna Fernandez 1 , Sreenivas Kanugula 3 , Robert H E Hudson 4 , Freddys Rodriguez 1 , John D Lipscomb 2 , Natalia Tretyakova 1
Biopolymers ( IF 3.2 ) Pub Date : 2020-10-24 , DOI: 10.1002/bip.23405 Delshanee Kotandeniya 1 , Melanie S Rogers 2 , Jenna Fernandez 1 , Sreenivas Kanugula 3 , Robert H E Hudson 4 , Freddys Rodriguez 1 , John D Lipscomb 2 , Natalia Tretyakova 1
Affiliation
|
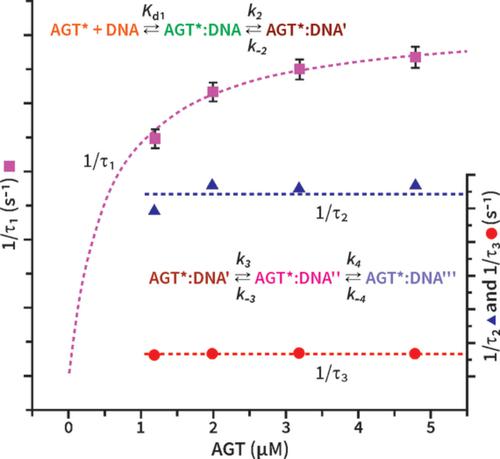
Cellular exposure to tobacco‐specific nitrosamines causes formation of promutagenic O6‐[4‐oxo‐4‐(3‐pyridyl)but‐1‐yl]guanine (O6‐POB‐G) and O6‐methylguanine (O6‐Me‐G) adducts in DNA. These adducts can be directly repaired by O6‐alkylguanine‐DNA alkyltransferase (AGT). Repair begins by flipping the damaged base out of the DNA helix. AGT binding and base‐flipping have been previously studied using pyrrolocytosine as a fluorescent probe paired to the O6‐alkylguanine lesion, but low fluorescence yield limited the resolution of steps in the repair process. Here, we utilize the highly fluorescent 6‐phenylpyrrolo‐2′‐deoxycytidine (6‐phenylpyrrolo‐C) to investigate AGT‐DNA interactions. Synthetic oligodeoxynucleotide duplexes containing O6‐POB‐G and O6‐Me‐G adducts were placed within the CpG sites of codons 158, 245, and 248 of the p53 tumor suppressor gene and base‐paired to 6‐phenylpyrrolo‐C in the opposite strand. Neighboring cytosine was either unmethylated or methylated. Stopped‐flow fluorescence measurements were performed by mixing the DNA duplexes with C145A or R128G AGT variants. We observe a rapid, two‐step, nearly irreversible binding of AGT to DNA followed by two slower steps, one of which is base‐flipping. Placing 5‐methylcytosine immediately 5′ to the alkylated guanosine causes a reduction in rate constant of nucleotide flipping. O6‐POB‐G at codon 158 decreased the base flipping rate constant by 3.5‐fold compared with O6‐Me‐G at the same position. A similar effect was not observed at other codons.
中文翻译:

6-苯基吡咯胞嘧啶作为荧光探针检测 DNA 修复蛋白催化的核苷酸翻转
细胞暴露于烟草特异性亚硝胺导致促突变 O6-[4-oxo-4-(3-pyridyl)but-1-yl] 鸟嘌呤 (O6-POB-G) 和 O6-甲基鸟嘌呤 (O6-Me-G) 的形成DNA 中的加合物。这些加合物可以被 O6-烷基鸟嘌呤-DNA 烷基转移酶 (AGT) 直接修复。修复始于将受损碱基从 DNA 螺旋中翻转出来。先前已经使用吡咯胞嘧啶作为与 O6-烷基鸟嘌呤病变配对的荧光探针研究了 AGT 结合和碱基翻转,但低荧光产率限制了修复过程中步骤的分辨率。在这里,我们利用高荧光 6-苯基吡咯并-2'-脱氧胞苷(6-苯基吡咯并-C)来研究 AGT-DNA 相互作用。将含有 O6-POB-G 和 O6-Me-G 加合物的合成寡脱氧核苷酸双链体置于密码子 158、245、和 p53 肿瘤抑制基因的 248 个,与相反链中的 6-苯基吡咯并-C 碱基配对。相邻胞嘧啶未甲基化或甲基化。通过将 DNA 双链体与 C145A 或 R128G AGT 变体混合来进行停流荧光测量。我们观察到 AGT 与 DNA 的快速、两步、几乎不可逆的结合,然后是两个较慢的步骤,其中一个是碱基翻转。将 5-甲基胞嘧啶紧邻烷基化鸟苷的 5' 端会导致核苷酸翻转的速率常数降低。与相同位置的 O6-Me-G 相比,密码子 158 处的 O6-POB-G 使碱基翻转速率常数降低了 3.5 倍。在其他密码子中没有观察到类似的效果。通过将 DNA 双链体与 C145A 或 R128G AGT 变体混合来进行停流荧光测量。我们观察到 AGT 与 DNA 的快速、两步、几乎不可逆的结合,然后是两个较慢的步骤,其中一个是碱基翻转。将 5-甲基胞嘧啶紧邻烷基化鸟苷的 5' 端会导致核苷酸翻转的速率常数降低。与相同位置的 O6-Me-G 相比,密码子 158 处的 O6-POB-G 使碱基翻转速率常数降低了 3.5 倍。在其他密码子中没有观察到类似的效果。通过将 DNA 双链体与 C145A 或 R128G AGT 变体混合来进行停流荧光测量。我们观察到 AGT 与 DNA 的快速、两步、几乎不可逆的结合,然后是两个较慢的步骤,其中一个是碱基翻转。将 5-甲基胞嘧啶紧邻烷基化鸟苷的 5' 端会导致核苷酸翻转的速率常数降低。与相同位置的 O6-Me-G 相比,密码子 158 处的 O6-POB-G 使碱基翻转速率常数降低了 3.5 倍。在其他密码子中没有观察到类似的效果。将 5-甲基胞嘧啶紧邻烷基化鸟苷的 5' 端会导致核苷酸翻转的速率常数降低。与相同位置的 O6-Me-G 相比,密码子 158 处的 O6-POB-G 使碱基翻转速率常数降低了 3.5 倍。在其他密码子中没有观察到类似的效果。将 5-甲基胞嘧啶紧邻烷基化鸟苷的 5' 端会导致核苷酸翻转的速率常数降低。与相同位置的 O6-Me-G 相比,密码子 158 处的 O6-POB-G 使碱基翻转速率常数降低了 3.5 倍。在其他密码子中没有观察到类似的效果。
更新日期:2020-10-24
中文翻译:

6-苯基吡咯胞嘧啶作为荧光探针检测 DNA 修复蛋白催化的核苷酸翻转
细胞暴露于烟草特异性亚硝胺导致促突变 O6-[4-oxo-4-(3-pyridyl)but-1-yl] 鸟嘌呤 (O6-POB-G) 和 O6-甲基鸟嘌呤 (O6-Me-G) 的形成DNA 中的加合物。这些加合物可以被 O6-烷基鸟嘌呤-DNA 烷基转移酶 (AGT) 直接修复。修复始于将受损碱基从 DNA 螺旋中翻转出来。先前已经使用吡咯胞嘧啶作为与 O6-烷基鸟嘌呤病变配对的荧光探针研究了 AGT 结合和碱基翻转,但低荧光产率限制了修复过程中步骤的分辨率。在这里,我们利用高荧光 6-苯基吡咯并-2'-脱氧胞苷(6-苯基吡咯并-C)来研究 AGT-DNA 相互作用。将含有 O6-POB-G 和 O6-Me-G 加合物的合成寡脱氧核苷酸双链体置于密码子 158、245、和 p53 肿瘤抑制基因的 248 个,与相反链中的 6-苯基吡咯并-C 碱基配对。相邻胞嘧啶未甲基化或甲基化。通过将 DNA 双链体与 C145A 或 R128G AGT 变体混合来进行停流荧光测量。我们观察到 AGT 与 DNA 的快速、两步、几乎不可逆的结合,然后是两个较慢的步骤,其中一个是碱基翻转。将 5-甲基胞嘧啶紧邻烷基化鸟苷的 5' 端会导致核苷酸翻转的速率常数降低。与相同位置的 O6-Me-G 相比,密码子 158 处的 O6-POB-G 使碱基翻转速率常数降低了 3.5 倍。在其他密码子中没有观察到类似的效果。通过将 DNA 双链体与 C145A 或 R128G AGT 变体混合来进行停流荧光测量。我们观察到 AGT 与 DNA 的快速、两步、几乎不可逆的结合,然后是两个较慢的步骤,其中一个是碱基翻转。将 5-甲基胞嘧啶紧邻烷基化鸟苷的 5' 端会导致核苷酸翻转的速率常数降低。与相同位置的 O6-Me-G 相比,密码子 158 处的 O6-POB-G 使碱基翻转速率常数降低了 3.5 倍。在其他密码子中没有观察到类似的效果。通过将 DNA 双链体与 C145A 或 R128G AGT 变体混合来进行停流荧光测量。我们观察到 AGT 与 DNA 的快速、两步、几乎不可逆的结合,然后是两个较慢的步骤,其中一个是碱基翻转。将 5-甲基胞嘧啶紧邻烷基化鸟苷的 5' 端会导致核苷酸翻转的速率常数降低。与相同位置的 O6-Me-G 相比,密码子 158 处的 O6-POB-G 使碱基翻转速率常数降低了 3.5 倍。在其他密码子中没有观察到类似的效果。将 5-甲基胞嘧啶紧邻烷基化鸟苷的 5' 端会导致核苷酸翻转的速率常数降低。与相同位置的 O6-Me-G 相比,密码子 158 处的 O6-POB-G 使碱基翻转速率常数降低了 3.5 倍。在其他密码子中没有观察到类似的效果。将 5-甲基胞嘧啶紧邻烷基化鸟苷的 5' 端会导致核苷酸翻转的速率常数降低。与相同位置的 O6-Me-G 相比,密码子 158 处的 O6-POB-G 使碱基翻转速率常数降低了 3.5 倍。在其他密码子中没有观察到类似的效果。











































 京公网安备 11010802027423号
京公网安备 11010802027423号