当前位置:
X-MOL 学术
›
J. Phys. Chem. C
›
论文详情
Our official English website, www.x-mol.net, welcomes your
feedback! (Note: you will need to create a separate account there.)
Molecular Self-Assembly: Quantifying the Balance between Intermolecular Attraction and Repulsion from Distance and Length Distributions
The Journal of Physical Chemistry C ( IF 3.3 ) Pub Date : 2020-09-03 , DOI: 10.1021/acs.jpcc.0c06676 Christoph Schiel 1 , Maximilian Vogtland 2 , Ralf Bechstein 2 , Angelika Kühnle 2 , Philipp Maass 1
The Journal of Physical Chemistry C ( IF 3.3 ) Pub Date : 2020-09-03 , DOI: 10.1021/acs.jpcc.0c06676 Christoph Schiel 1 , Maximilian Vogtland 2 , Ralf Bechstein 2 , Angelika Kühnle 2 , Philipp Maass 1
Affiliation
|
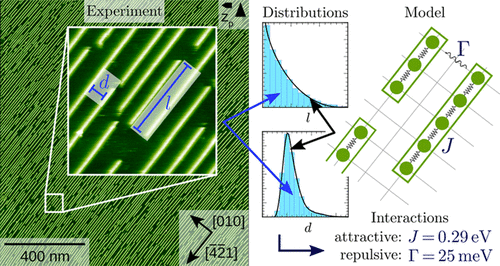
Molecular self-assembly on surfaces constitutes a powerful method for creating tailor-made surface structures with dedicated functionalities. Varying the intermolecular interactions allows for tuning the resulting molecular structures in a rational fashion. So far, however, the discussion on the involved intermolecular interactions is often limited to attractive forces only. In real systems, the intermolecular interaction can be composed of both attractive and repulsive forces. Adjusting the balance between these interactions provides a promising strategy for extending the structural variety of molecular self-assembly on surfaces. However, this strategy relies on a method to quantify the involved interactions. Here, we investigate a molecular model system of 3-hydroxybenzoic acid (3-HBA) molecules on calcite (10.4) in a ultrahigh vacuum. This system offers both anisotropic short-range attraction and long-range repulsive dipolar interactions between molecules, resulting in the self-assembly of molecular stripes. We analyze the stripe-to-stripe distance distribution and the stripe length distribution and compare these distributions with analytical expressions from an anisotropic Ising model with additional repulsive interactions. We show that this approach allows the extraction of quantitative information about the strength of the attractive and repulsive interactions. Our work demonstrates how the detailed analysis of the self-assembled structures can be used to obtain quantitative insight into the molecule–molecule interactions.
中文翻译:

分子自组装:根据距离和长度分布量化分子间吸引力和排斥力之间的平衡
表面上的分子自组装构成了创建具有专用功能的量身定制表面结构的强大方法。分子间相互作用的变化允许以合理的方式调节所得的分子结构。但是,到目前为止,有关分子间相互作用的讨论通常仅限于吸引力。在实际系统中,分子间的相互作用可以由吸引力和排斥力组成。调节这些相互作用之间的平衡为扩展表面上分子自组装的结构多样性提供了一种有前途的策略。但是,此策略依赖于一种量化所涉及交互的方法。在这里,我们研究了在超高真空下方解石(10.4)上的3-羟基苯甲酸(3-HBA)分子的分子模型系统。该系统提供了分子之间的各向异性短程吸引和长程排斥偶极相互作用,从而导致分子条纹的自组装。我们分析了条带到条带的距离分布和条带长度分布,并将这些分布与各向异性Ising模型具有附加排斥相互作用的解析表达式进行了比较。我们表明,这种方法可以提取有关吸引力和排斥性相互作用强度的定量信息。我们的工作表明,如何使用对自组装结构的详细分析来获得对分子-分子相互作用的定量了解。导致分子条纹的自组装。我们分析了条带到条带的距离分布和条带长度分布,并将这些分布与各向异性Ising模型具有附加排斥相互作用的解析表达式进行了比较。我们表明,这种方法可以提取有关吸引力和排斥性相互作用强度的定量信息。我们的工作证明了如何使用对自组装结构的详细分析来获得对分子与分子相互作用的定量分析。导致分子条纹的自组装。我们分析了条带到条带的距离分布和条带长度分布,并将这些分布与各向异性Ising模型具有附加排斥相互作用的解析表达式进行了比较。我们表明,这种方法可以提取有关吸引力和排斥性相互作用强度的定量信息。我们的工作表明,如何使用对自组装结构的详细分析来获得对分子-分子相互作用的定量了解。我们表明,这种方法可以提取有关吸引力和排斥性相互作用强度的定量信息。我们的工作表明,如何使用对自组装结构的详细分析来获得对分子-分子相互作用的定量了解。我们表明,这种方法可以提取有关吸引力和排斥性相互作用强度的定量信息。我们的工作表明,如何使用对自组装结构的详细分析来获得对分子-分子相互作用的定量了解。
更新日期:2020-10-02
中文翻译:

分子自组装:根据距离和长度分布量化分子间吸引力和排斥力之间的平衡
表面上的分子自组装构成了创建具有专用功能的量身定制表面结构的强大方法。分子间相互作用的变化允许以合理的方式调节所得的分子结构。但是,到目前为止,有关分子间相互作用的讨论通常仅限于吸引力。在实际系统中,分子间的相互作用可以由吸引力和排斥力组成。调节这些相互作用之间的平衡为扩展表面上分子自组装的结构多样性提供了一种有前途的策略。但是,此策略依赖于一种量化所涉及交互的方法。在这里,我们研究了在超高真空下方解石(10.4)上的3-羟基苯甲酸(3-HBA)分子的分子模型系统。该系统提供了分子之间的各向异性短程吸引和长程排斥偶极相互作用,从而导致分子条纹的自组装。我们分析了条带到条带的距离分布和条带长度分布,并将这些分布与各向异性Ising模型具有附加排斥相互作用的解析表达式进行了比较。我们表明,这种方法可以提取有关吸引力和排斥性相互作用强度的定量信息。我们的工作表明,如何使用对自组装结构的详细分析来获得对分子-分子相互作用的定量了解。导致分子条纹的自组装。我们分析了条带到条带的距离分布和条带长度分布,并将这些分布与各向异性Ising模型具有附加排斥相互作用的解析表达式进行了比较。我们表明,这种方法可以提取有关吸引力和排斥性相互作用强度的定量信息。我们的工作证明了如何使用对自组装结构的详细分析来获得对分子与分子相互作用的定量分析。导致分子条纹的自组装。我们分析了条带到条带的距离分布和条带长度分布,并将这些分布与各向异性Ising模型具有附加排斥相互作用的解析表达式进行了比较。我们表明,这种方法可以提取有关吸引力和排斥性相互作用强度的定量信息。我们的工作表明,如何使用对自组装结构的详细分析来获得对分子-分子相互作用的定量了解。我们表明,这种方法可以提取有关吸引力和排斥性相互作用强度的定量信息。我们的工作表明,如何使用对自组装结构的详细分析来获得对分子-分子相互作用的定量了解。我们表明,这种方法可以提取有关吸引力和排斥性相互作用强度的定量信息。我们的工作表明,如何使用对自组装结构的详细分析来获得对分子-分子相互作用的定量了解。











































 京公网安备 11010802027423号
京公网安备 11010802027423号