当前位置:
X-MOL 学术
›
J. Comput. Chem.
›
论文详情
Our official English website, www.x-mol.net, welcomes your
feedback! (Note: you will need to create a separate account there.)
HCVpred: A web server for predicting the bioactivity of hepatitis C virus NS5B inhibitors
Journal of Computational Chemistry ( IF 3.4 ) Pub Date : 2020-05-25 , DOI: 10.1002/jcc.26223 Aijaz Ahmad Malik 1 , Chuleeporn Phanus-Umporn 1 , Nalini Schaduangrat 1 , Watshara Shoombuatong 1 , Chartchalerm Isarankura-Na-Ayudhya 2 , Chanin Nantasenamat 1
Journal of Computational Chemistry ( IF 3.4 ) Pub Date : 2020-05-25 , DOI: 10.1002/jcc.26223 Aijaz Ahmad Malik 1 , Chuleeporn Phanus-Umporn 1 , Nalini Schaduangrat 1 , Watshara Shoombuatong 1 , Chartchalerm Isarankura-Na-Ayudhya 2 , Chanin Nantasenamat 1
Affiliation
|
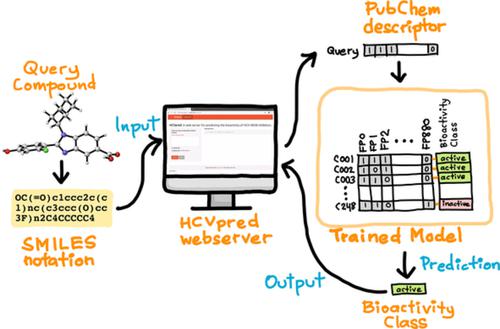
Hepatitis C virus (HCV) is one of the major causes of liver disease affecting an estimated 170 million people culminating in 300,000 deaths from cirrhosis or liver cancer. NS5B is one of three potential therapeutic targets against HCV (i.e., the other two being NS3/4A and NS5A) that is central to viral replication. In this study, we developed a classification structure–activity relationship (CSAR) model for identifying substructures giving rise to anti‐HCV activities among a set of 578 non‐redundant compounds. NS5B inhibitors were described by a set of 12 fingerprint descriptors and predictive models were constructed from 100 independent data splits using the random forest algorithm. The modelability (MODI index) of the data set was determined to be robust with a value of 0.88 exceeding established threshold of 0.65. The predictive performance was deduced by the accuracy, sensitivity, specificity, and Matthews correlation coefficient, which was found to be statistically robust (i.e., the former three parameters afforded values in excess of 0.8 while the latter statistical parameter provided a value >0.7). An in‐depth analysis of the top 20 important descriptors revealed that aromatic ring and alkyl side chains are important for NS5B inhibition. Finally, the predictive model is deployed as a publicly accessible HCVpred web server (available at http://codes.bio/hcvpred/) that would allow users to predict the biological activity as being active or inactive against HCV NS5B. Thus, the knowledge and web server presented herein can be used in the design of more potent and specific drugs against the HCV NS5B.
中文翻译:

HCVpred:用于预测丙型肝炎病毒 NS5B 抑制剂生物活性的网络服务器
丙型肝炎病毒 (HCV) 是导致肝病的主要原因之一,估计影响 1.7 亿人,最终导致 300,000 人死于肝硬化或肝癌。NS5B 是针对 HCV 的三个潜在治疗靶点之一(即另外两个是 NS3/4A 和 NS5A),它是病毒复制的中心。在这项研究中,我们开发了一个分类构效关系 (CSAR) 模型,用于识别在一组 578 种非冗余化合物中引起抗 HCV 活性的亚结构。NS5B 抑制剂由一组 12 个指纹描述符描述,并使用随机森林算法从 100 个独立的数据拆分构建预测模型。数据集的可建模性(MODI 指数)被确定为稳健,其值为 0.88,超过了既定阈值 0.65。预测性能由准确性、敏感性、特异性和 Matthews 相关系数推断,这些系数在统计学上是稳健的(即,前三个参数提供的值超过 0.8,而后一个统计参数提供的值 >0.7)。对前 20 个重要描述符的深入分析表明,芳环和烷基侧链对 NS5B 抑制很重要。最后,预测模型被部署为可公开访问的 HCVpred 网络服务器(可在 http://codes.bio/hcvpred/ 获得),允许用户预测生物活性对 HCV NS5B 的活性或非活性。因此,本文介绍的知识和网络服务器可用于设计针对 HCV NS5B 的更有效和特异性的药物。和 Matthews 相关系数,发现其具有统计稳健性(即,前三个参数提供的值超过 0.8,而后一个统计参数提供的值 > 0.7)。对前 20 个重要描述符的深入分析表明,芳环和烷基侧链对 NS5B 抑制很重要。最后,预测模型被部署为可公开访问的 HCVpred 网络服务器(可在 http://codes.bio/hcvpred/ 获得),允许用户预测生物活性对 HCV NS5B 的活性或非活性。因此,本文介绍的知识和网络服务器可用于设计针对 HCV NS5B 的更有效和特异性的药物。和 Matthews 相关系数,发现其具有统计稳健性(即,前三个参数提供的值超过 0.8,而后一个统计参数提供的值 > 0.7)。对前 20 个重要描述符的深入分析表明,芳环和烷基侧链对 NS5B 抑制很重要。最后,预测模型被部署为可公开访问的 HCVpred 网络服务器(可在 http://codes.bio/hcvpred/ 获得),允许用户预测生物活性对 HCV NS5B 的活性或非活性。因此,本文介绍的知识和网络服务器可用于设计针对 HCV NS5B 的更有效和特异性的药物。前三个参数提供的值超过 0.8,而后一个统计参数提供的值 > 0.7)。对前 20 个重要描述符的深入分析表明,芳环和烷基侧链对 NS5B 抑制很重要。最后,预测模型被部署为可公开访问的 HCVpred 网络服务器(可在 http://codes.bio/hcvpred/ 获得),允许用户预测生物活性对 HCV NS5B 的活性或非活性。因此,本文介绍的知识和网络服务器可用于设计针对 HCV NS5B 的更有效和特异性的药物。前三个参数提供的值超过 0.8,而后一个统计参数提供的值 > 0.7)。对前 20 个重要描述符的深入分析表明,芳环和烷基侧链对 NS5B 抑制很重要。最后,预测模型被部署为可公开访问的 HCVpred 网络服务器(可在 http://codes.bio/hcvpred/ 获得),允许用户预测生物活性对 HCV NS5B 的活性或非活性。因此,本文介绍的知识和网络服务器可用于设计针对 HCV NS5B 的更有效和特异性的药物。预测模型部署为可公开访问的 HCVpred Web 服务器(可在 http://codes.bio/hcvpred/ 获得),允许用户预测生物活性对 HCV NS5B 的活性或非活性。因此,本文介绍的知识和网络服务器可用于设计针对 HCV NS5B 的更有效和特异性的药物。预测模型部署为可公开访问的 HCVpred Web 服务器(可在 http://codes.bio/hcvpred/ 获得),允许用户预测生物活性对 HCV NS5B 的活性或非活性。因此,本文介绍的知识和网络服务器可用于设计针对 HCV NS5B 的更有效和特异性的药物。
更新日期:2020-05-25
中文翻译:

HCVpred:用于预测丙型肝炎病毒 NS5B 抑制剂生物活性的网络服务器
丙型肝炎病毒 (HCV) 是导致肝病的主要原因之一,估计影响 1.7 亿人,最终导致 300,000 人死于肝硬化或肝癌。NS5B 是针对 HCV 的三个潜在治疗靶点之一(即另外两个是 NS3/4A 和 NS5A),它是病毒复制的中心。在这项研究中,我们开发了一个分类构效关系 (CSAR) 模型,用于识别在一组 578 种非冗余化合物中引起抗 HCV 活性的亚结构。NS5B 抑制剂由一组 12 个指纹描述符描述,并使用随机森林算法从 100 个独立的数据拆分构建预测模型。数据集的可建模性(MODI 指数)被确定为稳健,其值为 0.88,超过了既定阈值 0.65。预测性能由准确性、敏感性、特异性和 Matthews 相关系数推断,这些系数在统计学上是稳健的(即,前三个参数提供的值超过 0.8,而后一个统计参数提供的值 >0.7)。对前 20 个重要描述符的深入分析表明,芳环和烷基侧链对 NS5B 抑制很重要。最后,预测模型被部署为可公开访问的 HCVpred 网络服务器(可在 http://codes.bio/hcvpred/ 获得),允许用户预测生物活性对 HCV NS5B 的活性或非活性。因此,本文介绍的知识和网络服务器可用于设计针对 HCV NS5B 的更有效和特异性的药物。和 Matthews 相关系数,发现其具有统计稳健性(即,前三个参数提供的值超过 0.8,而后一个统计参数提供的值 > 0.7)。对前 20 个重要描述符的深入分析表明,芳环和烷基侧链对 NS5B 抑制很重要。最后,预测模型被部署为可公开访问的 HCVpred 网络服务器(可在 http://codes.bio/hcvpred/ 获得),允许用户预测生物活性对 HCV NS5B 的活性或非活性。因此,本文介绍的知识和网络服务器可用于设计针对 HCV NS5B 的更有效和特异性的药物。和 Matthews 相关系数,发现其具有统计稳健性(即,前三个参数提供的值超过 0.8,而后一个统计参数提供的值 > 0.7)。对前 20 个重要描述符的深入分析表明,芳环和烷基侧链对 NS5B 抑制很重要。最后,预测模型被部署为可公开访问的 HCVpred 网络服务器(可在 http://codes.bio/hcvpred/ 获得),允许用户预测生物活性对 HCV NS5B 的活性或非活性。因此,本文介绍的知识和网络服务器可用于设计针对 HCV NS5B 的更有效和特异性的药物。前三个参数提供的值超过 0.8,而后一个统计参数提供的值 > 0.7)。对前 20 个重要描述符的深入分析表明,芳环和烷基侧链对 NS5B 抑制很重要。最后,预测模型被部署为可公开访问的 HCVpred 网络服务器(可在 http://codes.bio/hcvpred/ 获得),允许用户预测生物活性对 HCV NS5B 的活性或非活性。因此,本文介绍的知识和网络服务器可用于设计针对 HCV NS5B 的更有效和特异性的药物。前三个参数提供的值超过 0.8,而后一个统计参数提供的值 > 0.7)。对前 20 个重要描述符的深入分析表明,芳环和烷基侧链对 NS5B 抑制很重要。最后,预测模型被部署为可公开访问的 HCVpred 网络服务器(可在 http://codes.bio/hcvpred/ 获得),允许用户预测生物活性对 HCV NS5B 的活性或非活性。因此,本文介绍的知识和网络服务器可用于设计针对 HCV NS5B 的更有效和特异性的药物。预测模型部署为可公开访问的 HCVpred Web 服务器(可在 http://codes.bio/hcvpred/ 获得),允许用户预测生物活性对 HCV NS5B 的活性或非活性。因此,本文介绍的知识和网络服务器可用于设计针对 HCV NS5B 的更有效和特异性的药物。预测模型部署为可公开访问的 HCVpred Web 服务器(可在 http://codes.bio/hcvpred/ 获得),允许用户预测生物活性对 HCV NS5B 的活性或非活性。因此,本文介绍的知识和网络服务器可用于设计针对 HCV NS5B 的更有效和特异性的药物。











































 京公网安备 11010802027423号
京公网安备 11010802027423号