当前位置:
X-MOL 学术
›
Ecol Modell
›
论文详情
Our official English website, www.x-mol.net, welcomes your
feedback! (Note: you will need to create a separate account there.)
Use of transcriptomic data to inform biophysical models via Bayesian networks
Ecological Modelling ( IF 2.6 ) Pub Date : 2020-08-01 , DOI: 10.1016/j.ecolmodel.2020.109086 C.R. Guadagno , D. Millar , R. Lai , D.S. Mackay , J.R. Pleban , C.R. McClung , C. Weinig , D.R. Wang , B.E. Ewers
Ecological Modelling ( IF 2.6 ) Pub Date : 2020-08-01 , DOI: 10.1016/j.ecolmodel.2020.109086 C.R. Guadagno , D. Millar , R. Lai , D.S. Mackay , J.R. Pleban , C.R. McClung , C. Weinig , D.R. Wang , B.E. Ewers
|
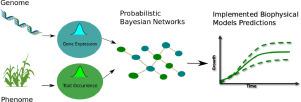
Abstract Process-based models of plant productivity provide a means of yield prediction that can inform best practices in agriculture and land management. However, current biophysical models fail in capturing both genotypic and phenotypic variation under changing environmental conditions. Physiological traits affecting final yield and ecosystem productivity are the result of gene expression, protein translation, and metabolite formation which are controlled by specific alleles acting alone and in response to time and environmental cues. Biophysical interactions take place simultaneously across several scales of time and space, giving rise to high levels of complexity and limiting the predictive capacity of existing analytical approaches. While statistical methods can partially quantify genotype-by-environment connections, biophysical process models miss exploring genotypic diversity and they cannot currently be implemented with -omics data, i.e. the entire collection of entities such as gene transcripts, metabolites, and proteins. Here we present a novel framework that utilizes Bayesian networks to provide probabilities that link gene expression levels to trait occurrence. We propose the use of these gained probabilities to inform parameters of existing biophysical models. Merging transcriptomic information into process-based models allows for greater integration of empirical data and processes across different scales in complex systems, while increasing the likelihood of growth predictions under unknown environmental conditions.
中文翻译:

使用转录组数据通过贝叶斯网络为生物物理模型提供信息
摘要 基于过程的植物生产力模型提供了一种预测产量的方法,可以为农业和土地管理的最佳实践提供信息。然而,当前的生物物理模型无法在不断变化的环境条件下捕获基因型和表型变异。影响最终产量和生态系统生产力的生理性状是基因表达、蛋白质翻译和代谢物形成的结果,它们由特定的等位基因单独控制并响应时间和环境线索。生物物理相互作用在多个时间和空间尺度上同时发生,导致高度复杂性并限制了现有分析方法的预测能力。虽然统计方法可以部分量化基因型与环境的联系,生物物理过程模型错过了对基因型多样性的探索,它们目前无法通过组学数据实现,即基因转录、代谢物和蛋白质等实体的整个集合。在这里,我们提出了一个新的框架,它利用贝叶斯网络提供将基因表达水平与性状发生联系起来的概率。我们建议使用这些获得的概率来告知现有生物物理模型的参数。将转录组信息合并到基于过程的模型中,可以更好地整合复杂系统中不同规模的经验数据和过程,同时增加在未知环境条件下进行生长预测的可能性。整个实体集合,例如基因转录本、代谢物和蛋白质。在这里,我们提出了一个新的框架,它利用贝叶斯网络提供将基因表达水平与性状发生联系起来的概率。我们建议使用这些获得的概率来告知现有生物物理模型的参数。将转录组信息合并到基于过程的模型中,可以更好地整合复杂系统中不同规模的经验数据和过程,同时增加在未知环境条件下进行生长预测的可能性。整个实体集合,例如基因转录本、代谢物和蛋白质。在这里,我们提出了一个新的框架,它利用贝叶斯网络提供将基因表达水平与性状发生联系起来的概率。我们建议使用这些获得的概率来告知现有生物物理模型的参数。将转录组信息合并到基于过程的模型中,可以更好地整合复杂系统中不同规模的经验数据和过程,同时增加在未知环境条件下进行生长预测的可能性。我们建议使用这些获得的概率来告知现有生物物理模型的参数。将转录组信息合并到基于过程的模型中,可以更好地整合复杂系统中不同规模的经验数据和过程,同时增加在未知环境条件下进行生长预测的可能性。我们建议使用这些获得的概率来告知现有生物物理模型的参数。将转录组信息合并到基于过程的模型中,可以更好地整合复杂系统中不同规模的经验数据和过程,同时增加在未知环境条件下进行生长预测的可能性。
更新日期:2020-08-01
中文翻译:

使用转录组数据通过贝叶斯网络为生物物理模型提供信息
摘要 基于过程的植物生产力模型提供了一种预测产量的方法,可以为农业和土地管理的最佳实践提供信息。然而,当前的生物物理模型无法在不断变化的环境条件下捕获基因型和表型变异。影响最终产量和生态系统生产力的生理性状是基因表达、蛋白质翻译和代谢物形成的结果,它们由特定的等位基因单独控制并响应时间和环境线索。生物物理相互作用在多个时间和空间尺度上同时发生,导致高度复杂性并限制了现有分析方法的预测能力。虽然统计方法可以部分量化基因型与环境的联系,生物物理过程模型错过了对基因型多样性的探索,它们目前无法通过组学数据实现,即基因转录、代谢物和蛋白质等实体的整个集合。在这里,我们提出了一个新的框架,它利用贝叶斯网络提供将基因表达水平与性状发生联系起来的概率。我们建议使用这些获得的概率来告知现有生物物理模型的参数。将转录组信息合并到基于过程的模型中,可以更好地整合复杂系统中不同规模的经验数据和过程,同时增加在未知环境条件下进行生长预测的可能性。整个实体集合,例如基因转录本、代谢物和蛋白质。在这里,我们提出了一个新的框架,它利用贝叶斯网络提供将基因表达水平与性状发生联系起来的概率。我们建议使用这些获得的概率来告知现有生物物理模型的参数。将转录组信息合并到基于过程的模型中,可以更好地整合复杂系统中不同规模的经验数据和过程,同时增加在未知环境条件下进行生长预测的可能性。整个实体集合,例如基因转录本、代谢物和蛋白质。在这里,我们提出了一个新的框架,它利用贝叶斯网络提供将基因表达水平与性状发生联系起来的概率。我们建议使用这些获得的概率来告知现有生物物理模型的参数。将转录组信息合并到基于过程的模型中,可以更好地整合复杂系统中不同规模的经验数据和过程,同时增加在未知环境条件下进行生长预测的可能性。我们建议使用这些获得的概率来告知现有生物物理模型的参数。将转录组信息合并到基于过程的模型中,可以更好地整合复杂系统中不同规模的经验数据和过程,同时增加在未知环境条件下进行生长预测的可能性。我们建议使用这些获得的概率来告知现有生物物理模型的参数。将转录组信息合并到基于过程的模型中,可以更好地整合复杂系统中不同规模的经验数据和过程,同时增加在未知环境条件下进行生长预测的可能性。











































 京公网安备 11010802027423号
京公网安备 11010802027423号