Scientific Reports ( IF 3.8 ) Pub Date : 2020-03-27 , DOI: 10.1038/s41598-020-62508-8 Aman Chandra Kaushik 1, 2 , Aamir Mehmood 2 , Xiaofeng Dai 1 , Dong-Qing Wei 2
|
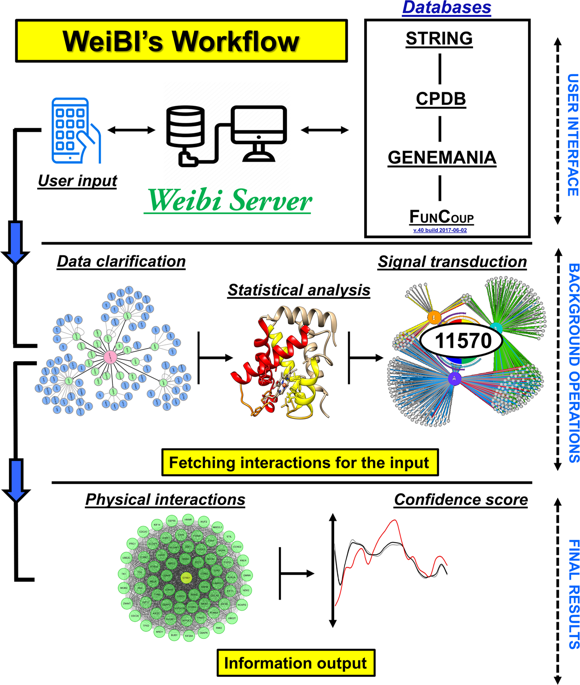
Many molecular system biology approaches recognize various interactions and functional associations of proteins that occur in cellular processing. Further understanding of the characterization technique reveals noteworthy information. These types of known and predicted interactions, gained through multiple resources, are thought to be important for experimental data to satisfy comprehensive and quality needs. The current work proposes the “WeiBI (WeiBiologicalInteractions)” database that clarifies direct and indirect partnerships associated with biological interactions. This database contains information concerning protein’s functional partnerships and interactions along with their integration into a statistical model that can be computationally predicted for humans. This novel approach in WeiBI version 1.0 collects information using an improved algorithm by transferring interactions between more than 115570 entries, allowing statistical analysis with the automated background for the given inputs for functional enrichment. This approach also allows the input of an entity’s list from a database along with the visualization of subsets as an interaction network and successful performance of the enrichment analysis for a gene set. This wisely improved algorithm is user-friendly, and its accessibility and higher accuracy make it the best database for exploring interactions among genomes’ network and reflects the importance of this study. The proposed server “WeiBI” is accessible at http://weislab.com/WeiDOCK/?page=PKPD.
中文翻译:

WeiBI(基于Web的平台):利用覆盖全基因组的实验OMICS数据,增强了覆盖范围和功能蛋白的集成交互网络。
许多分子系统生物学方法认识到细胞加工中发生的蛋白质的各种相互作用和功能关联。对表征技术的进一步了解揭示了值得注意的信息。通过多种资源获得的这些已知和预测的交互类型,被认为对于满足综合和质量需求的实验数据很重要。当前的工作提出了一个“ WeiBI(WeiBiologicalInteractions)”数据库,该数据库阐明了与生物相互作用有关的直接和间接的伙伴关系。该数据库包含有关蛋白质功能伙伴关系和相互作用的信息,以及将其整合到可以对人类进行计算预测的统计模型中的信息。WeiBI版本1中的这种新颖方法。0通过在超过115570个条目之间进行交互来使用改进的算法来收集信息,从而允许针对给定输入的功能丰富的自动背景进行统计分析。这种方法还允许从数据库中输入实体列表,以及将子集可视化为交互网络,并成功进行基因集的富集分析。这种经过明智改进的算法是用户友好的,其可访问性和更高的准确性使其成为探索基因组网络之间相互作用的最佳数据库,并反映了这项研究的重要性。建议的服务器“ WeiBI”可从http://weislab.com/WeiDOCK/?page=PKPD访问。允许使用给定功能输入的自动背景进行统计分析。这种方法还允许从数据库中输入实体列表,以及将子集可视化为交互网络,并成功进行基因集的富集分析。这种经过明智改进的算法是用户友好的,其可访问性和更高的准确性使其成为探索基因组网络之间相互作用的最佳数据库,并反映了这项研究的重要性。建议的服务器“ WeiBI”可从http://weislab.com/WeiDOCK/?page=PKPD访问。可以在给定功能输入的给定输入的情况下,利用自动背景进行统计分析。这种方法还允许从数据库中输入实体列表,以及将子集可视化为交互网络,并成功进行基因集的富集分析。这种经过明智改进的算法是用户友好的,它的可访问性和更高的准确性使其成为探索基因组网络之间相互作用的最佳数据库,并反映了这项研究的重要性。建议的服务器“ WeiBI”可从http://weislab.com/WeiDOCK/?page=PKPD访问。这种经过明智改进的算法是用户友好的,其可访问性和更高的准确性使其成为探索基因组网络之间相互作用的最佳数据库,并反映了这项研究的重要性。建议的服务器“ WeiBI”可从http://weislab.com/WeiDOCK/?page=PKPD访问。这种经过明智改进的算法是用户友好的,它的可访问性和更高的准确性使其成为探索基因组网络之间相互作用的最佳数据库,并反映了这项研究的重要性。建议的服务器“ WeiBI”可从http://weislab.com/WeiDOCK/?page=PKPD访问。











































 京公网安备 11010802027423号
京公网安备 11010802027423号