当前位置:
X-MOL 学术
›
Prog. Nucl. Magn. Reson. Spectrosc.
›
论文详情
Our official English website, www.x-mol.net, welcomes your
feedback! (Note: you will need to create a separate account there.)
A solid-state NMR tool box for the investigation of ATP-fueled protein engines
Progress in Nuclear Magnetic Resonance Spectroscopy ( IF 7.3 ) Pub Date : 2020-04-01 , DOI: 10.1016/j.pnmrs.2020.02.001 Thomas Wiegand 1
Progress in Nuclear Magnetic Resonance Spectroscopy ( IF 7.3 ) Pub Date : 2020-04-01 , DOI: 10.1016/j.pnmrs.2020.02.001 Thomas Wiegand 1
Affiliation
|
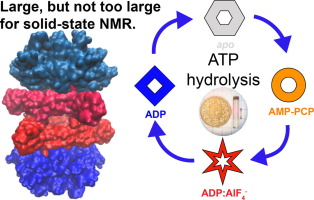
Motor proteins are involved in a variety of cellular processes. Their main purpose is to convert the chemical energy released during adenosine triphosphate (ATP) hydrolysis into mechanical work. In this review, solid-state Nuclear Magnetic Resonance (NMR) approaches are discussed allowing studies of structures, conformational events and dynamic features of motor proteins during a variety of enzymatic reactions. Solid-state NMR benefits from straightforward sample preparation based on sedimentation of the proteins directly into the Magic-Angle Spinning (MAS) rotor. Protein resonance assignment is the crucial and often time-limiting step in interpreting the wealth of information encoded in the NMR spectra. Herein, potentials, challenges and limitations in resonance assignment for large motor proteins are presented, focussing on both biochemical and spectroscopic approaches. This work highlights NMR tools available to study the action of the motor domain and its coupling to functional processes, as well as to identify protein-nucleotide interactions during events such as DNA replication. Arrested protein states of reaction coordinates such as ATP hydrolysis can be trapped for NMR studies by using stable, non-hydrolysable ATP analogues that mimic the physiological relevant states as accurately as possible. Recent advances in solid-state NMR techniques ranging from Dynamic Nuclear Polarization (DNP), 31P-based heteronuclear correlation experiments, 1H-detected spectra at fast MAS frequencies >100 kHz to paramagnetic NMR are summarized and their applications to the bacterial DnaB helicase from Helicobacter pylori are discussed.
中文翻译:

用于研究 ATP 燃料蛋白质发动机的固态 NMR 工具箱
运动蛋白参与多种细胞过程。它们的主要目的是将三磷酸腺苷 (ATP) 水解过程中释放的化学能转化为机械功。在这篇综述中,讨论了固态核磁共振 (NMR) 方法,允许研究各种酶促反应过程中运动蛋白的结构、构象事件和动态特征。固态 NMR 得益于基于蛋白质直接沉降到魔角旋转 (MAS) 转子中的简单样品制备。蛋白质共振分配是解释 NMR 光谱中编码的大量信息的关键且通常有时间限制的步骤。在此,提出了大运动蛋白共振分配的潜力、挑战和限制,专注于生化和光谱方法。这项工作重点介绍了 NMR 工具,可用于研究运动域的作用及其与功能过程的耦合,以及识别 DNA 复制等事件期间的蛋白质-核苷酸相互作用。通过使用尽可能准确地模拟生理相关状态的稳定、不可水解的 ATP 类似物,可以捕获反应坐标(如 ATP 水解)的受阻蛋白质状态,用于 NMR 研究。总结了固态 NMR 技术的最新进展,包括动态核极化 (DNP)、基于 31P 的异核相关实验、快速 MAS 频率 >100 kHz 的 1H 检测光谱到顺磁 NMR 以及它们在来自螺杆菌的细菌 DnaB 解旋酶中的应用幽门螺杆菌进行讨论。这项工作重点介绍了 NMR 工具,可用于研究运动域的作用及其与功能过程的耦合,以及识别 DNA 复制等事件期间的蛋白质-核苷酸相互作用。通过使用尽可能准确地模拟生理相关状态的稳定、不可水解的 ATP 类似物,可以捕获反应坐标(如 ATP 水解)的受阻蛋白质状态,用于 NMR 研究。总结了固态 NMR 技术的最新进展,包括动态核极化 (DNP)、基于 31P 的异核相关实验、快速 MAS 频率 >100 kHz 的 1H 检测光谱到顺磁 NMR 以及它们在来自螺杆菌的细菌 DnaB 解旋酶中的应用幽门螺杆菌进行讨论。这项工作重点介绍了 NMR 工具,可用于研究运动域的作用及其与功能过程的耦合,以及识别 DNA 复制等事件期间的蛋白质-核苷酸相互作用。通过使用尽可能准确地模拟生理相关状态的稳定、不可水解的 ATP 类似物,可以捕获反应坐标(例如 ATP 水解)的受阻蛋白质状态,用于 NMR 研究。总结了固态 NMR 技术的最新进展,包括动态核极化 (DNP)、基于 31P 的异核相关实验、快速 MAS 频率 >100 kHz 的 1H 检测光谱到顺磁 NMR 以及它们在来自螺杆菌的细菌 DnaB 解旋酶中的应用幽门螺杆菌进行讨论。
更新日期:2020-04-01
中文翻译:

用于研究 ATP 燃料蛋白质发动机的固态 NMR 工具箱
运动蛋白参与多种细胞过程。它们的主要目的是将三磷酸腺苷 (ATP) 水解过程中释放的化学能转化为机械功。在这篇综述中,讨论了固态核磁共振 (NMR) 方法,允许研究各种酶促反应过程中运动蛋白的结构、构象事件和动态特征。固态 NMR 得益于基于蛋白质直接沉降到魔角旋转 (MAS) 转子中的简单样品制备。蛋白质共振分配是解释 NMR 光谱中编码的大量信息的关键且通常有时间限制的步骤。在此,提出了大运动蛋白共振分配的潜力、挑战和限制,专注于生化和光谱方法。这项工作重点介绍了 NMR 工具,可用于研究运动域的作用及其与功能过程的耦合,以及识别 DNA 复制等事件期间的蛋白质-核苷酸相互作用。通过使用尽可能准确地模拟生理相关状态的稳定、不可水解的 ATP 类似物,可以捕获反应坐标(如 ATP 水解)的受阻蛋白质状态,用于 NMR 研究。总结了固态 NMR 技术的最新进展,包括动态核极化 (DNP)、基于 31P 的异核相关实验、快速 MAS 频率 >100 kHz 的 1H 检测光谱到顺磁 NMR 以及它们在来自螺杆菌的细菌 DnaB 解旋酶中的应用幽门螺杆菌进行讨论。这项工作重点介绍了 NMR 工具,可用于研究运动域的作用及其与功能过程的耦合,以及识别 DNA 复制等事件期间的蛋白质-核苷酸相互作用。通过使用尽可能准确地模拟生理相关状态的稳定、不可水解的 ATP 类似物,可以捕获反应坐标(如 ATP 水解)的受阻蛋白质状态,用于 NMR 研究。总结了固态 NMR 技术的最新进展,包括动态核极化 (DNP)、基于 31P 的异核相关实验、快速 MAS 频率 >100 kHz 的 1H 检测光谱到顺磁 NMR 以及它们在来自螺杆菌的细菌 DnaB 解旋酶中的应用幽门螺杆菌进行讨论。这项工作重点介绍了 NMR 工具,可用于研究运动域的作用及其与功能过程的耦合,以及识别 DNA 复制等事件期间的蛋白质-核苷酸相互作用。通过使用尽可能准确地模拟生理相关状态的稳定、不可水解的 ATP 类似物,可以捕获反应坐标(例如 ATP 水解)的受阻蛋白质状态,用于 NMR 研究。总结了固态 NMR 技术的最新进展,包括动态核极化 (DNP)、基于 31P 的异核相关实验、快速 MAS 频率 >100 kHz 的 1H 检测光谱到顺磁 NMR 以及它们在来自螺杆菌的细菌 DnaB 解旋酶中的应用幽门螺杆菌进行讨论。











































 京公网安备 11010802027423号
京公网安备 11010802027423号