当前位置:
X-MOL 学术
›
J. Comput. Chem.
›
论文详情
Our official English website, www.x-mol.net, welcomes your
feedback! (Note: you will need to create a separate account there.)
Distributed and multicore QuBiLS‐MIDAS software v2.0: Computing chiral, fuzzy, weighted and truncated geometrical molecular descriptors based on tensor algebra
Journal of Computational Chemistry ( IF 3.4 ) Pub Date : 2020-05-05 , DOI: 10.1002/jcc.26167 César R García-Jacas 1 , Yovani Marrero-Ponce 2, 3, 4, 5 , Ricardo Vivas-Reyes 6, 7 , José Suárez-Lezcano 8 , Felix Martinez-Rios 9 , Julio E Terán 10 , Longendri Aguilera-Mendoza 11
Journal of Computational Chemistry ( IF 3.4 ) Pub Date : 2020-05-05 , DOI: 10.1002/jcc.26167 César R García-Jacas 1 , Yovani Marrero-Ponce 2, 3, 4, 5 , Ricardo Vivas-Reyes 6, 7 , José Suárez-Lezcano 8 , Felix Martinez-Rios 9 , Julio E Terán 10 , Longendri Aguilera-Mendoza 11
Affiliation
|
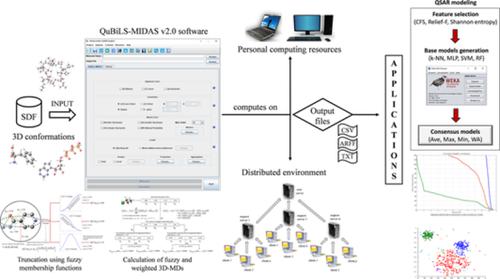
Advances to the distributed, multi‐core and fully cross‐platform QuBiLS‐MIDAS software v2.0 (http://tomocomd.com/qubils-midas) are reported in this article since the v1.0 release. The QuBiLS‐MIDAS software is the only one that computes atom‐pair and alignment‐free geometrical MDs (3D‐MDs) from several distance metrics other than the Euclidean distance, as well as alignment‐free 3D‐MDs that codify structural information regarding the relations among three and four atoms of a molecule. The most recent features added to the QuBiLS‐MIDAS software v2.0 are related (a) to the calculation of atomic weightings from indices based on the vertex‐degree invariant (e.g., Alikhanidi index); (b) to consider central chirality during the molecular encoding; (c) to use measures based on clustering methods and statistical functions to codify structural information among more than two atoms; (d) to the use of a novel method based on fuzzy membership functions to spherically truncate inter‐atomic relations; and (e) to the use of weighted and fuzzy aggregation operators to compute global 3D‐MDs according to the importance and/or interrelation of the atoms of a molecule during the molecular encoding. Moreover, a novel module to compute QuBiLS‐MIDAS 3D‐MDs from their headings was also developed. This module can be used either by the graphical user interface or by means of the software library. By using the library, both the predictive models built with the QuBiLS‐MIDAS 3D‐MDs and the QuBiLS‐MIDAS 3D‐MDs calculation can be embedded in other tools. A set of predefined QuBiLS‐MIDAS 3D‐MDs with high information content and low redundancy on a set comprised of 20,469 compounds is also provided to be employed in further cheminformatics tasks. This set of predefined 3D‐MDs evidenced better performance than all the universe of Dragon (v5.5) and PaDEL 0D‐to‐3D MDs in variability studies, whereas a linear independence study proved that these QuBiLS‐MIDAS 3D‐MDs codify chemical information orthogonal to the Dragon 0D‐to‐3D MDs. This set of predefined 3D‐MDs would be periodically updated as long as new results be achieved. In general, this report highlights our continued efforts to provide a better tool for a most suitable characterization of compounds, and in this way, to contribute to obtaining better outcomes in future applications.
中文翻译:

分布式和多核 QuBiLS-MIDAS 软件 v2.0:基于张量代数计算手性、模糊、加权和截断几何分子描述符
自 v1.0 发布以来,本文报告了分布式、多核和完全跨平台的 QuBiLS-MIDAS 软件 v2.0 (http://tomocomd.com/qubils-midas) 的进展。QuBiLS-MIDAS 软件是唯一一个从欧几里德距离以外的几个距离度量计算原子对和无对齐几何 MD (3D-MD) 的软件,以及编码有关结构信息的无对齐 3D-MD一个分子的三个和四个原子之间的关系。QuBiLS-MIDAS 软件 v2.0 中添加的最新功能与 (a) 计算基于顶点度不变量的索引的原子权重(例如,Alikhanidi 索引)有关;(b) 在分子编码过程中考虑中心手性;(c) 使用基于聚类方法和统计函数的措施来编码两个以上原子之间的结构信息;(d) 使用基于模糊隶属函数的新方法来球形截断原子间关系;(e) 根据分子编码过程中分子原子的重要性和/或相互关系,使用加权和模糊聚合算子来计算全局 3D-MD。此外,还开发了一个新的模块,用于根据其标题计算 QuBiLS-MIDAS 3D-MD。该模块既可以通过图形用户界面使用,也可以通过软件库使用。通过使用该库,使用 QuBiLS-MIDAS 3D-MDs 构建的预测模型和 QuBiLS-MIDAS 3D-MDs 计算都可以嵌入到其他工具中。还提供了一组预定义的 QuBiLS-MIDAS 3D-MD,在由 20,469 种化合物组成的集合上具有高信息含量和低冗余,以用于进一步的化学信息学任务。这组预定义的 3D-MD 在变异性研究中表现出比所有 Dragon (v5.5) 和 PaDEL 0D-to-3D MD 更好的性能,而线性独立性研究证明这些 QuBiLS-MIDAS 3D-MD 编码化学信息与 Dragon 0D 到 3D MD 正交。只要取得新的结果,这组预定义的 3D-MD 就会定期更新。总的来说,本报告强调了我们不断努力为化合物的最合适表征提供更好的工具,并以这种方式有助于在未来的应用中获得更好的结果。还提供了 469 种化合物以用于进一步的化学信息学任务。这组预定义的 3D-MD 在变异性研究中表现出比所有 Dragon (v5.5) 和 PaDEL 0D-to-3D MD 更好的性能,而线性独立性研究证明这些 QuBiLS-MIDAS 3D-MD 编码化学信息与 Dragon 0D 到 3D MD 正交。只要取得新的结果,这组预定义的 3D-MD 就会定期更新。总的来说,本报告强调了我们不断努力为化合物的最合适表征提供更好的工具,并以这种方式有助于在未来的应用中获得更好的结果。还提供了 469 种化合物以用于进一步的化学信息学任务。这组预定义的 3D-MD 在变异性研究中表现出比所有 Dragon (v5.5) 和 PaDEL 0D-to-3D MD 更好的性能,而线性独立性研究证明这些 QuBiLS-MIDAS 3D-MD 编码化学信息与 Dragon 0D 到 3D MD 正交。只要取得新的结果,这组预定义的 3D-MD 就会定期更新。总的来说,本报告强调了我们不断努力为化合物的最合适表征提供更好的工具,并以这种方式有助于在未来的应用中获得更好的结果。5) 和 PaDEL 0D-to-3D MDs 在变异性研究中,而线性独立研究证明这些 QuBiLS-MIDAS 3D-MDs 编码与 Dragon 0D-to-3D MDs 正交的化学信息。只要取得新的结果,这组预定义的 3D-MD 就会定期更新。总的来说,本报告强调了我们不断努力为化合物的最合适表征提供更好的工具,并以这种方式有助于在未来的应用中获得更好的结果。5) 和 PaDEL 0D-to-3D MDs 在变异性研究中,而线性独立研究证明这些 QuBiLS-MIDAS 3D-MDs 编码与 Dragon 0D-to-3D MDs 正交的化学信息。只要取得新的结果,这组预定义的 3D-MD 就会定期更新。总的来说,本报告强调了我们不断努力为化合物的最合适表征提供更好的工具,并以这种方式有助于在未来的应用中获得更好的结果。
更新日期:2020-05-05
中文翻译:

分布式和多核 QuBiLS-MIDAS 软件 v2.0:基于张量代数计算手性、模糊、加权和截断几何分子描述符
自 v1.0 发布以来,本文报告了分布式、多核和完全跨平台的 QuBiLS-MIDAS 软件 v2.0 (http://tomocomd.com/qubils-midas) 的进展。QuBiLS-MIDAS 软件是唯一一个从欧几里德距离以外的几个距离度量计算原子对和无对齐几何 MD (3D-MD) 的软件,以及编码有关结构信息的无对齐 3D-MD一个分子的三个和四个原子之间的关系。QuBiLS-MIDAS 软件 v2.0 中添加的最新功能与 (a) 计算基于顶点度不变量的索引的原子权重(例如,Alikhanidi 索引)有关;(b) 在分子编码过程中考虑中心手性;(c) 使用基于聚类方法和统计函数的措施来编码两个以上原子之间的结构信息;(d) 使用基于模糊隶属函数的新方法来球形截断原子间关系;(e) 根据分子编码过程中分子原子的重要性和/或相互关系,使用加权和模糊聚合算子来计算全局 3D-MD。此外,还开发了一个新的模块,用于根据其标题计算 QuBiLS-MIDAS 3D-MD。该模块既可以通过图形用户界面使用,也可以通过软件库使用。通过使用该库,使用 QuBiLS-MIDAS 3D-MDs 构建的预测模型和 QuBiLS-MIDAS 3D-MDs 计算都可以嵌入到其他工具中。还提供了一组预定义的 QuBiLS-MIDAS 3D-MD,在由 20,469 种化合物组成的集合上具有高信息含量和低冗余,以用于进一步的化学信息学任务。这组预定义的 3D-MD 在变异性研究中表现出比所有 Dragon (v5.5) 和 PaDEL 0D-to-3D MD 更好的性能,而线性独立性研究证明这些 QuBiLS-MIDAS 3D-MD 编码化学信息与 Dragon 0D 到 3D MD 正交。只要取得新的结果,这组预定义的 3D-MD 就会定期更新。总的来说,本报告强调了我们不断努力为化合物的最合适表征提供更好的工具,并以这种方式有助于在未来的应用中获得更好的结果。还提供了 469 种化合物以用于进一步的化学信息学任务。这组预定义的 3D-MD 在变异性研究中表现出比所有 Dragon (v5.5) 和 PaDEL 0D-to-3D MD 更好的性能,而线性独立性研究证明这些 QuBiLS-MIDAS 3D-MD 编码化学信息与 Dragon 0D 到 3D MD 正交。只要取得新的结果,这组预定义的 3D-MD 就会定期更新。总的来说,本报告强调了我们不断努力为化合物的最合适表征提供更好的工具,并以这种方式有助于在未来的应用中获得更好的结果。还提供了 469 种化合物以用于进一步的化学信息学任务。这组预定义的 3D-MD 在变异性研究中表现出比所有 Dragon (v5.5) 和 PaDEL 0D-to-3D MD 更好的性能,而线性独立性研究证明这些 QuBiLS-MIDAS 3D-MD 编码化学信息与 Dragon 0D 到 3D MD 正交。只要取得新的结果,这组预定义的 3D-MD 就会定期更新。总的来说,本报告强调了我们不断努力为化合物的最合适表征提供更好的工具,并以这种方式有助于在未来的应用中获得更好的结果。5) 和 PaDEL 0D-to-3D MDs 在变异性研究中,而线性独立研究证明这些 QuBiLS-MIDAS 3D-MDs 编码与 Dragon 0D-to-3D MDs 正交的化学信息。只要取得新的结果,这组预定义的 3D-MD 就会定期更新。总的来说,本报告强调了我们不断努力为化合物的最合适表征提供更好的工具,并以这种方式有助于在未来的应用中获得更好的结果。5) 和 PaDEL 0D-to-3D MDs 在变异性研究中,而线性独立研究证明这些 QuBiLS-MIDAS 3D-MDs 编码与 Dragon 0D-to-3D MDs 正交的化学信息。只要取得新的结果,这组预定义的 3D-MD 就会定期更新。总的来说,本报告强调了我们不断努力为化合物的最合适表征提供更好的工具,并以这种方式有助于在未来的应用中获得更好的结果。











































 京公网安备 11010802027423号
京公网安备 11010802027423号