当前位置:
X-MOL 学术
›
J. Mol. Biol.
›
论文详情
Our official English website, www.x-mol.net, welcomes your
feedback! (Note: you will need to create a separate account there.)
Intracellular Positioning Systems Limit the Entropic Eviction of Secondary Replicons Toward the Nucleoid Edges in Bacterial Cells.
Journal of Molecular Biology ( IF 4.7 ) Pub Date : 2020-01-10 , DOI: 10.1016/j.jmb.2019.11.027 Charlène Planchenault 1 , Marine C Pons 2 , Caroline Schiavon 2 , Patricia Siguier 2 , Jérôme Rech 2 , Catherine Guynet 2 , Julie Dauverd-Girault 1 , Jean Cury 3 , Eduardo P C Rocha 3 , Ivan Junier 4 , François Cornet 2 , Olivier Espéli 1
Journal of Molecular Biology ( IF 4.7 ) Pub Date : 2020-01-10 , DOI: 10.1016/j.jmb.2019.11.027 Charlène Planchenault 1 , Marine C Pons 2 , Caroline Schiavon 2 , Patricia Siguier 2 , Jérôme Rech 2 , Catherine Guynet 2 , Julie Dauverd-Girault 1 , Jean Cury 3 , Eduardo P C Rocha 3 , Ivan Junier 4 , François Cornet 2 , Olivier Espéli 1
Affiliation
|
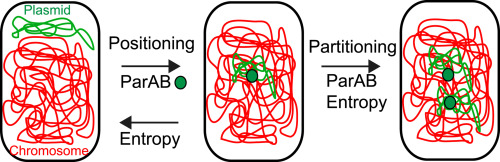
Bacterial genomes, organized intracellularly as nucleoids, are composed of the main chromosome coexisting with different types of secondary replicons. Secondary replicons are major drivers of bacterial adaptation by gene exchange. They are highly diverse in type and size, ranging from less than 2 to more than 1000 kb, and must integrate with bacterial physiology, including to the nucleoid dynamics, to limit detrimental costs leading to their counter-selection. We show that large DNA circles, whether from a natural plasmid or excised from the chromosome tend to localize in a dynamic manner in a zone separating the nucleoid from the cytoplasm at the edge of the nucleoid. This localization is in good agreement with silico simulations of DNA circles in the nucleoid volume. Subcellular positioning systems counteract this tendency, allowing replicons to enter the nucleoid space. In enterobacteria, these systems are found in replicons above 25 kb, defining the limit with small randomly segregated plasmids. Larger replicons carry at least one of the three described family of systems, ParAB, ParRM, and StbA. Replicons above 180 kb all carry a ParAB system, suggesting this system is specifically required in the cases of large replicons. Simulations demonstrated that replicon size profoundly affects localization, compaction, and dynamics of DNA circles in the nucleoid volume. The present work suggests that presence of partition systems on the larger plasmids or chromids is not only due to selection for accurate segregation but also to counteract their unmixing with the chromosome and consequent exclusion from the nucleoid.
中文翻译:

细胞内定位系统限制了二级复制子向细菌细胞核边缘的熵驱逐。
细菌基因组在细胞内组织为核苷,由与不同类型的次级复制子共存的主染色体组成。二级复制子是通过基因交换进行细菌适应的主要驱动力。它们的类型和大小差异很大,范围从小于2 kb到大于1000 kb,并且必须与细菌生理学结合,包括与核苷动力学结合,以限制导致反选择的有害成本。我们显示,无论是从天然质粒还是从染色体上切除的大DNA环,都倾向于以动态的方式位于将核苷与胞质分离的区域中。这种定位与核苷体积中DNA环的计算机模拟非常吻合。亚细胞定位系统可以抵消这种趋势,允许复制子进入类核苷酸空间。在肠杆菌中,这些系统存在于25 kb以上的复制子中,从而限定了随机分离的小质粒的限制。较大的复制子携带上述三个系统家族中的至少一个,即ParAB,ParRM和StbA。180 kb以上的复制子均带有ParAB系统,这表明在大型复制子的情况下特别需要此系统。模拟表明,复制子的大小会深刻影响核苷体积中DNA环的定位,紧密化和动力学。目前的工作表明,较大质粒或染色体上分配系统的存在,不仅是由于选择了正确的分离方法,而且还抵消了它们与染色体的解混以及随后从核仁中的排斥。这些系统存在于25 kb以上的复制子中,从而限定了随机分离的小质粒的限制。较大的复制子携带上述三个系统家族中的至少一个,即ParAB,ParRM和StbA。180 kb以上的复制子均带有ParAB系统,这表明在大型复制子的情况下特别需要此系统。模拟表明,复制子的大小会深刻影响核苷体积中DNA环的定位,紧密化和动力学。目前的工作表明,较大质粒或染色体上分配系统的存在,不仅是由于选择了正确的分离方法,而且还抵消了它们与染色体的解混以及随后从核仁中的排斥。在超过25 kb的复制子中发现了这些系统,从而确定了随机分离的小质粒的限制。较大的复制子携带上述三个系统家族中的至少一个,即ParAB,ParRM和StbA。180 kb以上的复制子均带有ParAB系统,这表明在大型复制子的情况下特别需要此系统。模拟表明,复制子的大小会深刻影响核苷体积中DNA环的定位,紧密化和动力学。目前的工作表明,较大质粒或染色体上分配系统的存在,不仅是由于选择了正确的分离方法,而且还抵消了它们与染色体的解混以及随后从核仁中的排斥。较大的复制子携带上述三个系统家族中的至少一个,即ParAB,ParRM和StbA。180 kb以上的复制子均带有ParAB系统,这表明在大型复制子的情况下特别需要此系统。模拟表明,复制子的大小会深刻影响核苷体积中DNA环的定位,紧密化和动力学。目前的工作表明,较大质粒或染色体上分配系统的存在,不仅是由于选择了正确的分离方法,而且还抵消了它们与染色体的解混以及随后从核仁中的排斥。较大的复制子携带上述三个系统家族中的至少一个,即ParAB,ParRM和StbA。180 kb以上的复制子均带有ParAB系统,这表明在大型复制子的情况下特别需要此系统。模拟表明,复制子的大小会深刻影响核苷体积中DNA环的定位,紧密化和动力学。目前的工作表明,较大质粒或染色体上分配系统的存在,不仅是由于选择了正确的分离方法,而且还抵消了它们与染色体的解混以及随后从核仁中的排斥。模拟表明,复制子的大小会深刻影响核苷体积中DNA环的定位,紧密化和动力学。目前的工作表明,较大质粒或染色体上分配系统的存在,不仅是由于选择了正确的分离方法,而且还抵消了它们与染色体的解混以及随后从核仁中的排斥。模拟表明,复制子的大小会深刻影响核苷体积中DNA环的定位,紧密化和动力学。目前的工作表明,较大质粒或染色体上分配系统的存在,不仅是由于选择了正确的分离方法,而且还抵消了它们与染色体的解混以及随后从核仁中的排斥。
更新日期:2020-01-13
中文翻译:

细胞内定位系统限制了二级复制子向细菌细胞核边缘的熵驱逐。
细菌基因组在细胞内组织为核苷,由与不同类型的次级复制子共存的主染色体组成。二级复制子是通过基因交换进行细菌适应的主要驱动力。它们的类型和大小差异很大,范围从小于2 kb到大于1000 kb,并且必须与细菌生理学结合,包括与核苷动力学结合,以限制导致反选择的有害成本。我们显示,无论是从天然质粒还是从染色体上切除的大DNA环,都倾向于以动态的方式位于将核苷与胞质分离的区域中。这种定位与核苷体积中DNA环的计算机模拟非常吻合。亚细胞定位系统可以抵消这种趋势,允许复制子进入类核苷酸空间。在肠杆菌中,这些系统存在于25 kb以上的复制子中,从而限定了随机分离的小质粒的限制。较大的复制子携带上述三个系统家族中的至少一个,即ParAB,ParRM和StbA。180 kb以上的复制子均带有ParAB系统,这表明在大型复制子的情况下特别需要此系统。模拟表明,复制子的大小会深刻影响核苷体积中DNA环的定位,紧密化和动力学。目前的工作表明,较大质粒或染色体上分配系统的存在,不仅是由于选择了正确的分离方法,而且还抵消了它们与染色体的解混以及随后从核仁中的排斥。这些系统存在于25 kb以上的复制子中,从而限定了随机分离的小质粒的限制。较大的复制子携带上述三个系统家族中的至少一个,即ParAB,ParRM和StbA。180 kb以上的复制子均带有ParAB系统,这表明在大型复制子的情况下特别需要此系统。模拟表明,复制子的大小会深刻影响核苷体积中DNA环的定位,紧密化和动力学。目前的工作表明,较大质粒或染色体上分配系统的存在,不仅是由于选择了正确的分离方法,而且还抵消了它们与染色体的解混以及随后从核仁中的排斥。在超过25 kb的复制子中发现了这些系统,从而确定了随机分离的小质粒的限制。较大的复制子携带上述三个系统家族中的至少一个,即ParAB,ParRM和StbA。180 kb以上的复制子均带有ParAB系统,这表明在大型复制子的情况下特别需要此系统。模拟表明,复制子的大小会深刻影响核苷体积中DNA环的定位,紧密化和动力学。目前的工作表明,较大质粒或染色体上分配系统的存在,不仅是由于选择了正确的分离方法,而且还抵消了它们与染色体的解混以及随后从核仁中的排斥。较大的复制子携带上述三个系统家族中的至少一个,即ParAB,ParRM和StbA。180 kb以上的复制子均带有ParAB系统,这表明在大型复制子的情况下特别需要此系统。模拟表明,复制子的大小会深刻影响核苷体积中DNA环的定位,紧密化和动力学。目前的工作表明,较大质粒或染色体上分配系统的存在,不仅是由于选择了正确的分离方法,而且还抵消了它们与染色体的解混以及随后从核仁中的排斥。较大的复制子携带上述三个系统家族中的至少一个,即ParAB,ParRM和StbA。180 kb以上的复制子均带有ParAB系统,这表明在大型复制子的情况下特别需要此系统。模拟表明,复制子的大小会深刻影响核苷体积中DNA环的定位,紧密化和动力学。目前的工作表明,较大质粒或染色体上分配系统的存在,不仅是由于选择了正确的分离方法,而且还抵消了它们与染色体的解混以及随后从核仁中的排斥。模拟表明,复制子的大小会深刻影响核苷体积中DNA环的定位,紧密化和动力学。目前的工作表明,较大质粒或染色体上分配系统的存在,不仅是由于选择了正确的分离方法,而且还抵消了它们与染色体的解混以及随后从核仁中的排斥。模拟表明,复制子的大小会深刻影响核苷体积中DNA环的定位,紧密化和动力学。目前的工作表明,较大质粒或染色体上分配系统的存在,不仅是由于选择了正确的分离方法,而且还抵消了它们与染色体的解混以及随后从核仁中的排斥。











































 京公网安备 11010802027423号
京公网安备 11010802027423号