用Cellar交互式分析单细胞数据
细胞分类是各种高通量单细胞数据面临的主要挑战。很多情况下,细胞分类需要借助外部数据和互补数据,并且还需要专业人员手工标注。这种方式不但耗时耗力,还会让结果具有非常强的主观性。而一些使用机器学习算法进行细胞分类的工具往往存在以下问题:(1)只适用于一部分的数据类型;(2)需要编程,不便于一些生物及医学从业人员的使用;(3)分析流程全自动,无法结合用户自己的知识。为解决这些问题,美国卡内基梅隆大学的Ziv Bar-Joseph教授团队(Euxhen Hasanaj, Arjun Sarathi)和加拿大麦吉尔大学的丁俊助理教授团队(王靖韬)开发了一款高交互性、便于使用的图形界面单细胞数据分析工具Cellar,并将其发表在期刊Nature Communications 上。
Cellar包含了主流用于单细胞数据分析的机器学习算法,并且支持几乎所有类型的单细胞数据。所有操作均在图形界面上完成。图1展示了Cellar的界面总览。界面主要分为:
侧边栏(图1蓝色数字)
1. 降维:将数据维度从基因数量降到40左右(可选)用于后续分析。并进一步降到2维用于可视化。
2. 聚类:提供大部分主流细胞聚类方法,参数可选。也提供半监督学习方法融合用户知识改进聚类,还提供从其他数据集转移标签的功能,以及提示可能错误分类细胞的“不确定性聚类”。
3. 标记:细胞类型标注
4. 工具:手工选定一部分的细胞,以便后续分析(标注、差异基因分析等)
5. 会话:导入、导出会话。便于分享分析结果。
分析栏(图1黄色数字)
1. 差异基因分析
2. 特征可视化(如基因表达热力图、小提琴图等)
3. 富集分析
其他功能(图1红色数字)
1. 导航栏:载入数据集、双数据集对比分析模式、数据预处理
2. 数据形状(细胞数量、基因数量、运行时间估计)
3. 图:数据集的散点图,一个点为一个细胞,颜色可选为聚类标签或者基因表达强度
4. 脚注(文档、Github、教程等)
一套典型的分析流程为:用导航栏载入数据集——(可选:用预处理筛除低质量细胞和基因)——用侧边栏进行降维及可视化、聚类——(可选:用工具栏选定一些聚类错误的细胞然后用半监督方法重新聚类)——用分析栏进行差异基因分析、各种特征的可视化、富集分析、以便确定各细胞种类——用侧边栏对细胞进行标注。

图1. Cellar总览。各个板块详情见上文。
另外,Cellar还支持对特定数据类型的附加功能,比如在分析栏的后续板块——空间板块中,支持对有空间信息的数据集(如CODEX数据集,10X Genomics的空间转录组数据集)的空间可视化。如下图展示了CODEX数据中结合了空间位置和聚类标签的可视化(1)以及聚类空间相似度热力图(2)和蛋白质表达空间相似度热力图(3)。
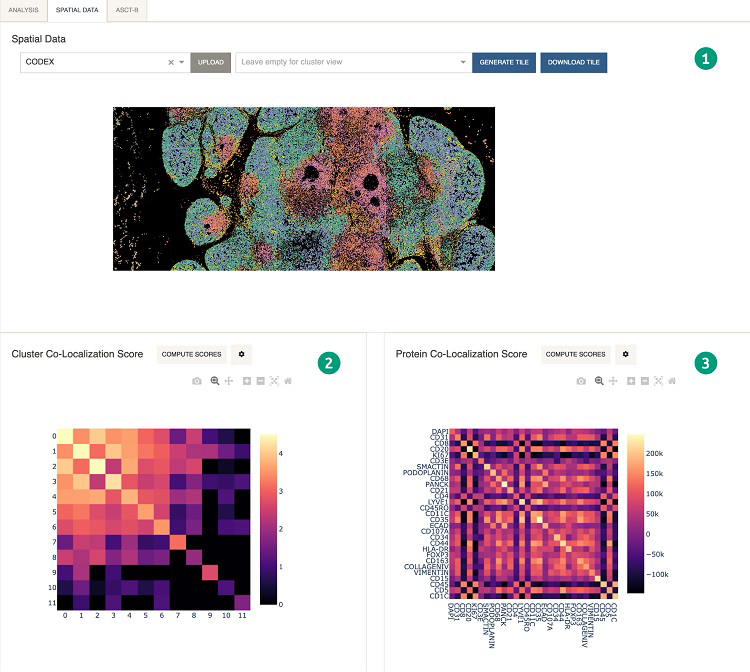
图2. 空间数据分析板块:(1)空间细胞可视化,标签和聚类结果一致;(2)聚类空间相似度热力图;(3)蛋白质表达空间相似度热力图
此外,Cellar还支持多模态数据的共同分析。下图展示了用导航栏进入双数据集模式后同时分析SNARE-seq中对相同细胞的ATAC-seq(左)和RNA-seq(右)。作者先用ATAC-seq数据进行聚类,然后将聚类标签转移到右侧的RNA-seq上。
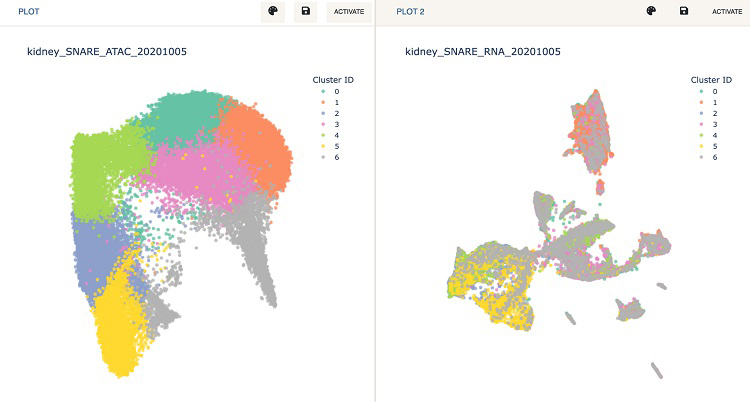
图3. 多组学数据协同分析,左侧为ATAC-seq数据,右侧为RNA-seq数据,右侧标签与左侧一致
Cellar的优势
首先,Cellar包含了主流单细胞分析的各个步骤的方法,如降维(PCA, Truncated SVD, TSNE, UMAP…)、聚类(Leiden,K-means,Spectral Clustering,...)、细胞标注、差异基因分析、富集分析,且包含了多个用于细胞标注以及富集分析的数据库。
另外,Cellar还采用了半自动的模式,让用户和机器学习算法协同作业进行分析。每个分析步骤都提供了大量可以调整的参数,且用户可以选定对聚类不满意的细胞用半监督学习方法重新聚类。
最后,Cellar提供对所有单细胞数据类型的支持(RNA-seq, ATAC-seq, CODEX, 10X Spatial Transcriptomics, ...)且对特定数据集提供特定功能,支持多模态协同分析。
原文(扫描或长按二维码,识别后直达原文页面,或点此查看原文):

Interactive single-cell data analysis using Cellar
Euxhen Hasanaj, Jingtao Wang, Arjun Sarathi, Jun Ding & Ziv Bar-Joseph
Nat. Commun., 2022, 13, 1998, DOI: 10.1038/s41467-022-29744-0
Cellar工具信息:
Cellar工具链接:
https://cellar.cmu.hubmapconsortium.org/app/cellar
Cellar是一款开源Github仓库:
https://github.com/euxhenh/cellar/
详细文档:
https://euxhenh.github.io/cellar/
主要分析流程的视频教程:
https://www.youtube.com/playlist?list=PL5sLSLkTYpWgfBQ0M8ObfBIqDMAzx0-D2
导师简介

丁俊(个人主页:https://meakinsmcgill.com/ding/ ),麦吉尔大学医学院、计算机学院助理教授。获美国中佛罗里达大学计算机博士学位后,与在美国卡内基美隆大学计算生物学系 Ziv Bar-Joseph (2012 Overton Prize 获奖者)教授实验室进行了4年博士后研究工作。2021年作为tenure-track助理教授加入麦吉尔大学Meakins-Christie Laboratories, Department of Medicine, Associate member of School of Computer Science, Associate member of Department of biomedical engineering 。主要研究方向为用概率图模型及深度无监督学习模型分析单细胞多组学数据,以深入了解生物系统中的细胞动力学,从而寻找生物标记物和疾病靶点并基此推动新型疾病诊断和治疗方案的发展。丁俊教授在Nature Communications, Nature Reviews Genetics, Genome Biology, Cell Stem Cell, Nucleic Acids Research, Cell Metabolisms 等期刊上发表论文30余篇,并获得FRQS Junior 1 fellow, NSERC Discovery, CIHR Project grant等科研资助及奖项。
如果篇首注明了授权来源,任何转载需获得来源方的许可!如果篇首未特别注明出处,本文版权属于 X-MOL ( x-mol.com ), 未经许可,谢绝转载!












































 京公网安备 11010802027423号
京公网安备 11010802027423号