EnzyHTP:“通向自动化酶设计”的高通量计算酶模拟平台
刚接触有机化学的时候,你有没有过这样一种体验:发现反应路线中存在一个看似极不合理的反应,高选择性,反应性,但条件却很温和?一查反应条件,发现原来是酶催化反应!即便我们不知道机理是什么,单纯听到酶起了作用,都会让我们产生释然的感觉。因为在我们的印象中,神秘的酶总能转化一切不可思议的分子过程。
但现实是残酷的。经过亿万年的进化,酶可以催化自己“认准”的一个天然反应。但对于大部分非天然反应,那些化学家感兴趣的、制药过程中用到的、环境污染物降解中需要的——即使反应物的结构与天然过程很接近——在天然的酶中都大多只呈现出很低的催化活性。为了能让酶为人所用,我们需要对天然的酶进行改造、对序列进行突变,从而让酶的突变体能够高效完成对非天然反应的催化。这个寻找超级酶突变体的过程充满了未知。虽然实验上有定向进化这种工程手段作为辅助,但这种手段涉及到的筛选周期和起始点都极大依赖于具体酶的特性。新的计算算法与模型,是让我们能够预测和探索未知突变空间的重要工具。
近日,J. Chem. Inf. Model. 杂志封面推荐了一篇来自范德堡大学(Vanderbilt University)的Qianzhen Shao等人的文章(Zhongyue Yang课题组),提供了一种帮助化学家自动探索酶突变体的计算手段。文中报道了该团队开发的一款自动化酶模拟的软件,EnzyHTP。该软件能够自动化地构建和修复结构(如给晶体结构加氢、决定质子态),生成突变,进行分子力学(MD)模拟,对模拟的结果进行量子力学(QM)、多尺度QM/MM计算分析,从而实现酶模拟的完整工作流程的自动化。这使得基于计算高效、高通量评估酶序列对于某种底物的催化能力变得可能,从而直接给出这些让反应性复苏的突变。

在此之前,人们针对改造酶来催化非天然反应这个问题也做过很多的努力和尝试。实验上,基于高通量实验筛选的定向进化给出了解决这个问题的一个方向,这种方法随机的产生大量的突变并在每一轮进行筛选,选出催化活性提升的突变进行下一轮的筛选,从而不断优化酶的序列得到想要的催化活性。但是由于在这个过程中,序列-结构-催化性能的关系是未知的,所需要进行的筛选数量高度依赖于初始的序列和当前的目标底物。这种黑箱式的现状使得需要花费的时间和资源十分不确定,并且对于原本没有酶催化活性的非天然反应,很有可能无法找到最初具有反应性酶突变/序列作为定向进化的起点。
理论计算上,人们使用了基于分子力学(MM)、量子力学(QM)以及多尺度QM/MM等分子模拟手段。通过分子模拟,人们得以对突变影响催化反应性的机制进行理解,对提高反应性的突变进行预测,对实验做出指导。然而这些模拟往往都是由研究者手动完成的,现有的分子模拟软件以及晶体结构的多样性要求大量的人工操作和文件格式校对,使得这类分子模拟能进行的数量有限。在很多研究中,研究者往往只评估离反应物最近的几个关键突变带来的影响并在这几个位置给出指导,并由于模拟的通量限制,无法发掘更广阔的序列空间中的潜力。
EnzyHTP通过四个模块自动化处理酶模拟整个工作周期会遇到的问题,从而实现了高通量的计算酶模拟。这些模块分别是:Protein Preparation模块(负责构建蛋白质结构、或是修复已有的晶体结构)、Mutant Generation模块(负责决定在哪里突变、生成突变后的结构)、Geometry Variation(负责运行MD等模拟)、Energy Engine(负责对模拟的结果进行能量计算及分析,支持MM、QM、QM/MM等多种理论方法)。

图1. EnzyHTP的四个模块
这些模块以层级递进的方式组成了EnzyHTP软件的功能架构。如果一次性输入多个序列、辅因子组成差异很大的酶,EnzyHTP将对每个酶进行结构准备——过滤结构中存在的问题、决定质子态等。之后对得到的拥有完整结构的酶,EnzyHTP支持对其进行多种规则的高通量突变,用户可以指定具体的一批突变,也可以通过某种规则产生随机突变(例如:只将底物5 Å范围内随机地将空阻较大的残基突变为较小的残基)。对于突变产生的结构,EnzyHTP支持对每个突变都进行分子动力学(MD)模拟,从而获得能评估酶在溶液中真实存在情况的一个轨迹(几何结构集合)。对于集合中的每个几何结构,EnzyHTP支持基于MD、QM、QM/MM的多种能量计算、波函数分析方式。由此得到的大量充分的基于物理模型的酶信息使得评估预测动力学催化活性变得可能。
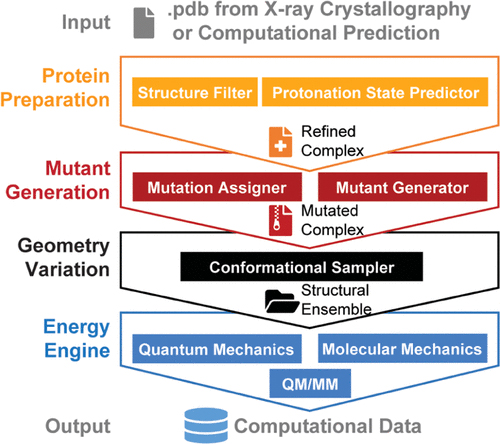
图2. EnzyHTP的功能架构
为测试软件的效率和潜力,文中使用了氟乙酸水解酶(FAcD)这一模型体系,评估了100个随机单点突变下,该酶的内部静电场的变化对反应物C-F键的影响。这个自动化工作流包含修复FAcD-氟乙酸复合物晶体结构中缺失的氢原子、确定质子态,随机产生100个单点突变后的结构,调用Amber进行1 ns的分子力学模拟,在模拟产生的轨迹中等间隔进行采样得到100个几何结构,对每个几何结构调用Gaussian16进行一次QM Cluster计算得到底物的波函数,从而调用Multiwfn对波函数进行分析得到底物C-F键的偶极,对每个几何结构中酶的电场在底物C-F键方向的分量进行计算,从而结合偶极得到酶的电场对C-F键的去稳定化/稳定化电势能。
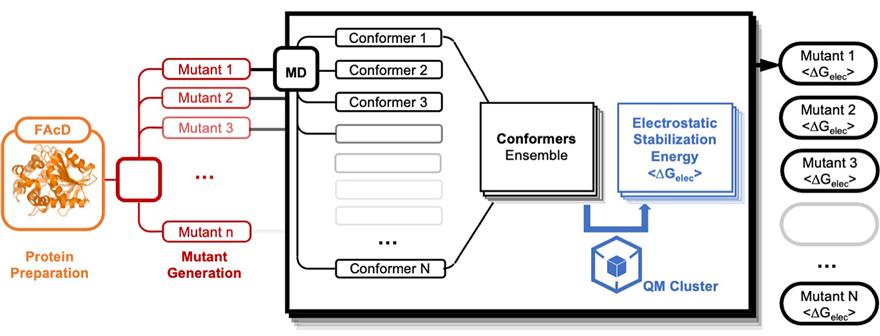
图3. 使用FAcD体系的测试工作流程图示
整个工作流程使用10个GPU核心和160个CPU线程在7小时内完成全部计算。虽然使用的QM理论层级较低(只是为了演示软件可能的工作流),但是很难想象如果整个流程都由研究者手动完成将花费多少时间(100个Amber输入文件、10000个高斯/Multiwfn输入文件)。
作者在文中对计算结果进行了分析以展示EnzyHTP给出结果中信息的价值和丰富程度,有兴趣的朋友可以阅读原文了解。

图4. 100个随机单点突变计算结果的分析
文中的软件EnzyHTP的代码开源在GitHub上 [1]。软件本身还处于预发行阶段,作者在GitHub上标记该软件的文档还在准备中,代码本身也在进行重构和打包。期待正式版EnzyHTP的上线,为生物化学研究者解决突变和序列评估的问题,从而真的实现用酶来催化各种非天然反应。
这一成果近日发表在J. Chem. Inf. Model.上,论文的通讯作者为范德堡大学Zhongyue Yang教授,论文第一作者为范德堡大学博士生Qianzhen (QZ) Shao同学。
原文(扫描或长按二维码,识别后直达原文页面,或点此查看原文):

EnzyHTP: A High-Throughput Computational Platform for Enzyme Modeling
Qianzhen Shao, Yaoyukun Jiang, and Zhongyue J. Yang*
J. Chem. Inf. Model., 2022, 62, 647–655, DOI: 10.1021/acs.jcim.1c01424
参考资料:
1. EnzyHTP的代码
https://github.com/ChemBioHTP/EnzyHTP
如果篇首注明了授权来源,任何转载需获得来源方的许可!如果篇首未特别注明出处,本文版权属于 X-MOL ( x-mol.com ), 未经许可,谢绝转载!












































 京公网安备 11010802027423号
京公网安备 11010802027423号