这个小bug,可能“坑”了上百篇化学论文
结构解析可以说是整个有机化学的核心技术。说白了,我们总得知道自己做的是个什么东西吧?做得什么都说不清楚,还谈什么性质、吹什么方法?说起解析结构,恐怕最有发言权的要说那些天然产物研究人员。与合成产物的结构鉴定不同,天然产物的结构鉴定几乎全部是未知的结构信息,加之天然产物多有手性中心,这就练就了该领域研究人员的一双慧眼。受到目前物理技术的制约,并不是所有化合物都能利用X-射线单晶衍射技术确定手性中心的立体构型。近些年,计算机硬件和计算化学迅速发展,在化学及其相关学科都能看到计算化学的影子。算能量,算构象,算反应,算波谱数据,算相互作用,甚至连生命起源都能算(剧毒氰化物,或是生命起源?),大有“万物皆可算”的感觉。利用计算化学确定手性中心立体构型更是不在话下,早已被全世界的学界认可。可是,凡事总有瑕疵,算来算去也难免算错。咱们今天就看一篇有趣的文章,来自夏威夷大学马诺阿分校Philip G. Williams等人,发表在Organic Letters上,主要工作是鉴定一些新发现的天然产物。
本君在这里就不给大家详细讲述该文章是如果解析核磁(NMR)图谱确定化合物结构骨架的,感兴趣的同行请直接阅读原文。老规矩,咱们直接上干货。这篇文章从蓝细菌Leptolyngbya sp.中鉴定出这样几个结构新颖的化合物。
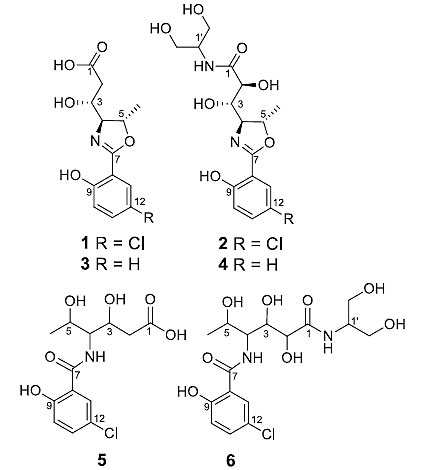
图片来源:Org. Lett.
对于化合物1的手性中心构型鉴定,作者也利用计算化学对其8个异构体进行了碳谱化学位移的计算,通过计算值与实测值的比较确定化合物的立体构型。

图片来源:Org. Lett.
这一切看上去都是那么的正常,可是意外总是不期而遇。当他们团队成员重复计算结果时发现,同样的结构输入文件,同样的软件,在不同的操作系统上(Windows、Linux、Mac)计算出的结果居然不一样!!!大白话说,就好比是一个牌子的计算器,在我家算1+1=2,在你家算1+1=3.1,在他家算1+1=1.8。这着实让人难以接受啊 …… 
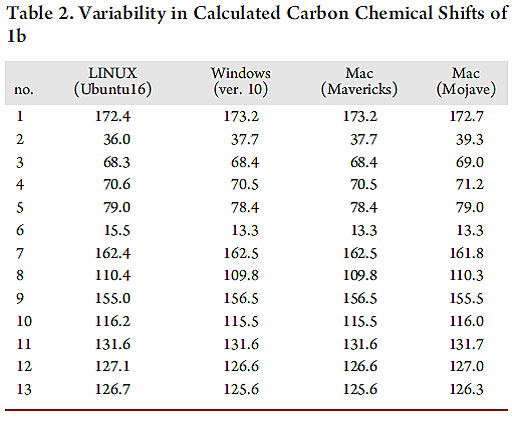
图片来源:Org. Lett.
于是,一组问号三连击出现了。为什么会这样?我该怎么办?到底相信谁?作者在经历了这样的灵魂拷问之后,给出了他们的答案。他们使用的是Willoughby等人2014年在Nature Protocols 上公开的Python脚本程序 [1]。在分析这个程序运行过程后作者发现这样的规律:对同一个输入文件进行计算时,在Windows平台这个程序会输出两组文件,一组命名为*opt_freq-*-ID.out存储了能量与频率,另一组命名为*nmr-*-ID.out存储了化学位移,程序会按照文件名自动匹配。在Linux平台运行程序时,虽然也生成了这两组文件,但是Linux系统中文件排序取决于使用者设置的环境变量。因此,计算程序会出现将能量频率文件与化学位移文件匹配错误的情况。这就造成了同一个软件在不同操作系统上运行相同的输入文件却得到了不完全一样的结果。
文章读到这,本君已经很佩服作者的能力了,这种瑕疵(程序员们一般称之为“bug”)都能找到。更绝的是,作者找到问题后直接加了几行代码把程序改了 [2],改完之后放到各个操作系统上一运行。嗯……结果终于一样了。字里行间都能感受到那种处女座“强迫症患者”的释然。
文章最后,作者“轻描淡写”地提道,这几年用这个程序解析的结构可不少啊(该文至今被引用超过160次),这些作者应该做一做不同系统的计算检验。

图片来源:Nature Protocols [1]
本君最后邪恶地出个计算题,那些引用了这篇文章来计算化学位移的研究者看到这篇OL或者本文之后,心理阴影面积大概有多大?
原文(扫描或长按二维码,识别后直达原文页面,或点此查看原文):

Characterization of Leptazolines A–D, Polar Oxazolines from the Cyanobacterium Leptolyngbya sp., Reveals a Glitch with the “Willoughby–Hoye” Scripts for Calculating NMR Chemical Shifts
Jayanti Bhandari Neupane, Ram P. Neupane, Yuheng Luo, Wesley Y. Yoshida, Rui Sun, Philip G. Williams
Org. Lett., 2019, 21, 8449-8453, DOI: 10.1021/acs.orglett.9b03216
参考资料:
1. A guide to small-molecule structure assignment through computation of (1H and 13C) NMR chemical shifts. Nature Protocols, 2014, 9, 643–660, DOI: 10.1038/nprot.2014.042
https://www.nature.com/articles/nprot.2014.042
2. Philip G. Williams修改后的程序:
https://pubs.acs.org/doi/suppl/10.1021/acs.orglett.9b03216/suppl_file/ol9b03216_si_003.zip
(本文由乐只君子供稿)
如果篇首注明了授权来源,任何转载需获得来源方的许可!如果篇首未特别注明出处,本文版权属于 X-MOL ( x-mol.com ), 未经许可,谢绝转载!













































 京公网安备 11010802027423号
京公网安备 11010802027423号