致敬Robert Roeder & 纪念真核生物RNA聚合酶发现50周年——转录调控研究简史
本文获“BioArt”授权转载,版权归“BioArt”所有,任何转载需获得“BioArt”授权。
编者按: 今年是杰出的基因转录调控研究大师Robert G. Roeder教授(小同行私下里一般直接简称“Bob”,他是美国国家科学院院士、美国艺术与科学学院院士、欧洲EMBO外籍会士、2000年盖尔德纳基金会国际奖、2003年拉斯克基础医学奖、2014年汤姆森路透社引文桂冠奖获得者)发现真核生物RNA聚合酶50周年(Bob时年27岁),清华大学结构生物学高精尖创新中心借此机会,将于8月15日8:30在清华大学主楼接待厅举办“1969-2019: 50 Years of Eukaryotic Transcription”会议,以致敬Robert G. Roeder教授在生命医学领域做出的杰出贡献(详细信息详见此前BioArt的推送)。此外,“2019年度表观遗传与染色质生物学大会” 将于2019年8月15-18日在北京召开,8月15日下午Bob还将应邀第一个做一小时大会特邀报告。今年5月,BioArt借Bob访问复旦大学生科院之机,特别邀请了Bob学术报告的主持人王纲教授撰文以纪念真核生物RNA聚合酶发现50周年并致敬Bob,王纲教授欣然接受并与课题组成员一道完成该文,BioArt现将全文编辑如下,以飨读者!

1942年6月,Robert G. Roeder出生在印第安纳州一个德裔美国人经营的农场里。他父亲毕生的愿望就是尽可能地扩大自家农场的规模。年幼的Robert Roeder在上学前和放学后都要帮家里干农活。农场养着各种各样的动物,Bob经常要扛着重达100磅的饲料,跑遍农场的各个角落给动物喂食。辛勤劳动赋予Bob Roeder强壮健硕的体格,体能上的优势还使他成为高中橄榄球队中出色的一员。虽然父母的教育程度不高,但他们刻苦勤奋自律的性格为小时候的Robert Roeder树立了榜样,营造了一个非常良好的家庭氛围。Bob优良的品德为大家有目共睹,在高中毕业典礼上还曾作为毕业学生代表上台致告别词。事实上,在后来的学术生涯中,正是因为他不畏艰苦、持续努力的工作使他成为了一名出色的生物化学家,并成为了真核基因转录调控领域研究的一代宗师【1】。
Robert G. Roeder对转录的兴趣起源于1963年,那时的他正在美国印第安纳州著名男校瓦伯希学院(Wabash College)化学专业念本科。在生物化学课上,他了解到了当时的科学家正在致力于理解基因转录的奥秘。后来他来到了华盛顿大学Bill Rutter实验室攻读博士,但Rutter正在研究的是胰腺的发育过程,实验室还没有涉及转录相关的研究,可Rutter教授颇有前瞻性,他有意将研究范围拓展到相关的RNA的合成上。这才让Robert Roeder得以启始了RNA聚合酶相关的研究工作【2】。
年轻的Bob Roeder一开始就奔着解开真核生物如何阅读“基因密码”这一宏大目标而去,他设定了他的具体科学目的是要以可控的方式内让转录发生在试管里。但在上个世纪六十年代,研究真核生物转录的手段却相当有限,因为真核生物的RNA聚合酶紧紧地结合在染色体上,这使得纯化工作变得十分困难。相比之下,由于原核生物的RNA聚合酶(编者注:关于原核生物RNA聚合酶,早在1955年,Marianne Grunberg-Manago 和 Severo Ochoa就声称他们分离到了能够催化RNA合成的酶,当时他们命名为“polynucleotide phosphorylase(多聚核苷磷酸化酶)”,然而后来发现这种酶不需要DNA作为模板来合成RNA,但是他们发现的酶在M.Nirenberg和J.H.Matthaei合成第一个遗传密码子中发挥了重要作用,S.Ochoa还因次阐明RNA生物合成机制而获得了1959年诺贝尔生理学或医学奖。由于Ochoa的发现引起了美国科学家Jerard Hurwitz对寻找真正RNA聚合酶的兴趣,于是Hurwitz在1959年开始从大肠杆菌中分离RNA聚合酶的研究。其时,1959年Samuel B. Weiss组报道了大鼠肝脏的核抽提物和再有四种rNTPs的情况下物能够催化RNA合成反应,研究发表在JBC上,随后1960年Hurwitz组和Audrey Stevens在大肠杆菌的抽提物重复了类似的实验,研究结果同期发表在BBRC上,稍晚些时候James Bonner组利用豌豆的核抽提物也做出了类似的实验结果,同样发表在当年的BBRC上。然而这只是开始,离真正的分离纯化RNA聚合酶还有一段路程。之后,Hurwitz和他的博后John J. Furth合作终于在1962年从大肠杆菌中分离纯化出真正意义上的RNA聚合酶,这一经典工作发表在当年的JBC上。)含量大且可溶性强,大量关于细菌转录机制的研究正在如火如荼地进行着。
面对眼前的困难,Roeder意识到:他必须开发一种新的方法来将RNA聚合酶与DNA和组蛋白分离开来。前期的一系列尝试并没有取得太多的进展,但Roeder偶然间看到Mirsky和Pollister1942年发表的一篇研究组蛋白的论文让他眼前一亮。又经过了一系列的摸索,Roeder发现:先用高浓度盐溶液处理细胞核——这会导致细胞核破裂及组蛋白解聚,紧接着利用超声波将DNA打碎,同时释放结合在DNA上的RNA聚合酶,最后以低浓度盐溶液快速稀释,使组蛋白选择性地沉淀掉。通过这个方法得到的上清液就含有可溶的RNA聚合酶。上述溶液通过“DEAE-Sephadex”离子交换柱,再用硫酸铵溶液洗脱并检测洗脱液的酶活。最终在1969年情人节(2月14日)凌晨3点钟,Roeder得到了下面这张图。
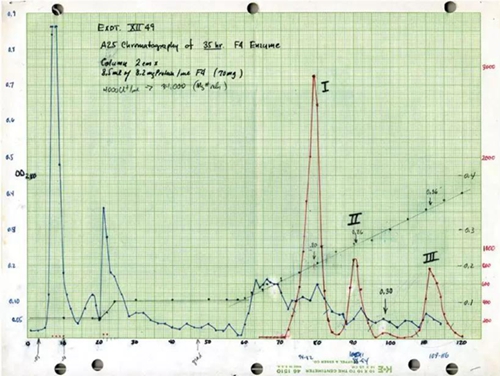
蓝色曲线代表总蛋白,红色曲线表示RNA聚合酶的活性。
Roeder在看到图中的三个RNA聚合酶的活性峰时,他兴奋得像是跳出洗澡盆的阿基米德!紧接着的一个礼拜,他几乎没有怎么睡觉,马不停蹄地完成了后续的相关实验。最初的RNA聚合酶是从海胆胚胎里分离的,这无疑是一个明智的选择,因为海胆胚胎里富含三种RNA聚合酶,采海胆更是一桩乐事,让Roeder在忙碌的实验之余能够稍微放松一下身心(编者注:笔者好几次和朋友吃到海胆的时候都会提到Bob选择海胆分离真核RNA聚合酶的历史,有一次是和党生老师,他是很清楚这段发现史的,希望同行读者今后吃海胆的时候也能记住Bob的重要发现)。

Robert Roeder在富卡海峡寒冷的海水中捕捞海胆(1968年)
然而,文章投稿的过程并不是很顺利。Nature杂志以“大家普遍不感兴趣”(“lack of general interst”)为理由拒绝了Roeder的文章。导师Bill Rutter据理力争,Nature最后才发表接受了这篇具有划时代意义的文章(下图)【3】。

一年之后,Robert Roeder在PNAS上发表另一篇文章,指出RNA聚合酶I(Pol I)定位在核仁上,核仁正是rRNA转录的地方,而RNA聚合酶II(Pol II)、RNA聚合酶III(Pol III)则是聚合在核质内【4】。这个发现说明了不同的RNA聚合酶可能负责转录不同类别的基因。
这时候的Roeder已经开始在华盛顿卡内基研究所Donald Brown实验室做博后了(编者注:Bob尽管只在Brown实验室做了短短两年博后,发表了一篇似乎不太显眼的文章,但是因为Brown实验室有很好的非洲爪蟾实验体系,为后面Bob独立后利用该体系构建体外无细胞系统研究真核基因转录调控奠定了重要的基础,该系统影响了后面几十年的体外转录研究。)。博后期间,他和同事试图用纯化出来的RNA聚合酶I和人工合成的DNA精确地转录rRNA,但结果却让他们失望。Roeder开始思考:难道真核生物的转录还需要其它的一些因子?又或是染色质环境是转录所必须的?
1971年,美国圣路易斯市的Washington University School of Medicine(WUSM)为Robert Roeder提供了助理教授的职位。带着博后期间未能解决的问题,Roeder在这里开启了他对真核基因转录调控研究的新篇章。他率领几个满腔热血的学生发现:三种RNA聚合酶对蘑菇中的一种毒素α-鹅膏蕈碱(α-amanitin)有不同的敏感性。利用不同α-鹅膏蕈碱浓度处理细胞,检测所转录出来的RNA,他们发现:rRNA是由Pol I转录出来的;Pol II能够合成腺病毒的pre-mRNA,而Pol III则能够转录5S RNA、tRNA以及腺病毒的VA RNA【5,6】。与此同时,Roeder实验室从小鼠浆细胞瘤中纯化三种RNA聚合酶的不同亚基,发现三种聚合酶的亚基组成有很大区别。这说明真核生物RNA聚合酶并不是同一种酶的三种不同变化,而是三种相对独立的转录机器。
既然已经搞清楚了三种RNA聚合酶的功能,下一个关键的科学问题就是如何才能起始转录?1976年,Roeder将目光对准了5S RNA,因为这个基因相对较小而且高度重复。博后期间的Roeder已经知道5S RNA在非洲爪蟾的卵细胞中是活跃的,因此他指导学生Carl Parker从非洲爪蟾卵细胞中分离了染色质,将分离出来的染色质与纯化的RNA聚合酶III进行孵育,结果大量的5S RNA瞬间被转录出来。若将RNA聚合酶III换成RNA聚合酶I或II,又或是将染色质换成人工合成的5S RNA的DNA模板都检测不到转录的发生【7】。这是Robert Roeder期待已久的结果,也是人类历史上第一次在无细胞体系中使用真核生物RNA聚合酶进行转录。Roeder大胆地猜测,染色质上存在着一些RNA聚合酶相关联的因子,而这些因子是转录发生所必需的。当时的表观遗传学尚未兴起,这些工作和思想无疑是具前瞻性的。
经过反复不断的体系优化,至1979年,Roeder实验室已经能够做到在体外转录任意类型的RNA【8】。同年在Cell杂志上的论文利用纯化出来的RNA聚合酶II和粗提的亚细胞组分成,以腺病毒DNA片段为模板成功转录出了mRNA【9】。这篇文章引起了领域内极大的关注,同时也向世人展示了一种研究转录的强大工具。体外转录系统现在仍然被广泛地使用,其严谨缜密的方法将有机体复杂的机制简化,让科学家有机会去了解单个蛋白或蛋白复合物在转录调控中的功能。这种技术的建立成为攻克基因转录调控难题的一把利剑。
在此之后,很多实验室开始寻找转录起始所必须的DNA序列,而Roeder实验室则认为细胞抽提物中能够起始转录的未知蛋白因子更为重要。于是他们回到了冷库中,不断地优化蛋白质纯化的方法,解析HeLa细胞核抽提物的多个组分。1980年,Roeder实验室纯化并鉴定出RNA聚合酶III起始转录所必须的TFIIIC、TFIIIB。类似的方法迅速被同行效仿,越来越多的转录因子被鉴定。其中包括第一个被证实启动子特异转录因子SP1(Bob Tjian,1984)、调控热休克蛋白转录的HSF(Carl Parker,1985)、以及结合TFIID的USF。这波研究的热潮导致了数百个能够识别特异DNA序列的转录因子得以发现,这些转录因子有的广泛存在于细胞中,有的却是组织细胞特异的。那些任何启动子中RNA聚合酶II起始转录所必须的蛋白被称为“通用转录因子”(General transcription factors,GTFs),简称为“TFIIX”,其中“X”为不同的字母,用于区分各个因子(包括THIID、TFIIA、TFIIB、TFIIE、TFIIF、TFIIH、TFIIS等等)。
到了90年代,Roeder以及领域内为数不多的几个实验室采用高度纯化的RNA聚合酶II和通用转录因子所重构的无细胞系统来研究调控因子的功能,而在对这个系统的不断优化过程中,关键的转录辅因子——中介体复合物(Mediator complex)得以发现。
Mediator存在的最早证据来源于对酵母的生化研究。1988年,Mark Ptashne实验室发现在酵母中过表达转录激活子GAL4能够抑制基因转录,这种现象被称作“竞争抑制(squelching)”【10】。当时认为,酵母中的激活子有“共同靶分子”,过多的激活子之间对靶分子的互相竞争最终会造成转录抑制,而当时已有的实验结果已经表明激活子可以直接和通用转录因子TFIID和RNA聚合酶II相互作用,“共同靶分子”因此被认为是RNA聚合酶II转录机器的一部分。1990年Roger Kornberg实验室以及1991年Robert Roeader实验室的发现却挑战了“激活子通过直接与RNA聚合酶ll和通用转录因子作用激活转录”这个观点【11,12】。Kornberg实验室发现,在“竞争抑制”现象存在的系统中添加更多纯化的通用转录因子TFIID和RNA聚合酶ll仍然无法消除这种“抑制”作用,但添加的如果是部分纯化的酵母组分却能够消除这种“抑制”,因此部分纯化的酵母组分中可能存在一种新的能够介导转录激活的因子,他们称之为“中介体(Mediator)”。Roeader实验室在体外重构系统中发现添加粗提的TFIID能放大激活子介导的转录,而纯化后的TFIID不能够行使这样的功能,他们把TFIID中包含的这种组分称为“新活性”。
综合看来,这些研究虽然表明中介体的存在,但中介体参与转录激活的直接证据是缺失的。到了1991年,这项直接证据最终由Kornberg实验室证明,他们通过建立重构酵母RNA聚合酶体外转录系统直接表明激活子的转录激活需要中介体粗提物的参与【13】。1994年,Kornberg实验室又首次纯化出了由大约二十个亚基组成的酵母中介体,这项重要的研究工作具有以下几个重要贡献:1)表明转录激活可以在一个特定、明确的系统中通过加入RNA聚合酶II和纯化的重要的基本因子进行;2)没有中介体存在,转录激活无法进行,否定了先前所认为的转录激活是由转录因子直接结合通用转录因子或RNA聚合酶II来激活转录的观点;3)中介体能够和RNA聚合酶II CTD作用,并对CTD的磷酸化具有重要影响【14】。
应该说,与此同时甚至更早,Richard Young实验利用酵母遗传学方法也发现了中介体。1989年Richard Young实验室利用RNA聚合酶II CTD截短的突变型酵母在低温下无法生长这一系统筛选到能解除生长抑制的突变,他们将筛选到的突变称为SRB(suppressor of RNA polyperase B)【15】;1993到1995年,Young实验室发表多篇文章报道一系列的SRB蛋白在体外转录系统中能够介导转录激活【16-18】。当Kornberg在1994年纯化和鉴定出中介体后,他们发现中介体的多个亚基蛋白就是1989年Richard Young实验室采用酵母遗传学筛选到的SRB蛋白。
回过头看,可以说,中介体的纯化和鉴定是生物化学和遗传学的一次美丽且重要的相逢:生物化学研究建立了中介体参与转录激活的体外系统;遗传学研究则通过筛选转录抑制的突变体发现中介体在体内也具有调控转录的功能。这些研究揭示了中介体复杂而重要的调控功能。人源中介体在1996年最终被Roeder实验室从Hela细胞中纯化出来,他们称之为TRAPs(thyroid hormone receptor-associated proteins)【19】,但当时TRAPs被认为只是TR受体特异的辅因子。到1999年, Danny Reinberg、Robert Tjian、Leonard Freedman、Arnold Berk以及Robert Roeder等实验室几乎同时发表文章发现了名称各异的转录中介体复合物,分别称为NAT(negative regulator of activated transcription)【20】、ARC(activator-recruited factor)【21】、CRSP(cofactor required for Sp1 activation)【22】、DRIP(vitamin-D-receptor interacting protein)、TRAP/SMCC(suppressor of RNA polymerase B(SRB)-mediator-containing cofactor)【23】、human Mediator【24】。综合看来,转录中介体复合物就是一个巨大的、组分可变且位于转录因子和RNA聚合酶之间的“桥梁”分子。1999年Robert Kingston在一篇Nature的评论文章中指出,上述多个实验室发现的存在“共享亚基”的中介体复合物似乎表明中介体能以多种形式存在于生物体中,中介体复合物的分子量如此巨大也暗示其能与众多的转录激活子相互作用,并参与调节转录的不同步骤【25】。为了方便理解和研究,2004年,领域内的46位科学家集体在Mol Cell杂志发表论文对已发现的转录中介体复合物进行统一命名,从MED1、MED2、MED3、……至MED31,并保留CDK8和CycC两个亚基的原始命名【26】。

Bob Roeder和施扬(前排左)、雷鸣(前排右),后排左起分别为王纲、翁杰敏、于明、孙晓健、黄传新、蓝斐在上海合影(2018)
近年来,随着人们对染色质的进一步了解,染色质科学与转录调控的关系越来越密切。目前Robert Roeder正在致力于将染色质上的各种修饰与许多转录相关事件的“因果关系”建立起来,如甲基化酶和乙酰化酶SET1和p300,去甲基化酶UTX,甲基化酶MLL4和乙酰化酶p300之间的相互协调等。这些工作为理解转录与表观遗传学之间提供了新的视角。

2018年4月,Bob与哈佛大学张毅教授在灵隐寺合影(张毅教授以及中科院生物物理研究所的朱冰研究员与李国红研究员的博后导师Danny Reinberg是Bob的博后,所以按学术辈分Bob是三位的亲师爷,本次全国染色质大会可以集齐)。
真核生物的基因调控是极其复杂的,五十年光阴白驹过隙,一代又一代科学家的不懈努力造就了该分子生物学领域今日的辉煌。而直至今日,Robert Roeder仍然活跃在学术研究的最前沿。半个世纪已过,他也已经从那位冰冷海水中捕海胆的健硕年轻人变成了学术领域的泰山北斗。他是这段历史的实践者,更是这段历史中最伟大的书写者。
编后记:BioArt在创办之初曾专题介绍过Bob的重要学术贡献,如今将近4年过去了,早该认真renew一下了,笔者去年同Bob交流的时候他还曾开玩笑的说笔者可以写2.0版了(他虽不会直接看中文版,但是他亲口告诉笔者他的中国太太看过,并转告给了他)。等到这篇文章顺利出来,笔者总算如释重负。在此,必须真诚的感谢复旦大学王纲教授团队付出的智慧和汗水!
Bob与中国非常有缘分,培养了一批优秀的中国科学家,多次到访中国,最近几年他每年都会到访国内几次,现在虽然年事已高,但是仍然精神矍铄、精力充沛,仍然活跃在科研第一线。Bob是当之无愧的学术大师!祝福老先生健康长寿!

2018年4月,Bob和张毅等应施一公教授邀请参观西湖大学。

2018年4月,Bob与朱冰交流,中间戴墨镜的为Science杂志的editor Steve Mao。

2018年4月,Bob在上海一家酒店大堂,利用短暂的等待时间处理邮件(Wifi是笔者的手机热点提供)
参考文献
1. https://www.the-scientist.com/first-person/robert-g-roeder-51013
2. Roeder,R. G. Lasker Basic Medical Research Award. The eukaryotic transcriptionalmachinery: complexities and mechanisms unforeseen. Nat Med 9, 1239-1244,doi:10.1038/nm938 (2003).
3. Roeder,R. G. & Rutter, W. J. Multiple forms of DNA-dependent RNA polymerase ineukaryotic organisms. Nature 224, 234-237, doi:10.1038/224234a0(1969).
4. Roeder,R. G. & Rutter, W. J. Specific nucleolar and nucleoplasmic RNA polymerases.Proc Natl Acad Sci U S A 65, 675-682, doi:10.1073/pnas.65.3.675(1970).
5. Weinmann,R. & Roeder, R. G. Role of DNA-dependent RNA polymerase 3 in thetranscription of the tRNA and 5S RNA genes. Proc Natl Acad Sci U S A 71,1790-1794, doi:10.1073/pnas.71.5.1790 (1974).
6. Weinmann,R., Raskas, H. J. & Roeder, R. G. Role of DNA-dependent RNA polymerases IIand III in transcription of the adenovirus genome late in productive infection.Proc Natl Acad Sci U S A 71, 3426-3439,doi:10.1073/pnas.71.9.3426 (1974).
7. Parker,C. S. & Roeder, R. G. Selective and accurate transcription of the Xenopuslaevis 5S RNA genes in isolated chromatin by purified RNA polymerase III. Proc Natl Acad Sci U S A 74, 44-48, doi:10.1073/pnas.74.1.44(1977).
8. Ng, S. Y., Parker, C. S., & Roeder, R. G. (1979). Transcription of cloned Xenopus 5S RNA genes by X. laevis RNA polymerase III in reconstituted systems. Proceedings of the National Academy of Sciences, 76(1), 136-140.
9. Weil, P. A., Luse, D. S., Segall, J., & Roeder, R. G. (1979). Selective and accurate initiation of transcription at the Ad2 major late promotor in a soluble system dependent on purified RNA polymerase II and DNA. Cell, 18(2), 469-484.
10. Gill,G. & Ptashne, M. Negative effect of the transcriptional activator GAL4. Nature 334, 721-724, doi:10.1038/334721a0 (1988).
11. Kelleher,R. J., 3rd, Flanagan, P. M. & Kornberg, R. D. A novel mediator betweenactivator proteins and the RNA polymerase II transcription apparatus. Cell 61, 1209-1215, doi:10.1016/0092-8674(90)90685-8 (1990).
12. Meisterernst,M., Roy, A. L., Lieu, H. M. & Roeder, R. G. Activation of class II genetranscription by regulatory factors is potentiated by a novel activity. Cell 66, 981-993, doi:10.1016/0092-8674(91)90443-3 (1991).
13. Flanagan,P. M., Kelleher, R. J., 3rd, Sayre, M. H., Tschochner, H. & Kornberg, R. D.A mediator required for activation of RNA polymerase II transcription in vitro.Nature 350, 436-438, doi:10.1038/350436a0 (1991).
14. Kim,Y. J., Bjorklund, S., Li, Y., Sayre, M. H. & Kornberg, R. D. A multiproteinmediator of transcriptional activation and its interaction with the C-terminalrepeat domain of RNA polymerase II. Cell77, 599-608,doi:10.1016/0092-8674(94)90221-6 (1994).
15. Nonet,M. L. & Young, R. A. Intragenic and extragenic suppressors of mutations inthe heptapeptide repeat domain of Saccharomyces cerevisiae RNA polymerase II. Genetics 123, 715-724 (1989).
16. Thompson,C. M., Koleske, A. J., Chao, D. M. & Young, R. A. A multisubunit complexassociated with the RNA polymerase II CTD and TATA-binding protein in yeast. Cell 73, 1361-1375, doi:10.1016/0092-8674(93)90362-t (1993).
17. Koleske,A. J. & Young, R. A. An RNA polymerase II holoenzyme responsive toactivators. Nature 368, 466-469, doi:10.1038/368466a0(1994).
18. Liao,S. M. et al. A kinase-cyclin pair inthe RNA polymerase II holoenzyme. Nature374, 193-196, doi:10.1038/374193a0(1995).
19. Fondell,J. D., Ge, H. & Roeder, R. G. Ligand induction of a transcriptionallyactive thyroid hormone receptor coactivator complex. Proc Natl Acad Sci U S A 93,8329-8333, doi:10.1073/pnas.93.16.8329 (1996).
20. Sun,X. et al. NAT, a human complexcontaining Srb polypeptides that functions as a negative regulator of activatedtranscription. Mol Cell 2, 213-222 (1998).
21. Naar,A. M. et al. Composite co-activatorARC mediates chromatin-directed transcriptional activation. Nature 398, 828-832, doi:10.1038/19789 (1999).
22. Pugh,B. F. & Tjian, R. Mechanism of transcriptional activation by Sp1: evidencefor coactivators. Cell 61, 1187-1197,doi:10.1016/0092-8674(90)90683-6 (1990).
23. Ito,M. et al. Identity between TRAP andSMCC complexes indicates novel pathways for the function of nuclear receptorsand diverse mammalian activators. Mol Cell3, 361-370 (1999).
24. Boyer,T. G., Martin, M. E., Lees, E., Ricciardi, R. P. & Berk, A. J. MammalianSrb/Mediator complex is targeted by adenovirus E1A protein. Nature 399, 276-279, doi:10.1038/20466 (1999).
25. Kingston,R. E. A shared but complex bridge. Nature399, 199-200, doi:10.1038/20302(1999).
26. Bourbon,H. M. et al. A unified nomenclaturefor protein subunits of mediator complexes linking transcriptional regulatorsto RNA polymerase II. Mol Cell 14, 553-557,doi:10.1016/j.molcel.2004.05.011 (2004).

如果篇首注明了授权来源,任何转载需获得来源方的许可!如果篇首未特别注明出处,本文版权属于 X-MOL ( x-mol.com ), 未经许可,谢绝转载!












































 京公网安备 11010802027423号
京公网安备 11010802027423号