Angew:计算化学是灯,照亮结构解析的坑
又是一年毕业季,对于做天然产物分离纯化结构鉴定的小伙伴来说,最难言的苦衷,莫过于日日夜夜在实验室辛苦耕耘,收获了许多“纯品”化合物,却面对一大堆NMR波谱数据“夙夜忧叹”:二维谱上这些“大坑小坑”到底是谁连谁?纯品在手,解不出结构怎么办?

图片来源于网络
当然,感到郁闷的还有做天然产物结构修饰和全合成的小伙伴,按照文献上的结构合成出来的化合物,怎么波谱数据对不上呢?莫不是被做分离纯化的那帮人坑了?大家都是靠化合物结构混饭吃的同道中人,难免要发出“本是同根生,相煎何太急”的感慨。更郁闷的估计就是拿某化合物研究了好多药理活性,最后发现结构错了(相关阅读:药物结构错了?你可能申请了一个假专利……; 你是我的眼:NMR在结构确证中的应用策略)。

图片来源于网络
这个时候,遇到困难的小伙伴最需要的,不是心灵和胃口上的慰藉,而是跪求各类“大佬”带路,指点迷津,让“坑底之蛙”早日跳出结构解析的“连环坑”。

图片来源于网络
天然产物的结构为何容易错解?笔者认为有三点原因:(一)现有的波谱数据太少,对于含季碳较多的化合物(如不饱和度极高的生物碱),缺少关联信号进行解析;(二)采集到的波谱数据太多,造成碳氢谱以及二维谱信号大量重叠(如复杂萜类化合物),信号难以归属进行解析;(三)化合物立体结构复杂,含有若干手性中心(如立体笼状结构、桥环化合物),目前能够直接鉴定手性中心构型的技术手段非常有限。面对上述结构谜题,恰当运用计算化学也许能助小伙伴们一臂之力。本文就带大家来看一个经典案例。
美国丹佛大学的Andrei G. Kutateladze(点击查看介绍)、澳大利亚昆士兰大学的Elizabeth H. Krenske(点击查看介绍)和Craig M. Williams(点击查看介绍)三位教授所组成的研究团队(图1)近日在Angew. Chem. Int. Ed.发文,运用计算化学方法对文献中的一些含氧桥环结构(oxo-bridged,简称“氧桥”)类天然产物进行了结构解析和修正。

图1. Andrei G. Kutateladze教授(左)、Elizabeth H. Krenske教授(中)和Craig M. Williams教授(右)。图片来源:丹佛大学、亚昆士兰大学[1-3]
氧桥类天然产物含有氧取代的桥头碳(也称anti-Bredt结构,最初是由化学家Bredt定义)。含这类结构的天然产物在自然界中被大量地分离鉴定出来,且已报道的化合物往往具有不寻常的分子框架、成键基序和附加官能团,其结构的疑问也颇多。因此,对于计算化学研究来说,这类化合物是检验结构解析方法准确性的试金石。
在这类天然产物结构解析中,他们采用基于烯烃应变能(olefin strain energy, OSE)的计算方法来判断含桥头烯天然产物的稳定性,以此评估结构的合理性。之前的研究将桥头烯按照OSE的特征性范围分为3种类型,即可孤立(isolable)、可观察(observable)、不稳定(unstable)。他们通过分子力学力场(molecular mechanics forcefield)来计算OSE,并且证实查到的桥头烯天然产物都属于可孤立类型。随后,他们在推测若干化合物假定结构时,发现了一些结构存在疑问,比如化合物1(rosacedrenoic acid)和2(piperkadsin C)(图2),因为这些桥头烯被归入不稳定类型。以往这种分类方法在只含碳氢的桥头烯中表现很好,却没有应用到杂环类桥头烯(比如氧取代)上,所以,他们计划将基于OSE的鉴定方法应用到氧取代桥头烯中,并结合核磁NMR计算,以验证一系列含氧桥头烯结构是否正确。

图2. 化合物1和2的结构及OSE值. 图片来源:Angew. Chem. Int. Ed.
首先,他们运用OPLS_2005力场(OPLS_2005 forcefield)设置,调试性计算16个双环氧桥烯(图3)。这些烯烃本身,或其母结构含有可孤立型桥头烯。其OSE的范围较宽(从-3.9到24.7 kcal mol-1)。其中,有5个化合物(5, 8, 10, 13, 17)是同系物,它们的氧桥和所有的碳结构造成OSE差距在1.2到4.5 kcal mol-1之间,因此无明显系统性变化。最紧张的孤立型桥头烯是17,其OSE为24.7 kcal mol-1,比18高3 kcal mol-1。OSE准确地预测出17和18是可孤立型的化合物。
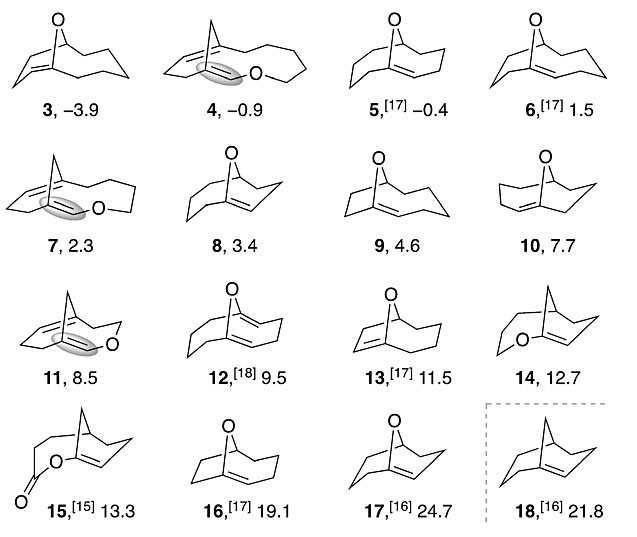
图3. 调试性计算选择的含氧桥头烯(3-18)及OSE值。图片来源:Angew. Chem. Int. Ed.
虽然,计算孤立型氧桥烯烃的OSE范围直接明了,但是Andrei G. Kutateladze教授等人发现,分析可观测或不稳定性结构就不那么清晰了。困难在于这两类氧桥烯烃更活泼,文献上少有关注,且从可观测到不稳定状态过渡的结构特点也研究甚少。于是,他们在计算中考虑了氧原子的存在对计算OSE值的影响,发现从可孤立型到可观测型再到不稳定型的过渡态OSE一般在20到30 kcal mol-1,OSE值超过30 kcal mol-1预示化合物属于不稳定型。
在此研究基础上,他们计算了文献中出现的11个氧桥天然产物(19到29,图4)。计算出OSE的范围从-13.9 kcal mol-1到32.1 kcal mol-1,只有hugonianene A(29)的OSE值超过30 kcal mol-1,被归入不稳定型。其余化合物的结构准确性都得到了验证。
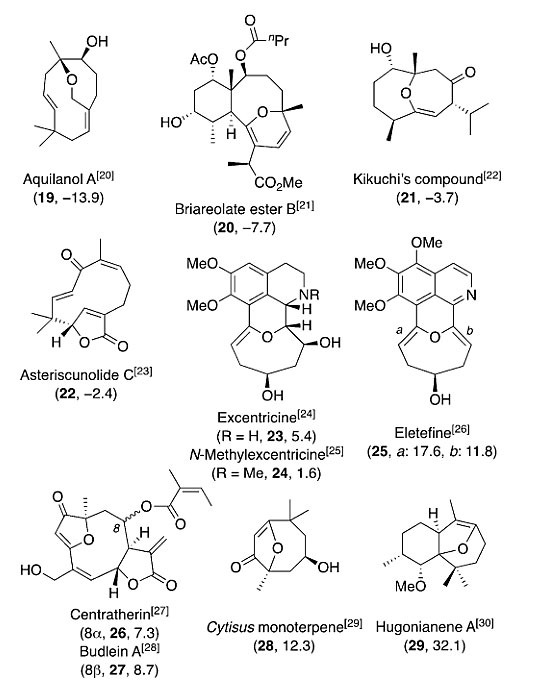
图4. 化合物19到29的结构和OSE值。图片来源:Angew. Chem. Int. Ed.
化合物29含有较高的OSE,结构极有可能解析有误。他们运用DU8+计算方法比较理论计算和实验测得的核磁化学位移和耦合常数,得出29的预测波谱数据与实验核磁数据不符,13C计算化学位移与实验值吻合度也很差,文献报道的29的结构很可能不对。
对29结构的重解析极具挑战性。最初,他们受到文献报道化合物hugonianene B(31)关联性启发,判断29可能是30的氧化加成产物,但是后者的耦合常数难以与29的实验值匹配。随后,他们探索了很多结构上的变化,如含有替代环系统的gurjunene(32)及其氧化产物33,都未能成功。回过头来分析,他们发现由于文献中的29没有高分辨质谱数据,其分子式的判断可能就不正确。且29的核磁碳信号不支持多重氧化(低场信号不足),故将其化学式修正为C16H26O(从氧原子从2减为1)。再考虑到关联化合物gurjunene的特点。他们运用DU8+分析推测,4-patchoulene-6α-ol的甲醚产物34可能是正确结构。34的计算13C化学位移和耦合常数与29吻合得很好,所以,hugonianene A(29)的结构更正为34。然而29和34的旋光值是相反的,因此34进一步更正为原结构的对映异构体。他们再用相似的研究思路去分析31,得出31的结构应该更正为35。后者在核磁波谱数据上与实验值更相吻合(图5)。
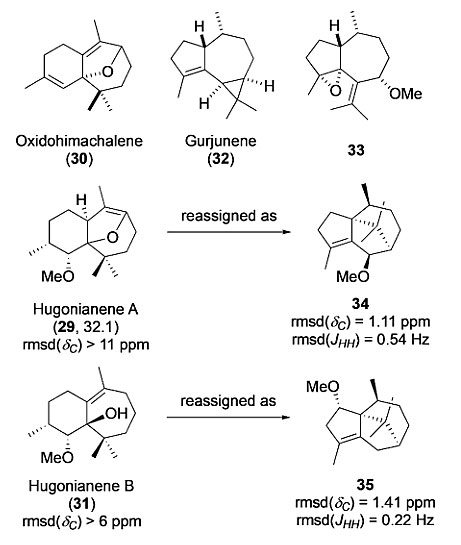
图5. 化合物29-35的结构及数据吻合度。图片来源:Angew. Chem. Int. Ed.
Andrei G. Kutateladze教授等人用DU8+回过头来计算化合物19-29,发现这些可孤立型化合物中,21和28的结构依然有错误解析的可能性。
对于化合物21,DU8+预测在7α位羟基存在H-H耦合,实验谱中却没有,如果C7取代基转为β取向,13C预测值和实验值似乎更吻合。基于上述分析,他们将21的结构更正为36,随后的生源合成理论分析也更支持后者,因为烯醇攻击E-式烯烃会派生出环氧结构(图6)。
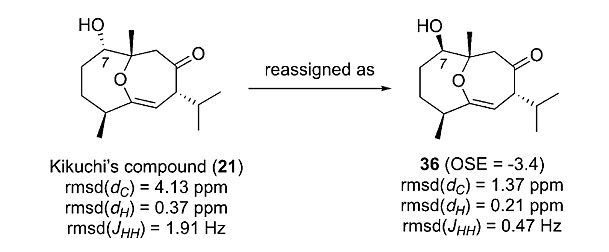
图6. 化合物21更正为36及数据吻合度。图片来源:Angew. Chem. Int. Ed.
对于Cytisus单萜28,通过生源分析发现,它可能来源于化合物chakyunglupulin A(37)或B(39),八环化合物39可能是备选结构。然而,计算化学位移与实测化学位移还是匹配得不理想。这时,28结构中一个酮基化学位移(δc 183.5 ppm)提供了重要线索,因为这个化学位移可能是α,β不饱和内酯的标志。沿着这个思路,在DU8+计算基础上,28被更正为含有五元不饱和内酯环的loliolide(40),旋光计算值判断出40的绝对构型(图 7)。
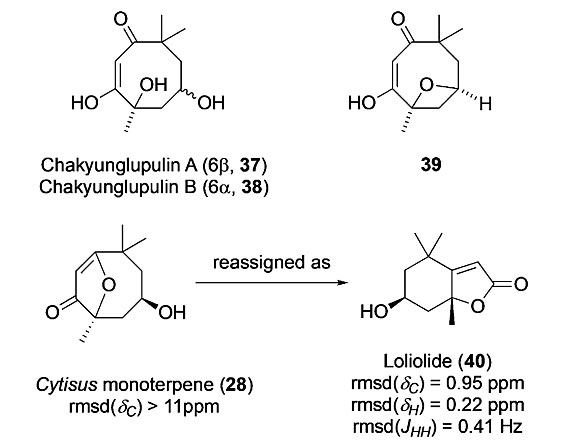
图7. 化合物28结构更正为40,及相关化合物37-38。图片来源:Angew. Chem. Int. Ed.
作者乘胜追击,利用OSE/DU8+方法更正了叶内酯folenolide(41)的结构。原结构41带有一个anti-Bredt烯,计算OSE高达39.6 kcal mol-1,数值偏高,结构可疑。结合文献报道,41所来源的植物也分离出了莽草酸化合物42。再将相关数据一比对,答案出来了:41的真正结构就是莽草酸42。解来解去,其实结构没有想象的那么复杂(图 8)。
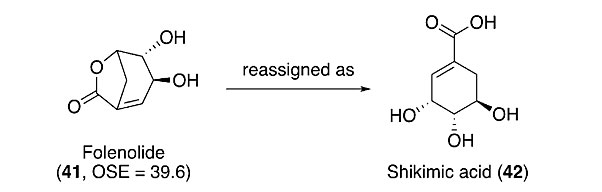
图8. 化合物41结构更正为42。图片来源:Angew. Chem. Int. Ed.
这项工作讲到这里就结束了。希望对各位坑里坑外的小伙伴们有所启发,找到适合自己的方法,早日确定正确的结构,发篇好文章,从此走上人生巅峰。
原文(扫描或长按二维码,识别后直达原文页面,或点此查看原文):

Reassignments and Corroborations of Oxo-Bridged Natural Products Directed by OSE and DU8+ NMR Computation
Angew. Chem. Int. Ed., 2019, 58, 7107-7112, DOI: 10.1002/anie.201902777
导师介绍
Andrei G. Kutateladze
https://www.x-mol.com/university/faculty/56117
Elizabeth H. Krenske
https://www.x-mol.com/university/faculty/29249
Craig M. Williams
https://www.x-mol.com/university/faculty/29255
参考资料:
1.https://chemistry.du.edu/about/faculty-directory/andrei-kutateladze
2.https://scmb.uq.edu.au/profile/999/elizabeth-krenske
3.http://archive.scmb.uq.edu.au/homepages/williams/contact.htm
(本文由水村山郭供稿)
如果篇首注明了授权来源,任何转载需获得来源方的许可!如果篇首未特别注明出处,本文版权属于 X-MOL ( x-mol.com ), 未经许可,谢绝转载!












































 京公网安备 11010802027423号
京公网安备 11010802027423号