当前位置:
X-MOL 学术
›
Soft Matter
›
论文详情
Our official English website, www.x-mol.net, welcomes your
feedback! (Note: you will need to create a separate account there.)
Resolving the structural interactions between antimicrobial peptides and lipid membranes using small-angle scattering methods: the case of indolicidin†
Soft Matter ( IF 2.9 ) Pub Date : 2018-10-16 00:00:00 , DOI: 10.1039/c8sm01888j Josefine Eilsø Nielsen 1, 2, 3, 4 , Victoria Ariel Bjørnestad 1, 2, 3, 4 , Reidar Lund 1, 2, 3, 4
Soft Matter ( IF 2.9 ) Pub Date : 2018-10-16 00:00:00 , DOI: 10.1039/c8sm01888j Josefine Eilsø Nielsen 1, 2, 3, 4 , Victoria Ariel Bjørnestad 1, 2, 3, 4 , Reidar Lund 1, 2, 3, 4
Affiliation
|
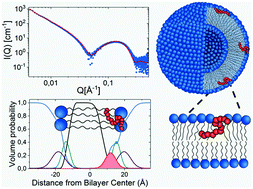
Using small angle X-ray and neutron scattering (SAXS/SANS) and detailed theoretical modelling we have elucidated the structure of the antimicrobial peptide, indolicidin, and the interaction with model lipid membranes of different anionic lipid compositions mimicking typical charge densities found in the cytoplasmic membrane of bacteria. First, we show that indolicidin displays a predominantly disordered, random chain conformation in solution with a small fraction (≈1%) of fiber-like nanostructures that are not dissolved at higher temperatures. The peptide is shown to strongly interact with the membranes at all charge densities without significantly perturbing the lipid bilayer structure. Instead, the results show that indolicidin inserts into the outer leaflet of the lipid vesicles causing a reduced local order of the lipid packing. This result is supported by an observed change in the melting point of the lipids upon addition of the peptide, as seen by differential scanning calorimetry experiments. The peptide does not to our observation affect the thickness of the membrane or form distinct structural pores in the membrane at physiologically relevant concentrations as has been previously suggested as an important mode of action. Finally, using sophisticated contrast variation SANS, we show that the peptide does not affect the random lateral distribution of anionic lipids in the membrane. Together, these results demonstrate that the structural aspects of the mode of action of antimicrobial peptides can be elucidated in detail using SAS techniques with liposomes as model systems.
中文翻译:

使用小角度散射方法解决抗菌肽和脂质膜之间的结构相互作用:吲哚美定†
使用小角度X射线和中子散射(SAXS / SANS)以及详细的理论模型,我们阐明了抗菌肽吲哚美定的结构,以及与模拟典型在细胞质中发现的典型电荷密度的不同阴离子脂质组成的模型脂质膜之间的相互作用细菌膜。首先,我们表明吲哚美定在溶液中显示出主要是无序的随机链构象,其中有一小部分(≈1%)的纤维状纳米结构在较高温度下不会溶解。显示该肽在所有电荷密度下均与膜强烈相互作用,而不会显着干扰脂质双层结构。取而代之的是,结果表明吲哚美定插入脂质囊泡的外部小叶中,导致脂质堆积的局部顺序降低。如差示扫描量热法实验所观察到的,该结果得到添加肽后脂质熔点的观察到的变化的支持。我们以前观察到,该肽不会影响膜的厚度或在生理上相关的浓度下在膜中形成明显的结构孔,如先前已提出的重要作用方式。最后,使用复杂的对比度变化SANS,我们显示该肽不会影响阴离子脂质在膜中的随机侧向分布。在一起,这些结果表明,使用脂质体作为模型系统的SAS技术,可以详细阐明抗菌肽作用方式的结构方面。如差示扫描量热法实验所见。我们以前观察到,该肽不会影响膜的厚度或在生理上相关的浓度下在膜中形成明显的结构孔,如先前已提出的重要作用方式。最后,使用复杂的对比变化SANS,我们显示该肽不会影响阴离子脂质在膜中的随机侧向分布。在一起,这些结果表明,使用脂质体作为模型系统的SAS技术,可以详细阐明抗菌肽作用方式的结构方面。如差示扫描量热法实验所见。我们以前观察到,该肽不会影响膜的厚度或在生理上相关的浓度下在膜中形成明显的结构孔,如先前已提出的重要作用方式。最后,使用复杂的对比变化SANS,我们显示该肽不会影响阴离子脂质在膜中的随机侧向分布。在一起,这些结果表明,使用脂质体作为模型系统的SAS技术,可以详细阐明抗菌肽作用方式的结构方面。我们以前观察到,该肽不会影响膜的厚度或在生理上相关的浓度下在膜中形成明显的结构孔,如先前已提出的重要作用方式。最后,使用复杂的对比变化SANS,我们显示该肽不会影响阴离子脂质在膜中的随机侧向分布。在一起,这些结果表明,使用脂质体作为模型系统的SAS技术,可以详细阐明抗菌肽作用方式的结构方面。我们以前观察到,该肽不会影响膜的厚度或在生理上相关的浓度下在膜中形成明显的结构孔,如先前已提出的重要作用方式。最后,使用复杂的对比变化SANS,我们显示该肽不会影响阴离子脂质在膜中的随机侧向分布。在一起,这些结果表明,使用脂质体作为模型系统的SAS技术,可以详细阐明抗菌肽作用方式的结构方面。
更新日期:2018-10-16
中文翻译:

使用小角度散射方法解决抗菌肽和脂质膜之间的结构相互作用:吲哚美定†
使用小角度X射线和中子散射(SAXS / SANS)以及详细的理论模型,我们阐明了抗菌肽吲哚美定的结构,以及与模拟典型在细胞质中发现的典型电荷密度的不同阴离子脂质组成的模型脂质膜之间的相互作用细菌膜。首先,我们表明吲哚美定在溶液中显示出主要是无序的随机链构象,其中有一小部分(≈1%)的纤维状纳米结构在较高温度下不会溶解。显示该肽在所有电荷密度下均与膜强烈相互作用,而不会显着干扰脂质双层结构。取而代之的是,结果表明吲哚美定插入脂质囊泡的外部小叶中,导致脂质堆积的局部顺序降低。如差示扫描量热法实验所观察到的,该结果得到添加肽后脂质熔点的观察到的变化的支持。我们以前观察到,该肽不会影响膜的厚度或在生理上相关的浓度下在膜中形成明显的结构孔,如先前已提出的重要作用方式。最后,使用复杂的对比度变化SANS,我们显示该肽不会影响阴离子脂质在膜中的随机侧向分布。在一起,这些结果表明,使用脂质体作为模型系统的SAS技术,可以详细阐明抗菌肽作用方式的结构方面。如差示扫描量热法实验所见。我们以前观察到,该肽不会影响膜的厚度或在生理上相关的浓度下在膜中形成明显的结构孔,如先前已提出的重要作用方式。最后,使用复杂的对比变化SANS,我们显示该肽不会影响阴离子脂质在膜中的随机侧向分布。在一起,这些结果表明,使用脂质体作为模型系统的SAS技术,可以详细阐明抗菌肽作用方式的结构方面。如差示扫描量热法实验所见。我们以前观察到,该肽不会影响膜的厚度或在生理上相关的浓度下在膜中形成明显的结构孔,如先前已提出的重要作用方式。最后,使用复杂的对比变化SANS,我们显示该肽不会影响阴离子脂质在膜中的随机侧向分布。在一起,这些结果表明,使用脂质体作为模型系统的SAS技术,可以详细阐明抗菌肽作用方式的结构方面。我们以前观察到,该肽不会影响膜的厚度或在生理上相关的浓度下在膜中形成明显的结构孔,如先前已提出的重要作用方式。最后,使用复杂的对比变化SANS,我们显示该肽不会影响阴离子脂质在膜中的随机侧向分布。在一起,这些结果表明,使用脂质体作为模型系统的SAS技术,可以详细阐明抗菌肽作用方式的结构方面。我们以前观察到,该肽不会影响膜的厚度或在生理上相关的浓度下在膜中形成明显的结构孔,如先前已提出的重要作用方式。最后,使用复杂的对比变化SANS,我们显示该肽不会影响阴离子脂质在膜中的随机侧向分布。在一起,这些结果表明,使用脂质体作为模型系统的SAS技术,可以详细阐明抗菌肽作用方式的结构方面。











































 京公网安备 11010802027423号
京公网安备 11010802027423号