当前位置:
X-MOL 学术
›
J. Proteome Res.
›
论文详情
Our official English website, www.x-mol.net, welcomes your feedback! (Note: you will need to create a separate account there.)
A Case Study and Methodology for OpenSWATH Parameter Optimization Using the ProCan90 Data Set and 45 810 Computational Analysis Runs.
Journal of Proteome Research ( IF 4.4 ) Pub Date : 2019-01-30 , DOI: 10.1021/acs.jproteome.8b00709 Sean Peters 1 , Peter G Hains 1, 2 , Natasha Lucas 1 , Phillip J Robinson 1 , Brett Tully 1
Journal of Proteome Research ( IF 4.4 ) Pub Date : 2019-01-30 , DOI: 10.1021/acs.jproteome.8b00709 Sean Peters 1 , Peter G Hains 1, 2 , Natasha Lucas 1 , Phillip J Robinson 1 , Brett Tully 1
Affiliation
|
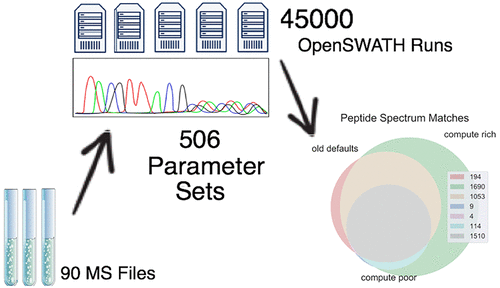
In the current study, we show how ProCan90, a curated data set of HEK293 technical replicates, can be used to optimize the configuration options for algorithms in the OpenSWATH pipeline. Furthermore, we use this case study as a proof of concept for horizontal scaling of such a pipeline to allow 45 810 computational analysis runs of OpenSWATH to be completed within four and a half days on a budget of US $10 000. Through the use of Amazon Web Services (AWS), we have successfully processed each of the ProCan 90 files with 506 combinations of input parameters. In total, the project consumed more than 340 000 core hours of compute and generated in excess of 26 TB of data. Using the resulting data and a set of quantitative metrics, we show an analysis pathway that allows the calculation of two optimal parameter sets, one for a compute rich environment (where run time is not a constraint), and another for a compute poor environment (where run time is optimized). For the same input files and the compute rich parameter set, we show a 29.8% improvement in the number of quality protein (>2 peptide) identifications found compared to the current OpenSWATH defaults, with negligible adverse effects on quantification reproducibility or drop in identification confidence, and a median run time of 75 min (103% increase). For the compute poor parameter set, we find a 55% improvement in the run time from the default parameter set, at the expense of a 3.4% decrease in the number of quality protein identifications, and an intensity CV decrease from 14.0% to 13.7%.
中文翻译:

使用ProCan90数据集和45 810计算分析运行进行OpenSWATH参数优化的案例研究和方法。
在当前的研究中,我们展示了如何将ProCan90(HEK293技术复制的精选数据集)用于优化OpenSWATH管道中算法的配置选项。此外,我们使用此案例研究作为这种管道的水平扩展的概念验证,以允许在4天半的时间内完成OpenWATH的45 810个计算分析运行,预算为10,000美元。通过使用Amazon Web服务(AWS),我们已经成功处理了506个输入参数组合的每个ProCan 90文件。总体而言,该项目消耗了超过340 000核心小时的计算时间,并生成了超过26 TB的数据。使用所得数据和一组定量指标,我们展示了一种分析路径,可以计算两个最佳参数集,一个用于计算丰富的环境(运行时间不受限制),另一个用于计算较差的环境(运行时间已优化)。对于相同的输入文件和计算丰富的参数集,与当前的OpenSWATH默认设置相比,我们发现发现的高质量蛋白质(> 2个肽段)鉴定数量提高了29.8%,对定量重现性的不利影响可忽略不计,或者降低了鉴定可信度,并且平均运行时间为75分钟(增加了103%)。对于计算较差的参数集,我们发现运行时间比默认参数集提高了55%,但代价是质量蛋白质鉴定的数量减少了3.4%,强度CV从14.0%降低到13.7% 。对于相同的输入文件和计算丰富的参数集,与当前的OpenSWATH默认设置相比,我们发现发现的高质量蛋白质(> 2个肽段)鉴定数量提高了29.8%,对定量重现性的不利影响可忽略不计,或者降低了鉴定可信度,并且平均运行时间为75分钟(增加了103%)。对于计算较差的参数集,我们发现运行时间比默认参数集提高了55%,但代价是质量蛋白质鉴定的数量减少了3.4%,强度CV从14.0%降低到13.7% 。对于相同的输入文件和计算丰富的参数集,与当前的OpenSWATH默认设置相比,我们发现发现的高质量蛋白质(> 2个肽段)鉴定数量提高了29.8%,对定量重现性的不利影响可忽略不计,或者降低了鉴定可信度,并且平均运行时间为75分钟(增加了103%)。对于计算较差的参数集,我们发现运行时间比默认参数集提高了55%,但代价是质量蛋白质鉴定的数量减少了3.4%,强度CV从14.0%降低到13.7% 。对定量重现性的不利影响或鉴定置信度的下降可忽略不计,并且平均运行时间为75分钟(增加103%)。对于计算较差的参数集,我们发现运行时间比默认参数集提高了55%,但代价是质量蛋白质鉴定的数量减少了3.4%,强度CV从14.0%降低到13.7% 。对定量重现性的不利影响或鉴定置信度的下降可忽略不计,并且平均运行时间为75分钟(增加103%)。对于计算较差的参数集,我们发现运行时间比默认参数集提高了55%,但代价是质量蛋白质鉴定的数量减少了3.4%,强度CV从14.0%降低到13.7% 。
更新日期:2019-02-07
中文翻译:

使用ProCan90数据集和45 810计算分析运行进行OpenSWATH参数优化的案例研究和方法。
在当前的研究中,我们展示了如何将ProCan90(HEK293技术复制的精选数据集)用于优化OpenSWATH管道中算法的配置选项。此外,我们使用此案例研究作为这种管道的水平扩展的概念验证,以允许在4天半的时间内完成OpenWATH的45 810个计算分析运行,预算为10,000美元。通过使用Amazon Web服务(AWS),我们已经成功处理了506个输入参数组合的每个ProCan 90文件。总体而言,该项目消耗了超过340 000核心小时的计算时间,并生成了超过26 TB的数据。使用所得数据和一组定量指标,我们展示了一种分析路径,可以计算两个最佳参数集,一个用于计算丰富的环境(运行时间不受限制),另一个用于计算较差的环境(运行时间已优化)。对于相同的输入文件和计算丰富的参数集,与当前的OpenSWATH默认设置相比,我们发现发现的高质量蛋白质(> 2个肽段)鉴定数量提高了29.8%,对定量重现性的不利影响可忽略不计,或者降低了鉴定可信度,并且平均运行时间为75分钟(增加了103%)。对于计算较差的参数集,我们发现运行时间比默认参数集提高了55%,但代价是质量蛋白质鉴定的数量减少了3.4%,强度CV从14.0%降低到13.7% 。对于相同的输入文件和计算丰富的参数集,与当前的OpenSWATH默认设置相比,我们发现发现的高质量蛋白质(> 2个肽段)鉴定数量提高了29.8%,对定量重现性的不利影响可忽略不计,或者降低了鉴定可信度,并且平均运行时间为75分钟(增加了103%)。对于计算较差的参数集,我们发现运行时间比默认参数集提高了55%,但代价是质量蛋白质鉴定的数量减少了3.4%,强度CV从14.0%降低到13.7% 。对于相同的输入文件和计算丰富的参数集,与当前的OpenSWATH默认设置相比,我们发现发现的高质量蛋白质(> 2个肽段)鉴定数量提高了29.8%,对定量重现性的不利影响可忽略不计,或者降低了鉴定可信度,并且平均运行时间为75分钟(增加了103%)。对于计算较差的参数集,我们发现运行时间比默认参数集提高了55%,但代价是质量蛋白质鉴定的数量减少了3.4%,强度CV从14.0%降低到13.7% 。对定量重现性的不利影响或鉴定置信度的下降可忽略不计,并且平均运行时间为75分钟(增加103%)。对于计算较差的参数集,我们发现运行时间比默认参数集提高了55%,但代价是质量蛋白质鉴定的数量减少了3.4%,强度CV从14.0%降低到13.7% 。对定量重现性的不利影响或鉴定置信度的下降可忽略不计,并且平均运行时间为75分钟(增加103%)。对于计算较差的参数集,我们发现运行时间比默认参数集提高了55%,但代价是质量蛋白质鉴定的数量减少了3.4%,强度CV从14.0%降低到13.7% 。



























 京公网安备 11010802027423号
京公网安备 11010802027423号