American Journal of Human Genetics ( IF 9.8 ) Pub Date : 2022-04-12 , DOI: 10.1016/j.ajhg.2022.03.016 Caoqi Fan 1 , Nicholas Mancuso 2 , Charleston W K Chiang 1
|
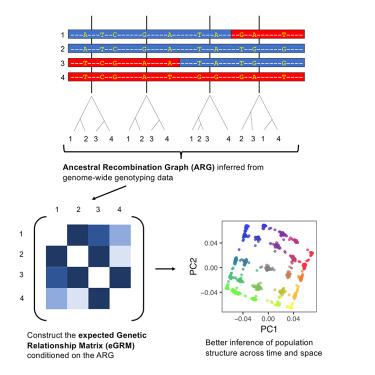
The application of genetic relationships among individuals, characterized by a genetic relationship matrix (GRM), has far-reaching effects in human genetics. However, the current standard to calculate the GRM treats linked markers as independent and does not explicitly model the underlying genealogical history of the study sample. Here, we propose a coalescent-informed framework, namely the expected GRM (eGRM), to infer the expected relatedness between pairs of individuals given an ancestral recombination graph (ARG) of the sample. Through extensive simulations, we show that the eGRM is an unbiased estimate of latent pairwise genome-wide relatedness and is robust when computed with ARG inferred from incomplete genetic data. As a result, the eGRM better captures the structure of a population than the canonical GRM, even when using the same genetic information. More importantly, our framework allows a principled approach to estimate the eGRM at different time depths of the ARG, thereby revealing the time-varying nature of population structure in a sample. When applied to SNP array genotypes from a population sample from Northern and Eastern Finland, we find that clustering analysis with the eGRM reveals population structure driven by subpopulations that would not be apparent via the canonical GRM and that temporally the population model is consistent with recent divergence and expansion. Taken together, our proposed eGRM provides a robust tree-centric estimate of relatedness with wide application to genetic studies.
中文翻译:

遗传关系的系谱估计
以遗传关系矩阵(GRM)为特征的个体间遗传关系的应用在人类遗传学中具有深远的影响。然而,当前计算 GRM 的标准将关联标记视为独立的,并且没有明确模拟研究样本的潜在家谱历史。在这里,我们提出了一个合并通知框架,即预期 GRM (eGRM),以在给定样本的祖先重组图 (ARG) 的情况下推断个体对之间的预期相关性。通过广泛的模拟,我们表明 eGRM 是潜在成对全基因组相关性的无偏估计,并且在使用从不完整遗传数据推断的 ARG 计算时是稳健的。因此,eGRM 比规范 GRM 更好地捕捉人口结构,即使使用相同的遗传信息。更重要的是,我们的框架允许采用有原则的方法来估计 ARG 不同时间深度的 eGRM,从而揭示样本中人口结构的时变性质。当应用于来自芬兰北部和东部的人口样本的 SNP 阵列基因型时,我们发现使用 eGRM 的聚类分析揭示了由通过规范 GRM 不明显的亚群驱动的人口结构,并且人口模型在时间上与最近的分歧一致和扩展。总而言之,我们提出的 eGRM 提供了一个稳健的以树为中心的相关性估计,并广泛应用于遗传研究。从而揭示样本中人口结构的时变性质。当应用于来自芬兰北部和东部的人口样本的 SNP 阵列基因型时,我们发现使用 eGRM 的聚类分析揭示了由通过规范 GRM 不明显的亚群驱动的人口结构,并且人口模型在时间上与最近的分歧一致和扩展。总而言之,我们提出的 eGRM 提供了一个稳健的以树为中心的相关性估计,并广泛应用于遗传研究。从而揭示样本中人口结构的时变性质。当应用于来自芬兰北部和东部的人口样本的 SNP 阵列基因型时,我们发现使用 eGRM 的聚类分析揭示了由通过规范 GRM 不明显的亚群驱动的人口结构,并且人口模型在时间上与最近的分歧一致和扩展。总而言之,我们提出的 eGRM 提供了一个稳健的以树为中心的相关性估计,并广泛应用于遗传研究。我们发现使用 eGRM 的聚类分析揭示了由典型 GRM 不明显的亚群驱动的人口结构,并且人口模型在时间上与最近的分歧和扩张一致。总而言之,我们提出的 eGRM 提供了一个稳健的以树为中心的相关性估计,并广泛应用于遗传研究。我们发现使用 eGRM 的聚类分析揭示了由典型 GRM 不明显的亚群驱动的人口结构,并且人口模型在时间上与最近的分歧和扩张一致。总而言之,我们提出的 eGRM 提供了一个稳健的以树为中心的相关性估计,并广泛应用于遗传研究。


























 京公网安备 11010802027423号
京公网安备 11010802027423号