Computational and Structural Biotechnology Journal ( IF 6 ) Pub Date : 2021-11-23 , DOI: 10.1016/j.csbj.2021.11.021 Wenbo Wang 1 , Junlin Wang 1 , Zhaoyu Li 1 , Dong Xu 1, 2 , Yi Shang 1
|
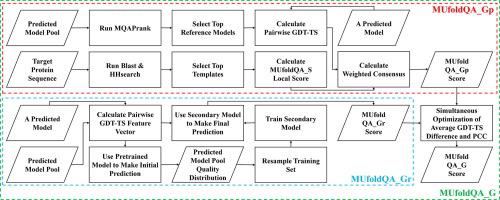
Protein tertiary structure prediction is an active research area and has attracted significant attention recently due to the success of AlphaFold from DeepMind. Methods capable of accurately evaluating the quality of predicted models are of great importance. In the past, although many model quality assessment (QA) methods have been developed, their accuracies are not consistently high across different QA performance metrics for diverse target proteins. In this paper, we propose MUfoldQA_G, a new multi-model QA method that aims at simultaneously optimizing Pearson correlation and average GDT-TS difference, two commonly used QA performance metrics. This method is based on two new algorithms MUfoldQA_Gp and MUfoldQA_Gr. MUfoldQA_Gp uses a new technique to combine information from protein templates and reference protein models to maximize the Pearson correlation QA metric. MUfoldQA_Gr employs a new machine learning technique that resamples training data and retrains adaptively to learn a consensus model that is better than naïve consensus while minimizing average GDT-TS difference. MUfoldQA_G uses a new method to combine the results of MUfoldQA_Gr and MUfoldQA_Gp so that the final QA prediction results achieve low average GDT-TS difference that is close to the results from MUfoldQA_Gr, while maintaining high Pearson correlation that is the same as the results from MUfoldQA_Gp. In CASP14 QA categories, MUfoldQA_G ranked No. 1 in Pearson correlation and No. 2 in average GDT-TS difference.
中文翻译:

MUfoldQA_G:通过再训练和转换实现的高精度蛋白质模型 QA
蛋白质三级结构预测是一个活跃的研究领域,由于 DeepMind 的 AlphaFold 的成功,最近引起了极大的关注。能够准确评估预测模型质量的方法非常重要。过去,尽管已经开发了许多模型质量评估 (QA) 方法,但它们在不同目标蛋白质的不同 QA 性能指标中的准确度并不一致。在本文中,我们提出了 MUfoldQA_G,这是一种新的多模型 QA 方法,旨在同时优化 Pearson 相关性和平均 GDT-TS 差异,这两个常用的 QA 性能指标。该方法基于两个新算法 MUfoldQA_Gp 和 MUfoldQA_Gr。MUfoldQA_Gp 使用新技术将来自蛋白质模板和参考蛋白质模型的信息结合起来,以最大化 Pearson 相关 QA 度量。MUfoldQA_Gr 采用了一种新的机器学习技术,可以对训练数据进行重新采样并自适应地重新训练,以学习一个优于原始共识的共识模型,同时最小化平均 GDT-TS 差异。MUfoldQA_G 使用新方法将 MUfoldQA_Gr 和 MUfoldQA_Gp 的结果结合起来,使最终的 QA 预测结果实现低平均 GDT-TS 差异,接近 MUfoldQA_Gr 的结果,同时保持与 MUfoldQA_Gp 的结果相同的高 Pearson 相关性. 在 CASP14 QA 类别中,MUfoldQA_G 在 Pearson 相关性中排名第一,在平均 GDT-TS 差异中排名第二。MUfoldQA_Gr 采用了一种新的机器学习技术,可以对训练数据进行重新采样并自适应地重新训练,以学习一个优于原始共识的共识模型,同时最小化平均 GDT-TS 差异。MUfoldQA_G 使用新方法将 MUfoldQA_Gr 和 MUfoldQA_Gp 的结果结合起来,使最终的 QA 预测结果实现低平均 GDT-TS 差异,接近 MUfoldQA_Gr 的结果,同时保持与 MUfoldQA_Gp 的结果相同的高 Pearson 相关性. 在 CASP14 QA 类别中,MUfoldQA_G 在 Pearson 相关性中排名第一,在平均 GDT-TS 差异中排名第二。MUfoldQA_Gr 采用了一种新的机器学习技术,可以对训练数据进行重新采样并自适应地重新训练,以学习一个优于原始共识的共识模型,同时最小化平均 GDT-TS 差异。MUfoldQA_G 使用新方法将 MUfoldQA_Gr 和 MUfoldQA_Gp 的结果结合起来,使最终的 QA 预测结果实现低平均 GDT-TS 差异,接近 MUfoldQA_Gr 的结果,同时保持与 MUfoldQA_Gp 的结果相同的高 Pearson 相关性. 在 CASP14 QA 类别中,MUfoldQA_G 在 Pearson 相关性中排名第一,在平均 GDT-TS 差异中排名第二。MUfoldQA_G 使用新方法将 MUfoldQA_Gr 和 MUfoldQA_Gp 的结果结合起来,使最终的 QA 预测结果实现低平均 GDT-TS 差异,接近 MUfoldQA_Gr 的结果,同时保持与 MUfoldQA_Gp 的结果相同的高 Pearson 相关性. 在 CASP14 QA 类别中,MUfoldQA_G 在 Pearson 相关性中排名第一,在平均 GDT-TS 差异中排名第二。MUfoldQA_G 使用新方法将 MUfoldQA_Gr 和 MUfoldQA_Gp 的结果结合起来,使最终的 QA 预测结果实现低平均 GDT-TS 差异,接近 MUfoldQA_Gr 的结果,同时保持与 MUfoldQA_Gp 的结果相同的高 Pearson 相关性. 在 CASP14 QA 类别中,MUfoldQA_G 在 Pearson 相关性中排名第一,在平均 GDT-TS 差异中排名第二。


























 京公网安备 11010802027423号
京公网安备 11010802027423号