Journal of Molecular Biology ( IF 5.6 ) Pub Date : 2021-08-08 , DOI: 10.1016/j.jmb.2021.167186 Albert Jeltsch 1 , Sabrina Adam 1 , Michael Dukatz 1 , Max Emperle 1 , Pavel Bashtrykov 1
|
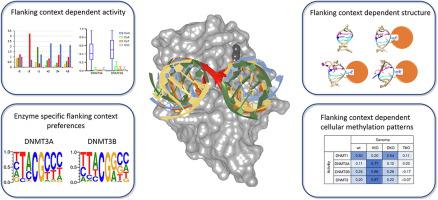
DNA interacting enzymes recognize their target sequences embedded in variable flanking sequence context. The influence of flanking sequences on enzymatic activities of DNA methyltransferases (DNMTs) can be systematically studied with “deep enzymology” approaches using pools of double-stranded DNA substrates, which contain target sites in random flanking sequence context. After incubation with DNMTs and bisulfite conversion, the methylation states and flanking sequences of individual DNA molecules are determined by NGS. Deep enzymology studies with different human and mouse DNMTs revealed strong influences of flanking sequences on their CpG and non-CpG methylation activity and the structures of DNMT-DNA complexes. Differences in flanking sequence preferences of DNMT3A and DNMT3B were shown to be related to the prominent role of DNMT3B in the methylation of human SATII repeat elements. Mutational studies in DNMT3B discovered alternative interaction networks between the enzyme and the DNA leading to a partial equalization of the effects of different flanking sequences. Structural studies in DNMT1 revealed striking correlations between enzymatic activities and flanking sequence dependent conformational changes upon DNA binding. Correlation of the biochemical data with cellular methylation patterns demonstrated that flanking sequence preferences are an important parameter that influences genomic DNA methylation patterns together with other mechanisms targeting DNMTs to genomic sites.
中文翻译:

DNA 甲基转移酶的深度酶学研究揭示了侧翼序列和酶活性之间的新联系
DNA 相互作用酶识别嵌入在可变侧翼序列上下文中的目标序列。侧翼序列对 DNA 甲基转移酶 (DNMT) 酶活性的影响可以通过“深度酶学”方法使用双链 DNA 底物池进行系统研究,其中包含随机侧翼序列上下文中的靶位点。在与 DNMT 和亚硫酸氢盐转化孵育后,单个 DNA 分子的甲基化状态和侧翼序列由 NGS 确定。对不同人类和小鼠 DNMT 的深入酶学研究揭示了侧翼序列对其 CpG 和非 CpG 甲基化活性以及 DNMT-DNA 复合物结构的强烈影响。DNMT3A 和 DNMT3B 侧翼序列偏好的差异被证明与 DNMT3B 在人 SATII 重复元件甲基化中的突出作用有关。DNMT3B 中的突变研究发现了酶和 DNA 之间的替代相互作用网络,导致不同侧翼序列的影响部分均衡。DNMT1 中的结构研究揭示了酶活性与 DNA 结合后依赖于侧翼序列的构象变化之间的惊人相关性。生化数据与细胞甲基化模式的相关性表明,侧翼序列偏好是影响基因组 DNA 甲基化模式以及将 DNMT 靶向基因组位点的其他机制的重要参数。DNMT3B 中的突变研究发现了酶和 DNA 之间的替代相互作用网络,导致不同侧翼序列的影响部分均衡。DNMT1 中的结构研究揭示了酶活性与 DNA 结合后依赖于侧翼序列的构象变化之间的惊人相关性。生化数据与细胞甲基化模式的相关性表明,侧翼序列偏好是影响基因组 DNA 甲基化模式以及将 DNMT 靶向基因组位点的其他机制的重要参数。DNMT3B 中的突变研究发现了酶和 DNA 之间的替代相互作用网络,导致不同侧翼序列的影响部分均衡。DNMT1 中的结构研究揭示了酶活性与 DNA 结合后依赖于侧翼序列的构象变化之间的显着相关性。生化数据与细胞甲基化模式的相关性表明,侧翼序列偏好是影响基因组 DNA 甲基化模式以及将 DNMT 靶向基因组位点的其他机制的重要参数。DNMT1 中的结构研究揭示了酶活性与 DNA 结合后依赖于侧翼序列的构象变化之间的惊人相关性。生化数据与细胞甲基化模式的相关性表明,侧翼序列偏好是影响基因组 DNA 甲基化模式以及将 DNMT 靶向基因组位点的其他机制的重要参数。DNMT1 中的结构研究揭示了酶活性与 DNA 结合后依赖于侧翼序列的构象变化之间的惊人相关性。生化数据与细胞甲基化模式的相关性表明,侧翼序列偏好是影响基因组 DNA 甲基化模式以及将 DNMT 靶向基因组位点的其他机制的重要参数。


























 京公网安备 11010802027423号
京公网安备 11010802027423号