Journal of Pharmaceutical Analysis ( IF 8.8 ) Pub Date : 2021-03-24 , DOI: 10.1016/j.jpha.2021.03.005 Karol Jaroch 1 , Paulina Taczyńska 1 , Marta Czechowska 1 , Joanna Bogusiewicz 1 , Kamil Łuczykowski 1 , Katarzyna Burlikowska 1 , Barbara Bojko 1
|
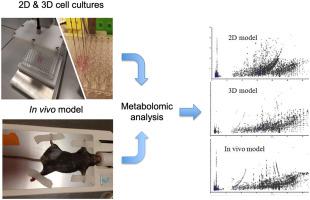
Solid phase microextraction (SPME) in combination with high-resolution mass spectrometry was employed for the determination of metabolomic profile of mouse melanoma growth within in vitro 2D, in vitro 3D, and in vivo models. Such multi-model approach had never been investigated before. Due to the low-invasiveness of SPME, it was possible to perform time-course analysis, which allowed building time profile of biochemical reactions in the studied material. Such approach does not require the multiplication of samples as subsequent analyses are performed from the very same cell culture or from the same individual. SPME already reduces the number of animals required for experiment; therefore, it is with good concordance with the 3Rs rule (replacement, reduction, and refinement). Among tested models, the largest number of compounds was found within the in vitro 2D cell culture model, while in vivo and in vitro 3D models had the lowest number of detected compounds. These results may be connected with a higher metabolic rate, as well as lower integrity of the in vitro 2D model compared to the in vitro 3D model resulting in a lower number of compounds released into medium in the latter model. In terms of in vitro-in vivo extrapolation, the in vitro 2D model performed more similar to in vivo model compared to in vitro 3D model; however, it might have been due to the fact that only compounds secreted to medium were investigated. Thus, in further experiments to obtain full metabolome information, the intraspheroidal assessment or spheroid dissociation would be necessary.
中文翻译:

一种用于体外-体内外推的提取工具?基于 SPME 的体外 2D、3D 和体内小鼠黑色素瘤模型代谢组学
固相微萃取 (SPME) 结合高分辨率质谱法用于测定体外 2D、体外 3D 和体内模型中小鼠黑色素瘤生长的代谢组学特征。以前从未研究过这种多模型方法。由于 SPME 的低侵入性,可以进行时程分析,从而可以在研究材料中建立生化反应的时间分布。这种方法不需要倍增样品,因为随后的分析是从完全相同的细胞培养物或同一个人进行的。SPME 已经减少了实验所需的动物数量;因此,它与 3Rs 规则(替换、减少和细化)具有很好的一致性。在测试的模型中,在体外 2D 细胞培养模型中发现的化合物数量最多,而在体内和体外 3D 模型中检测到的化合物数量最少。与体外 3D 模型相比,这些结果可能与更高的代谢率以及体外 2D 模型的完整性较低有关,导致后者模型中释放到培养基中的化合物数量较少。在体外-体内外推方面,与体外3D模型相比,体外2D模型的表现更类似于体内模型;然而,这可能是因为只研究了分泌到培养基中的化合物。因此,在获得完整代谢组信息的进一步实验中,需要进行球体内评估或球体解离。而体内和体外 3D 模型检测到的化合物数量最少。与体外 3D 模型相比,这些结果可能与更高的代谢率以及体外 2D 模型的完整性较低有关,导致后者模型中释放到培养基中的化合物数量较少。在体外-体内外推方面,与体外3D模型相比,体外2D模型的表现更接近体内模型;然而,这可能是因为只研究了分泌到培养基中的化合物。因此,在获得完整代谢组信息的进一步实验中,需要进行球体内评估或球体解离。而体内和体外 3D 模型检测到的化合物数量最少。与体外 3D 模型相比,这些结果可能与更高的代谢率以及体外 2D 模型的完整性较低有关,导致后者模型中释放到培养基中的化合物数量较少。在体外-体内外推方面,与体外3D模型相比,体外2D模型的表现更类似于体内模型;然而,这可能是因为只研究了分泌到培养基中的化合物。因此,在获得完整代谢组信息的进一步实验中,需要进行球体内评估或球体解离。以及与体外 3D 模型相比,体外 2D 模型的完整性较低,导致后者模型中释放到培养基中的化合物数量较少。在体外-体内外推方面,与体外3D模型相比,体外2D模型的表现更类似于体内模型;然而,这可能是因为只研究了分泌到培养基中的化合物。因此,在获得完整代谢组信息的进一步实验中,需要进行球体内评估或球体解离。以及与体外 3D 模型相比,体外 2D 模型的完整性较低,导致后者模型中释放到培养基中的化合物数量较少。在体外-体内外推方面,与体外3D模型相比,体外2D模型的表现更接近体内模型;然而,这可能是因为只研究了分泌到培养基中的化合物。因此,在获得完整代谢组信息的进一步实验中,需要进行球体内评估或球体解离。这可能是因为只研究了分泌到培养基中的化合物。因此,在获得完整代谢组信息的进一步实验中,需要进行球体内评估或球体解离。这可能是因为只研究了分泌到培养基中的化合物。因此,在获得完整代谢组信息的进一步实验中,需要进行球体内评估或球体解离。



























 京公网安备 11010802027423号
京公网安备 11010802027423号