Chaos, Solitons & Fractals ( IF 7.8 ) Pub Date : 2021-01-16 , DOI: 10.1016/j.chaos.2021.110649 Qian Zhou , Saibing Qi , Cong Ren
|
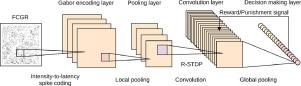
Chaos game representation (CGR) is a useful one-to-one visualization tool to represent nucleotide sequences, in which both local and global patterns of nucleotides can be graphically described. Deep learning networks have been proved to achieve outstanding performance on feature extraction and image recognition. In this paper, we use convolutional spiking neural networks (SNNs) with reward-modulated spike-timing-dependent plasticity (R-STDP) learning rule to learn from the frequency matrix chaos game representation (FCGR) images of essential and non-essential genes of 32 bacteria in the DEG database and make intra-organism and cross-organism essential gene predictions. For intra-organism predictions, our highest accuracy(ACC) score is 0.90 and the average ACC is 0.78, and for cross-organism predictions, our highest ACC is 0.79 and the average ACC is 0.68. Compared with the results of traditional machine learning classifiers training with FCGR images or numerical fractal features pre-calculated from CGR representations, our intra-organism prediction results are much better for all the bacteria or most bacteria, respectively, indicating that our spiking neural networks can make better essential gene prediction by extracting the gene features directly from the FCGR images of essential and nonessential genes. Compared with essential gene prediction methods using gene sequence features and topological features, our cross-organism prediction results can achieve performance close to or even better than such methods, while requiring much fewer input features.
中文翻译:

基于混沌博弈表示和尖峰神经网络的基因本质预测
混沌游戏表示法(CGR)是一种有用的一对一可视化工具,用于表示核苷酸序列,其中可以图形化地描述核苷酸的局部和全局模式。事实证明,深度学习网络在特征提取和图像识别方面具有出色的性能。在本文中,我们将卷积加标神经网络(SNN)与奖励调制的依赖尖峰时序的可塑性(R-STDP)学习规则结合使用,以从必不可少的基因的频率矩阵混沌博弈表示(FCGR)图像中学习对DEG数据库中的32种细菌进行分析,并做出生物体内和跨生物必需的基因预测。对于有机体内的预测,我们的最高准确度(ACC)得分为0.90,平均ACC为0.78;对于跨生物体的预测,我们的最高ACC得分为0。79,平均ACC为0.68。与使用CGR表示预先计算的使用FCGR图像或数字分形特征训练的传统机器学习分类器的结果相比,我们对所有细菌或大多数细菌的生物体内预测结果要好得多,这表明我们的尖峰神经网络可以通过直接从必需基因和非必需基因的FCGR图像中提取基因特征,可以更好地预测必需基因。与使用基因序列特征和拓扑特征的基本基因预测方法相比,我们的跨生物体预测结果可以实现接近或什至优于此类方法的性能,同时所需的输入特征要少得多。与使用CGR表示预先计算的使用FCGR图像或数字分形特征训练的传统机器学习分类器的结果相比,我们对所有细菌或大多数细菌的生物体内预测结果要好得多,这表明我们的尖峰神经网络可以通过直接从必需基因和非必需基因的FCGR图像中提取基因特征,可以更好地预测必需基因。与使用基因序列特征和拓扑特征的基本基因预测方法相比,我们的跨生物体预测结果可以实现接近或什至优于此类方法的性能,同时所需的输入特征要少得多。与使用CGR表示预先计算的使用FCGR图像或数字分形特征训练的传统机器学习分类器的结果相比,我们对所有细菌或大多数细菌的生物体内预测结果要好得多,这表明我们的尖峰神经网络可以通过直接从必需基因和非必需基因的FCGR图像中提取基因特征,可以更好地预测必需基因。与使用基因序列特征和拓扑特征的基本基因预测方法相比,我们的跨生物体预测结果可以实现接近或什至优于此类方法的性能,同时所需的输入特征要少得多。分别表明,通过直接从必需基因和非必需基因的FCGR图像中提取基因特征,我们的尖峰神经网络可以更好地预测必需基因。与使用基因序列特征和拓扑特征的基本基因预测方法相比,我们的跨有机体预测结果可以实现接近或什至优于此类方法的性能,同时所需的输入特征要少得多。分别表明,通过直接从必需基因和非必需基因的FCGR图像中提取基因特征,我们的尖峰神经网络可以更好地预测必需基因。与使用基因序列特征和拓扑特征的基本基因预测方法相比,我们的跨生物体预测结果可以实现接近或什至优于此类方法的性能,同时所需的输入特征要少得多。


























 京公网安备 11010802027423号
京公网安备 11010802027423号