当前位置:
X-MOL 学术
›
J. Raman Spectrosc.
›
论文详情
Our official English website, www.x-mol.net, welcomes your feedback! (Note: you will need to create a separate account there.)
Automated weak signal extraction of hyperspectral Raman imaging data by adaptive low‐rank matrix approximation
Journal of Raman Spectroscopy ( IF 2.5 ) Pub Date : 2020-11-02 , DOI: 10.1002/jrs.6024 Hao He 1 , Chun Lin 1 , Cheng Zong 2 , Mengxi Xu 2 , Peng Zheng 1 , Ruiqian Ye 1 , Lei Wang 1 , Bin Ren 2
Journal of Raman Spectroscopy ( IF 2.5 ) Pub Date : 2020-11-02 , DOI: 10.1002/jrs.6024 Hao He 1 , Chun Lin 1 , Cheng Zong 2 , Mengxi Xu 2 , Peng Zheng 1 , Ruiqian Ye 1 , Lei Wang 1 , Bin Ren 2
Affiliation
|
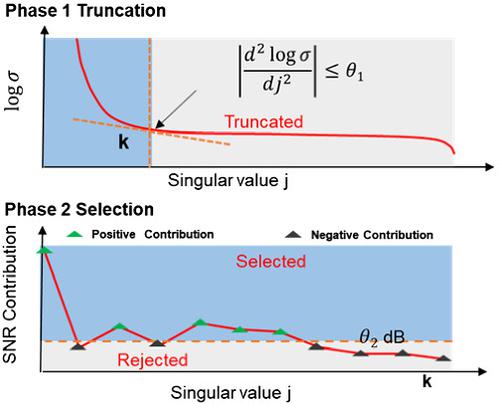
Hyperspectral Raman imaging has emerged as a promising spectroscopic tool that can provide spatial and molecular information of the sample in a label‐free and noninvasive manner, which is very suitable to the biological and biomedical research. However, the intrinsically weak Raman scattering effect results in the low signal quality of the measured Raman spectra, which has largely limited the application of Raman imaging. In this paper, we develop an adaptive low‐rank matrix approximation method to automatically extract the signal from the noisy hyperspectral Raman imaging data. After spike removal, the hyperspectral Raman imaging data are decomposed into a linear combination of submatrices by singular value decomposition. Next, the submatrices are classified into positive and negative groups according to the SNR contribution. The negative group, reflecting the instrumental noise, is discarded, and the positive group is used to reconstruct the denoised signal. We prove on the simulated data that this algorithm can significantly decrease the normalized mean noise error from 34.35% down to 1.9%. Such a strong denoising performance enables it to efficiently extract the signal from the noisy hyperspectral Raman imaging data, especially under the low SNR condition. We finally apply this algorithm to the fast speed Raman imaging of HeLa cell and surprisingly find that the slight difference of the spectra can be differentiated after signal extraction. This algorithm offers a promising tool in the Raman imaging and also can be extended to other spectroscopic imaging platforms such as fluorescent and IR spectroscopy.
中文翻译:

自适应低秩矩阵逼近自动提取高光谱拉曼成像数据的弱信号
高光谱拉曼成像已经成为一种有前途的光谱学工具,它可以以无标签且无创的方式提供样品的空间和分子信息,非常适合生物学和生物医学研究。然而,本质上较弱的拉曼散射效应导致所测量的拉曼光谱的信号质量低,这在很大程度上限制了拉曼成像的应用。在本文中,我们开发了一种自适应的低秩矩阵逼近方法,可以从嘈杂的高光谱拉曼成像数据中自动提取信号。在去除尖峰之后,通过奇异值分解将高光谱拉曼成像数据分解为子矩阵的线性组合。接下来,根据SNR贡献将子矩阵分为正组和负组。负面团体 反映了工具噪声的信号被丢弃,正组被用于重建去噪的信号。我们在仿真数据上证明了该算法可以将归一化平均噪声误差从34.35%降低到1.9%。如此强大的降噪性能使其能够有效地从嘈杂的高光谱拉曼成像数据中提取信号,尤其是在低SNR条件下。我们最终将该算法应用于HeLa细胞的快速拉曼成像,令人惊讶地发现,信号提取后可以区分出光谱的细微差别。该算法在拉曼成像中提供了有前途的工具,还可以扩展到其他光谱成像平台,例如荧光和红外光谱。正组用于重建去噪信号。我们在仿真数据上证明了该算法可以将归一化平均噪声误差从34.35%降低到1.9%。如此强大的降噪性能使其能够有效地从嘈杂的高光谱拉曼成像数据中提取信号,尤其是在低SNR条件下。我们最终将该算法应用于HeLa细胞的快速拉曼成像,令人惊讶地发现,信号提取后可以区分出光谱的细微差别。该算法在拉曼成像中提供了有前途的工具,还可以扩展到其他光谱成像平台,例如荧光和红外光谱。正组用于重建去噪信号。我们在仿真数据上证明了该算法可以将归一化平均噪声误差从34.35%降低到1.9%。如此强大的降噪性能使其能够有效地从嘈杂的高光谱拉曼成像数据中提取信号,尤其是在低SNR条件下。我们最终将该算法应用于HeLa细胞的快速拉曼成像,令人惊讶地发现,信号提取后可以区分出光谱的细微差别。该算法在拉曼成像中提供了有前途的工具,还可以扩展到其他光谱成像平台,例如荧光和红外光谱。我们在仿真数据上证明了该算法可以将归一化平均噪声误差从34.35%降低到1.9%。如此强大的降噪性能使其能够有效地从嘈杂的高光谱拉曼成像数据中提取信号,尤其是在低SNR条件下。我们最终将该算法应用于HeLa细胞的快速拉曼成像,令人惊讶地发现,信号提取后可以区分出光谱的细微差别。该算法在拉曼成像中提供了有前途的工具,还可以扩展到其他光谱成像平台,例如荧光和红外光谱。我们在仿真数据上证明了该算法可以将归一化平均噪声误差从34.35%降低到1.9%。如此强大的降噪性能使其能够有效地从嘈杂的高光谱拉曼成像数据中提取信号,尤其是在低SNR条件下。我们最终将该算法应用于HeLa细胞的快速拉曼成像,令人惊讶地发现,信号提取后可以区分出光谱的细微差别。该算法在拉曼成像中提供了有前途的工具,还可以扩展到其他光谱成像平台,例如荧光和红外光谱。特别是在低SNR条件下。我们最终将该算法应用于HeLa细胞的快速拉曼成像,令人惊讶地发现,信号提取后可以区分出光谱的细微差别。该算法在拉曼成像中提供了有前途的工具,还可以扩展到其他光谱成像平台,例如荧光和红外光谱。特别是在低SNR条件下。最后,我们将该算法应用于HeLa细胞的快速拉曼成像,令人惊讶地发现,信号提取后可以区分出光谱的细微差别。该算法在拉曼成像中提供了有前途的工具,还可以扩展到其他光谱成像平台,例如荧光和红外光谱。
更新日期:2020-12-10
中文翻译:

自适应低秩矩阵逼近自动提取高光谱拉曼成像数据的弱信号
高光谱拉曼成像已经成为一种有前途的光谱学工具,它可以以无标签且无创的方式提供样品的空间和分子信息,非常适合生物学和生物医学研究。然而,本质上较弱的拉曼散射效应导致所测量的拉曼光谱的信号质量低,这在很大程度上限制了拉曼成像的应用。在本文中,我们开发了一种自适应的低秩矩阵逼近方法,可以从嘈杂的高光谱拉曼成像数据中自动提取信号。在去除尖峰之后,通过奇异值分解将高光谱拉曼成像数据分解为子矩阵的线性组合。接下来,根据SNR贡献将子矩阵分为正组和负组。负面团体 反映了工具噪声的信号被丢弃,正组被用于重建去噪的信号。我们在仿真数据上证明了该算法可以将归一化平均噪声误差从34.35%降低到1.9%。如此强大的降噪性能使其能够有效地从嘈杂的高光谱拉曼成像数据中提取信号,尤其是在低SNR条件下。我们最终将该算法应用于HeLa细胞的快速拉曼成像,令人惊讶地发现,信号提取后可以区分出光谱的细微差别。该算法在拉曼成像中提供了有前途的工具,还可以扩展到其他光谱成像平台,例如荧光和红外光谱。正组用于重建去噪信号。我们在仿真数据上证明了该算法可以将归一化平均噪声误差从34.35%降低到1.9%。如此强大的降噪性能使其能够有效地从嘈杂的高光谱拉曼成像数据中提取信号,尤其是在低SNR条件下。我们最终将该算法应用于HeLa细胞的快速拉曼成像,令人惊讶地发现,信号提取后可以区分出光谱的细微差别。该算法在拉曼成像中提供了有前途的工具,还可以扩展到其他光谱成像平台,例如荧光和红外光谱。正组用于重建去噪信号。我们在仿真数据上证明了该算法可以将归一化平均噪声误差从34.35%降低到1.9%。如此强大的降噪性能使其能够有效地从嘈杂的高光谱拉曼成像数据中提取信号,尤其是在低SNR条件下。我们最终将该算法应用于HeLa细胞的快速拉曼成像,令人惊讶地发现,信号提取后可以区分出光谱的细微差别。该算法在拉曼成像中提供了有前途的工具,还可以扩展到其他光谱成像平台,例如荧光和红外光谱。我们在仿真数据上证明了该算法可以将归一化平均噪声误差从34.35%降低到1.9%。如此强大的降噪性能使其能够有效地从嘈杂的高光谱拉曼成像数据中提取信号,尤其是在低SNR条件下。我们最终将该算法应用于HeLa细胞的快速拉曼成像,令人惊讶地发现,信号提取后可以区分出光谱的细微差别。该算法在拉曼成像中提供了有前途的工具,还可以扩展到其他光谱成像平台,例如荧光和红外光谱。我们在仿真数据上证明了该算法可以将归一化平均噪声误差从34.35%降低到1.9%。如此强大的降噪性能使其能够有效地从嘈杂的高光谱拉曼成像数据中提取信号,尤其是在低SNR条件下。我们最终将该算法应用于HeLa细胞的快速拉曼成像,令人惊讶地发现,信号提取后可以区分出光谱的细微差别。该算法在拉曼成像中提供了有前途的工具,还可以扩展到其他光谱成像平台,例如荧光和红外光谱。特别是在低SNR条件下。我们最终将该算法应用于HeLa细胞的快速拉曼成像,令人惊讶地发现,信号提取后可以区分出光谱的细微差别。该算法在拉曼成像中提供了有前途的工具,还可以扩展到其他光谱成像平台,例如荧光和红外光谱。特别是在低SNR条件下。最后,我们将该算法应用于HeLa细胞的快速拉曼成像,令人惊讶地发现,信号提取后可以区分出光谱的细微差别。该算法在拉曼成像中提供了有前途的工具,还可以扩展到其他光谱成像平台,例如荧光和红外光谱。


























 京公网安备 11010802027423号
京公网安备 11010802027423号