当前位置:
X-MOL 学术
›
Mol. Omics
›
论文详情
Our official English website, www.x-mol.net, welcomes your feedback! (Note: you will need to create a separate account there.)
Guide for protein fold change and p-value calculation for non-experts in proteomics.
Molecular Omics ( IF 2.9 ) Pub Date : 2020-09-18 , DOI: 10.1039/d0mo00087f Jennifer T Aguilan 1 , Katarzyna Kulej , Simone Sidoli
Molecular Omics ( IF 2.9 ) Pub Date : 2020-09-18 , DOI: 10.1039/d0mo00087f Jennifer T Aguilan 1 , Katarzyna Kulej , Simone Sidoli
Affiliation
|
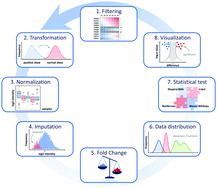
Proteomics studies generate tables with thousands of entries. A significant component of being a proteomics scientist is the ability to process these tables to identify regulated proteins. Many bioinformatics tools are freely available for the community, some of which within reach for scientists with limited or no background in programming and statistics. However, proteomics has become popular in most other biological and biomedical disciplines, resulting in more and more studies where data processing is delegated to specialists that are not lead authors of the scientific project. This creates a risk or at least a limiting factor, as the biological interpretation of a dataset is contingent of a third-party specialist transforming data without the input of the project leader. We acknowledge in advance that dedicated scripts and software have a higher level of sophistication; but we hereby claim that the approach we describe makes proteomics data processing immediately accessible to every scientist. In this paper, we describe key steps of the typical data transformation, normalization and statistics in proteomics data analysis using a simple spreadsheet. This manuscript aims to demonstrate to those who are not familiar with the math and statistics behind these workflows that a proteomics dataset can be processed, simplified and interpreted in software like Microsoft Excel. With this, we aim to reach the community of non-specialists in proteomics to find a common language and illustrate the basic steps of –omics data processing.
中文翻译:

非蛋白质组学专家的蛋白质倍数变化和 p 值计算指南。
蛋白质组学研究生成包含数千个条目的表格。成为蛋白质组学科学家的一个重要组成部分是处理这些表格以识别受调控蛋白质的能力。许多生物信息学工具可供社区免费使用,其中一些可供编程和统计背景有限或没有背景的科学家使用。然而,蛋白质组学在大多数其他生物和生物医学学科中变得流行,导致越来越多的研究将数据处理委托给不是科学项目主要作者的专家。这会产生风险或至少是一个限制因素,因为数据集的生物学解释取决于第三方专家在没有项目负责人输入的情况下转换数据。我们事先承认专用脚本和软件具有更高的复杂程度;但我们在此声明,我们描述的方法使每个科学家都可以立即进行蛋白质组学数据处理。在本文中,我们使用简单的电子表格描述了蛋白质组学数据分析中典型数据转换、归一化和统计的关键步骤。这份手稿旨在向那些不熟悉这些工作流程背后的数学和统计学的人证明,可以在 Microsoft Excel 等软件中处理、简化和解释蛋白质组学数据集。有了这个,我们的目标是接触蛋白质组学的非专家社区,以找到一种通用语言并说明组学数据处理的基本步骤。但我们在此声明,我们描述的方法使每个科学家都可以立即进行蛋白质组学数据处理。在本文中,我们使用简单的电子表格描述了蛋白质组学数据分析中典型数据转换、归一化和统计的关键步骤。这份手稿旨在向那些不熟悉这些工作流程背后的数学和统计学的人证明,可以在 Microsoft Excel 等软件中处理、简化和解释蛋白质组学数据集。有了这个,我们的目标是接触蛋白质组学的非专家社区,以找到一种通用语言并说明组学数据处理的基本步骤。但我们在此声明,我们描述的方法使每个科学家都可以立即进行蛋白质组学数据处理。在本文中,我们使用简单的电子表格描述了蛋白质组学数据分析中典型数据转换、归一化和统计的关键步骤。这份手稿旨在向那些不熟悉这些工作流程背后的数学和统计学的人证明,可以在 Microsoft Excel 等软件中处理、简化和解释蛋白质组学数据集。有了这个,我们的目标是接触蛋白质组学的非专家社区,以找到一种通用语言并说明组学数据处理的基本步骤。使用简单电子表格进行蛋白质组学数据分析中的标准化和统计。这份手稿旨在向那些不熟悉这些工作流程背后的数学和统计学的人证明,可以在 Microsoft Excel 等软件中处理、简化和解释蛋白质组学数据集。有了这个,我们的目标是接触蛋白质组学的非专家社区,以找到一种通用语言并说明组学数据处理的基本步骤。使用简单电子表格进行蛋白质组学数据分析中的标准化和统计。这份手稿旨在向那些不熟悉这些工作流程背后的数学和统计学的人证明,可以在 Microsoft Excel 等软件中处理、简化和解释蛋白质组学数据集。有了这个,我们的目标是接触蛋白质组学的非专家社区,以找到一种通用语言并说明组学数据处理的基本步骤。
更新日期:2020-11-03
中文翻译:

非蛋白质组学专家的蛋白质倍数变化和 p 值计算指南。
蛋白质组学研究生成包含数千个条目的表格。成为蛋白质组学科学家的一个重要组成部分是处理这些表格以识别受调控蛋白质的能力。许多生物信息学工具可供社区免费使用,其中一些可供编程和统计背景有限或没有背景的科学家使用。然而,蛋白质组学在大多数其他生物和生物医学学科中变得流行,导致越来越多的研究将数据处理委托给不是科学项目主要作者的专家。这会产生风险或至少是一个限制因素,因为数据集的生物学解释取决于第三方专家在没有项目负责人输入的情况下转换数据。我们事先承认专用脚本和软件具有更高的复杂程度;但我们在此声明,我们描述的方法使每个科学家都可以立即进行蛋白质组学数据处理。在本文中,我们使用简单的电子表格描述了蛋白质组学数据分析中典型数据转换、归一化和统计的关键步骤。这份手稿旨在向那些不熟悉这些工作流程背后的数学和统计学的人证明,可以在 Microsoft Excel 等软件中处理、简化和解释蛋白质组学数据集。有了这个,我们的目标是接触蛋白质组学的非专家社区,以找到一种通用语言并说明组学数据处理的基本步骤。但我们在此声明,我们描述的方法使每个科学家都可以立即进行蛋白质组学数据处理。在本文中,我们使用简单的电子表格描述了蛋白质组学数据分析中典型数据转换、归一化和统计的关键步骤。这份手稿旨在向那些不熟悉这些工作流程背后的数学和统计学的人证明,可以在 Microsoft Excel 等软件中处理、简化和解释蛋白质组学数据集。有了这个,我们的目标是接触蛋白质组学的非专家社区,以找到一种通用语言并说明组学数据处理的基本步骤。但我们在此声明,我们描述的方法使每个科学家都可以立即进行蛋白质组学数据处理。在本文中,我们使用简单的电子表格描述了蛋白质组学数据分析中典型数据转换、归一化和统计的关键步骤。这份手稿旨在向那些不熟悉这些工作流程背后的数学和统计学的人证明,可以在 Microsoft Excel 等软件中处理、简化和解释蛋白质组学数据集。有了这个,我们的目标是接触蛋白质组学的非专家社区,以找到一种通用语言并说明组学数据处理的基本步骤。使用简单电子表格进行蛋白质组学数据分析中的标准化和统计。这份手稿旨在向那些不熟悉这些工作流程背后的数学和统计学的人证明,可以在 Microsoft Excel 等软件中处理、简化和解释蛋白质组学数据集。有了这个,我们的目标是接触蛋白质组学的非专家社区,以找到一种通用语言并说明组学数据处理的基本步骤。使用简单电子表格进行蛋白质组学数据分析中的标准化和统计。这份手稿旨在向那些不熟悉这些工作流程背后的数学和统计学的人证明,可以在 Microsoft Excel 等软件中处理、简化和解释蛋白质组学数据集。有了这个,我们的目标是接触蛋白质组学的非专家社区,以找到一种通用语言并说明组学数据处理的基本步骤。

























 京公网安备 11010802027423号
京公网安备 11010802027423号