当前位置:
X-MOL 学术
›
BBA Proteins Proteom.
›
论文详情
Our official English website, www.x-mol.net, welcomes your feedback! (Note: you will need to create a separate account there.)
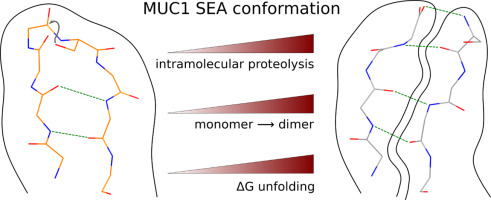
High-resolution structure of intramolecularly proteolyzed human mucin-1 SEA domain.
Biochimica et Biophysica Acta (BBA) - Proteins and Proteomics ( IF 3.2 ) Pub Date : 2020-01-07 , DOI: 10.1016/j.bbapap.2020.140361 Martín E Noguera 1 , Jean Jakoncic 2 , Mario R Ermácora 3
Biochimica et Biophysica Acta (BBA) - Proteins and Proteomics ( IF 3.2 ) Pub Date : 2020-01-07 , DOI: 10.1016/j.bbapap.2020.140361 Martín E Noguera 1 , Jean Jakoncic 2 , Mario R Ermácora 3
Affiliation
|
SEA domains are ubiquitous in large proteins associated with highly glycosylated environments. Certain SEA domains undergo intramolecular proteolysis involving a nucleophilic attack of a serine hydroxyl group on the preceding glycine carbonyl. The mucin-1 (MUC1) SEA domain has been extensively investigated as a model of intramolecular proteolysis. Since neither a general base, a general acid, nor an oxyanion hole could be identified in MUC1 SEA, it has been suggested that proteolysis is accelerated by a non-planarity of the scissile peptide bond imposed by protein folding. A reactant distorted peptide bond has been also invoked to explain the autoproteolysis of several unrelated proteins. However, the only evidence of peptide distortion in MUC1 SEA stems from molecular dynamic simulations of the reactant modeled upon a single NMR structure of the cleaved product. We report the first high-resolution X-ray structure of cleaved MUC1 SEA. Structural comparison with uncleaved SEA domains suggests that the number of residues evolutionarily inserted in the cleaved loop of MUC1 SEA precludes the formation of a properly hydrogen-bonded beta turn. By sequence analysis, we show that this conformational frustration is shared by all known cleaved SEA domains. In addition, alternative conformations of the uncleaved precursor could be modeled in which the scissile peptide bond is planar. The implications of these structures for autoproteolysis are discussed in the light of the previous research on autoproteolysis.
中文翻译:

分子内蛋白水解的人类粘蛋白1 SEA域的高分辨率结构。
SEA结构域在与高度糖基化环境相关的大型蛋白质中无处不在。某些SEA结构域经历分子内蛋白水解,涉及到先前甘氨酸羰基上丝氨酸羟基的亲核攻击。mucin-1(MUC1)SEA域已被广泛研究为分子内蛋白水解的模型。由于无法在MUC1 SEA中识别出通用碱,通用酸或氧阴离子孔,因此,有人提出,蛋白质折叠所引起的易裂肽键的非平面性会加速蛋白水解。还调用了反应物扭曲的肽键来解释几种无关蛋白的自蛋白水解作用。然而,MUC1 SEA中肽失真的唯一证据来自基于裂解产物的单个NMR结构建模的反应物的分子动力学模拟。我们报告分裂的MUC1 SEA的第一个高分辨率X射线结构。与未切割的SEA结构域的结构比较表明,进化插入MUC1 SEA的切割环中的残基数量无法形成适当的氢键合β匝。通过序列分析,我们表明这种构象挫折是所有已知的裂解SEA域所共有的。另外,可以对未裂解的前体的替代构象进行建模,其中易裂肽键是平面的。这些结构对自蛋白水解的影响是根据先前对自蛋白水解的研究进行讨论的。我们报告分裂的MUC1 SEA的第一个高分辨率X射线结构。与未切割的SEA结构域的结构比较表明,进化插入MUC1 SEA的切割环中的残基数量无法形成适当的氢键合β匝。通过序列分析,我们表明这种构象挫折是所有已知的裂解SEA域所共有的。另外,可以对未裂解的前体的替代构象进行建模,其中易裂肽键是平面的。这些结构对自蛋白水解的影响是根据先前对自蛋白水解的研究进行讨论的。我们报告分裂的MUC1 SEA的第一个高分辨率X射线结构。与未切割的SEA结构域的结构比较表明,进化插入MUC1 SEA的切割环中的残基数量无法形成适当的氢键合β匝。通过序列分析,我们表明这种构象挫折是所有已知的裂解SEA域所共有的。另外,可以对未裂解的前体的替代构象进行建模,其中易裂肽键是平面的。这些结构对自蛋白水解的影响是根据先前对自蛋白水解的研究进行讨论的。与未切割的SEA结构域的结构比较表明,进化插入MUC1 SEA的切割环中的残基数量无法形成适当的氢键合β匝。通过序列分析,我们表明这种构象挫折是所有已知的裂解SEA域所共有的。另外,可以对未裂解的前体的替代构象进行建模,其中易裂肽键是平面的。这些结构对自蛋白水解的影响是根据先前对自蛋白水解的研究进行讨论的。与未切割的SEA结构域的结构比较表明,进化插入MUC1 SEA的切割环中的残基数量无法形成适当的氢键合β环。通过序列分析,我们表明这种构象挫折是所有已知的裂解SEA域所共有的。另外,可以对未裂解的前体的替代构象进行建模,其中易裂肽键是平面的。这些结构对自蛋白水解的影响是根据先前对自蛋白水解的研究进行讨论的。
更新日期:2020-01-07
中文翻译:

分子内蛋白水解的人类粘蛋白1 SEA域的高分辨率结构。
SEA结构域在与高度糖基化环境相关的大型蛋白质中无处不在。某些SEA结构域经历分子内蛋白水解,涉及到先前甘氨酸羰基上丝氨酸羟基的亲核攻击。mucin-1(MUC1)SEA域已被广泛研究为分子内蛋白水解的模型。由于无法在MUC1 SEA中识别出通用碱,通用酸或氧阴离子孔,因此,有人提出,蛋白质折叠所引起的易裂肽键的非平面性会加速蛋白水解。还调用了反应物扭曲的肽键来解释几种无关蛋白的自蛋白水解作用。然而,MUC1 SEA中肽失真的唯一证据来自基于裂解产物的单个NMR结构建模的反应物的分子动力学模拟。我们报告分裂的MUC1 SEA的第一个高分辨率X射线结构。与未切割的SEA结构域的结构比较表明,进化插入MUC1 SEA的切割环中的残基数量无法形成适当的氢键合β匝。通过序列分析,我们表明这种构象挫折是所有已知的裂解SEA域所共有的。另外,可以对未裂解的前体的替代构象进行建模,其中易裂肽键是平面的。这些结构对自蛋白水解的影响是根据先前对自蛋白水解的研究进行讨论的。我们报告分裂的MUC1 SEA的第一个高分辨率X射线结构。与未切割的SEA结构域的结构比较表明,进化插入MUC1 SEA的切割环中的残基数量无法形成适当的氢键合β匝。通过序列分析,我们表明这种构象挫折是所有已知的裂解SEA域所共有的。另外,可以对未裂解的前体的替代构象进行建模,其中易裂肽键是平面的。这些结构对自蛋白水解的影响是根据先前对自蛋白水解的研究进行讨论的。我们报告分裂的MUC1 SEA的第一个高分辨率X射线结构。与未切割的SEA结构域的结构比较表明,进化插入MUC1 SEA的切割环中的残基数量无法形成适当的氢键合β匝。通过序列分析,我们表明这种构象挫折是所有已知的裂解SEA域所共有的。另外,可以对未裂解的前体的替代构象进行建模,其中易裂肽键是平面的。这些结构对自蛋白水解的影响是根据先前对自蛋白水解的研究进行讨论的。与未切割的SEA结构域的结构比较表明,进化插入MUC1 SEA的切割环中的残基数量无法形成适当的氢键合β匝。通过序列分析,我们表明这种构象挫折是所有已知的裂解SEA域所共有的。另外,可以对未裂解的前体的替代构象进行建模,其中易裂肽键是平面的。这些结构对自蛋白水解的影响是根据先前对自蛋白水解的研究进行讨论的。与未切割的SEA结构域的结构比较表明,进化插入MUC1 SEA的切割环中的残基数量无法形成适当的氢键合β环。通过序列分析,我们表明这种构象挫折是所有已知的裂解SEA域所共有的。另外,可以对未裂解的前体的替代构象进行建模,其中易裂肽键是平面的。这些结构对自蛋白水解的影响是根据先前对自蛋白水解的研究进行讨论的。



























 京公网安备 11010802027423号
京公网安备 11010802027423号