营养中的“暗物质”:你了解自己在吃什么吗? BMC Bioinformatics
论文标题:PhyteByte: identification of foods containing compounds with specific pharmacological properties
作者:Kenneth E. Westerman, Sean Harrington et.al
发表时间:2020/06/10
数字识别码:10.1186/s12859-020-03582-7
微信链接:点击此处阅读微信文章
原文作者:Dr. Larry Parnell
食物中的植物化学物质和其他分子可以给人体健康带来很多好处,但它们具体是什么,通过什么机制工作呢?由于存在无法分配给已知分子的光谱数据——营养中的“暗物质”,人们对上述问题仍知之甚少。Parnell及其同事使用一种名为PhyteByte的计算预测工具对这种暗物质进行了研究,其结果现已发表在开放获取期刊BMC Bioinformatics上。

图1
想象一下,吃一碗当季的草莓,这是一种美味、健康的零食。深红的色泽和浆果的香气惹人怜爱,那是因为长春新碱3-葡萄糖苷(1)和甲氧基呋喃醇(2)吸引着人们的感官。这些浆果中也含有γ-癸内酯(3)。事实上,这种水果中还天然存在着数百种其他的生化化合物。对于其他食物,如姜黄、生姜、大蒜、莴苣、巧克力、咖啡甚至葡萄酒,情况也基本类似。
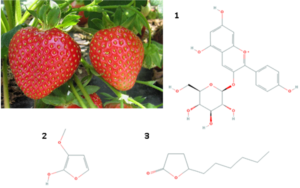
图2
在草莓中发现的一些化合物包括天竺葵素-3-氯化葡萄糖苷葡萄糖苷(1,PubChem ID 443648)、甲氧基呋喃醇(2,53929577)和γ-葵内酯(3,12813)。
但这些化合物能产生哪些健康效应呢?当然,答案很多,因为任何一种食物中的化合物目录都很长而且种类繁多,通过实验的方式测试每一种化合物并不现实。然而,通过将严格的计算方法用于探索营养学的"暗物质",可以获得一些答案。
营养学中的暗物质
在代谢组学中,这种"暗物质"是指大量的光谱数据,这些数据不容易分配给已知的分子。至于与营养学有关的,最近我们从两个方面探索了暗物质的概念。首先,努力扩大存在于生的、发酵的、加工的、储存的和代谢的食物中的化合物目录。有了这个基础,第二步探索性的工作自然是确定每种化合物的生物学功能。为了在第二步中取得进展,我们选择使用具有良好特征的药物化合物作为预测食品化合物的基础。具体来说,我们提出了这样一个问题:“我们是否可以通过识别结构相似的药物来预测一种化合物的生物学功能?”于是,便开始了PhyteByte的设计和编码。
PhyteByte的功能
PhyteByte是一款设计用于预测食物化合物生物效应的软件。实现方法很简单:终端用户输入一个蛋白质靶标,PhyteByte能识别与靶标相互作用可能性相对较高的食物化合物,从而预测生物学效应。PhyteByte利用随机森林分类器来完成大部分繁重的工作。该模型经过训练,可以从ChEMBL等数据库中识别药物化合物。化合物被编码为“指纹”,即存储复杂化学信息学子结构信息的简单位向量。我们的模型学习给定靶标的所需子结构,并将其泛化以识别我们测试数据集中的新型食品化合物。
重要的是,PhyteByte是一种预测工具。它输出的是一份极有可能产生特定生物学效应的分子及其来源食物的列表,但它并非是设计膳食计划或提供饮食建议的工具。
计算营养学的未来之路
PhyteByte只是在基因组学、代谢组学和遗传学数据的基础上进行营养研究的新计算方法(如复杂系统理论和机器学习)的众多例子之一。事实上,我们可以说,计算营养学只是营养学与组学(omics)结合的自然产物。
计算营养学可能会以几种方式发展。其一,给定一种感兴趣的表型或蛋白质,提出可能影响该表型或蛋白质的食物,并以靶向、无宏量营养素的方式完成。其二,更详细的流行病学和食物频率问卷调查数据可以支持某些食物和健康结局之间联系的试验。在这里,所研究的食物包含的化合物,根据与已知药物的相似性,可能提供狭义的健康获益。其三,这种计算得出的药理学-食物化合物联系可以为药物开发提供信息。例如,药物疗效研究可以指导志愿者避免食用含有可能表现出与药物一样的生理效应的化合物的特定食物。
重要的是,虽然上述一些科学努力已经融入到营养研究中有一段时间了,但这里所描述的所有内容都需要在更大的范围内作出努力。而且,为了使这些方法最有效,必须进行广泛的协调努力,以确定和准确量化每种食物中大约2000种不同的化合物,这是新兴的食物组学领域的一个核心主题。
BMC Bioinformatics is an open access, peer-reviewed journal that considers articles on all aspects of the development, testing and novel application of computational and statistical methods for the modeling and analysis of all kinds of biological data, as well as other areas of computational biology.
Citation Impact
3.242 - 2-year IF
3.213 - 5-year IF
1.156 - SNIP
1.626 - SJR
摘要:
Background
Phytochemicals and other molecules in foods elicit positive health benefits, often by poorly established or unknown mechanisms. While there is a wealth of data on the biological and biophysical properties of drugs and therapeutic compounds, there is a notable lack of similar data for compounds commonly present in food. Computational methods for high-throughput identification of food compounds with specific biological effects, especially when accompanied by relevant food composition data, could enable more effective and more personalized dietary planning. We have created a machine learning-based tool (PhyteByte) to leverage existing pharmacological data to predict bioactivity across a comprehensive molecular database of foods and food compounds.
Results
PhyteByte uses a cheminformatic approach to structure-based activity prediction and applies it to uncover the putative bioactivity of food compounds. The tool takes an input protein target and develops a random forest classifier to predict the effect of an input molecule based on its molecular fingerprint, using structure and activity data available from the ChEMBL database. It then predicts the relevant bioactivity of a library of food compounds with known molecular structures from the FooDB database. The output is a list of food compounds with high confidence of eliciting relevant biological effects, along with their source foods and associated quantities in those foods, where available. Applying PhyteByte to the humanPPARGgene, we identified irigenin, sesamin, fargesin, and delta-sanshool as putative agonists of PPARG, along with previously identified agonists of this important metabolic regulator.
Conclusions
PhyteByte identifies food-based compounds that are predicted to interact with specific protein targets. The identified relationships can be used to prioritize food compounds for experimental or epidemiological follow-up and can contribute to the rapid development of precision approaches to new nutraceuticals as well as personalized dietary planning.
如果篇首注明了授权来源,任何转载需获得来源方的许可!如果篇首未特别注明出处,本文版权属于 X-MOL ( x-mol.com ), 未经许可,谢绝转载!




























 京公网安备 11010802027423号
京公网安备 11010802027423号