计算化学生物学助力更强大基因编辑工具
CRISPR/Cas系统是存在于细菌和古生菌中抵御病毒的一种获得性免疫系统。近来利用CRISPR/Cas系统靶向识别并切割基因组的特性,CRISPR/Cas系统被发展成为一种可以广泛应用与多物种的基因编辑工具。一种在金黄色葡萄链球菌(Staphylococcus aureus)中发现的新型Cas9(SaCas9),具备了体积更小的优势,使其更利于递送及应用于基因治疗领域。今年刚刚被FDA批准的首个人类体内(In vivo)基因治疗的临床试验中,张锋教授等基因编辑专家共同创立的Editas Medicine就是使用这一SaCas9编辑系统,发挥其体内药物递送的优势,完成了首例遗传性失明基因编辑临床研究的设计及申报。
但是相比于更广泛应用的SpCas9在基因组上识别的NGG PAM序列,SaCas9识别一段更长的NNGRRT PAM序列,这很大程度限制了SaCas9在基因组上的识别范围。这样的PAM序列局限性也促进了对于SaCas9蛋白的工程化改造相关工作的开展,例如一种识别更短的NNNRRT PAM序列的SaCas9变体 SaCas9-KKH的发现。
近期发表在Journal of the American Chemical Society期刊上的一项研究中,IBM周如鸿研究组及斯坦福大学丛乐课题组结合基于蛋白质结构数据的分子动力学模拟、自由能扰动分析(FEP)以及生物实验技术三个跨学科的技术手段,建立了COMET基因编辑优化平台(Combined Molecular dynamics and Experimental Target validation,COMET)。这是一套针对CRISPR/Cas 系统基因编辑能力的计算预测与实验评估方法学(图1)。并利用此套方法分析了通过蛋白质进化手段获得的SaCas9-KKH对于PAM识别的分子机制,以此验证了COMET方法的可靠性。并且又进一步运用此技术平台获得了不同于SaCas9-KKH PAM识别模式的另外两种突变体:SaCas9-NR和SaCas9-RL(图2)。实验表明,这两种突变体对于野生型SaCas9 识别的PAM序列NNGRRT中第五位的T有明显减弱的偏好性。

图1. COMET工作流程总结
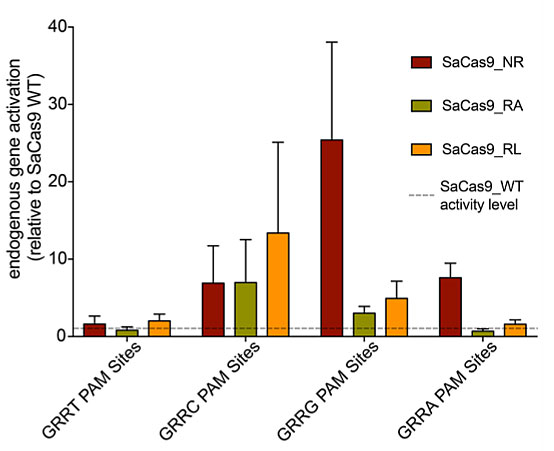
图2. 利用COMET获得的SaCas9突变体以及野生型SaCas9对于内源基因的切割效率比较。
此研究验证了其提出的计算和实验相结合的方法学COMET的可靠性,并且利用这套方法获得了可以识别更宽松PAM序列的SaCas9突变体。研究人员认为,这套方法学可以为今后进一步理解CRISPR/Cas分子作用机制和开发具备新特性的Cas蛋白突变体提供重要理论基础以及技术手段,实现了计算与生物技术的跨学科、跨平台结合,而且还可以应用于其他新出现的基因编辑工具的开发。
原文(扫描或长按二维码,识别后直达原文页面,或点此查看原文):

Combined Computational–Experimental Approach to Explore the Molecular Mechanism of SaCas9 with a Broadened DNA Targeting Range
Binquan Luan, Guangxue Xu, Mei Feng, Le Cong, Ruhong Zhou
J. Am. Chem. Soc., 2019, 141, 6545-6552, DOI: 10.1021/jacs.8b13144
目前,斯坦福大学丛乐实验室和IBM周如鸿研究组在开展进一步合作,运用计算模拟结合基因编辑实验,联合开发RNA编辑,精确多位点改造等新的编辑与治疗技术,团队正在招募新成员加入,如果你对这一新兴方向的博士研究生机会,或博士后职位感兴趣,欢迎发邮件咨询丛乐教授(congle@stanford.edu),并参考实验室网站(www.conglab.com )上的相关信息。
周如鸿
https://www.x-mol.com/university/faculty/41611
如果篇首注明了授权来源,任何转载需获得来源方的许可!如果篇首未特别注明出处,本文版权属于 X-MOL ( x-mol.com ), 未经许可,谢绝转载!




























 京公网安备 11010802027423号
京公网安备 11010802027423号