当前位置:
X-MOL 学术
›
J. Proteome Res.
›
论文详情
Our official English website, www.x-mol.net, welcomes your feedback! (Note: you will need to create a separate account there.)
KELM-CPPpred: Kernel Extreme Learning Machine Based Prediction Model for Cell-Penetrating Peptides
Journal of Proteome Research ( IF 4.4 ) Pub Date : 2018-08-13 , DOI: 10.1021/acs.jproteome.8b00322 Poonam Pandey 1 , Vinal Patel 2 , Nithin V. George 2 , Sairam S. Mallajosyula 3
Journal of Proteome Research ( IF 4.4 ) Pub Date : 2018-08-13 , DOI: 10.1021/acs.jproteome.8b00322 Poonam Pandey 1 , Vinal Patel 2 , Nithin V. George 2 , Sairam S. Mallajosyula 3
Affiliation
|
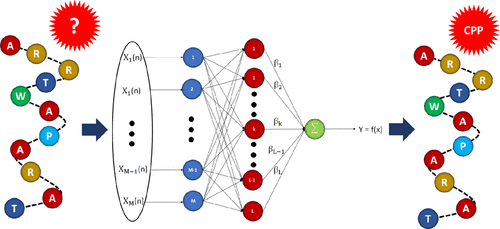
Cell-penetrating peptides (CPPs) facilitate the transport of pharmacologically active molecules, such as plasmid DNA, short interfering RNA, nanoparticles, and small peptides. The accurate identification of new and unique CPPs is the initial step to gain insight into CPP activity. Experiments can provide detailed insight into the cell-penetration property of CPPs. However, the synthesis and identification of CPPs through wet-lab experiments is both resource- and time-expensive. Therefore, the development of an efficient prediction tool is essential for the identification of unique CPP prior to experiments. To this end, we developed a kernel extreme learning machine (KELM) based CPP prediction model called KELM-CPPpred. The main data set used in this study consists of 408 CPPs and an equal number of non-CPPs. The input features, used to train the proposed prediction model, include amino acid composition, dipeptide amino acid composition, pseudo amino acid composition, and the motif-based hybrid features. We further used an independent data set to validate the proposed model. In addition, we have also tested the prediction accuracy of KELM-CPPpred models with the existing artificial neural network (ANN), random forest (RF), and support vector machine (SVM) approaches on respective benchmark data sets used in the previous studies. Empirical tests showed that KELM-CPPpred outperformed existing prediction approaches based on SVM, RF, and ANN. We developed a web interface named KELM-CPPpred, which is freely available at http://sairam.people.iitgn.ac.in/KELM-CPPpred.html
中文翻译:

KELM-CPPpred:基于核极限学习机的细胞穿透肽预测模型
细胞穿透肽(CPPs)促进了药理活性分子(如质粒DNA,短干扰RNA,纳米颗粒和小肽)的运输。准确识别新的和独特的CPP是迈进CPP活动的第一步。实验可以深入了解CPP的细胞穿透特性。但是,通过湿实验室实验合成和鉴定CPP既耗费资源又耗时。因此,开发有效的预测工具对于在实验前识别独特的CPP至关重要。为此,我们开发了一种基于内核极限学习机(KELM)的CPP预测模型,称为KELM-CPPpred。本研究中使用的主要数据集由408个CPP和相等数量的非CPP组成。输入功能 用于训练提出的预测模型的方法,包括氨基酸组成,二肽氨基酸组成,假氨基酸组成和基于基序的杂种特征。我们进一步使用了独立的数据集来验证所提出的模型。此外,我们还针对先前研究中使用的各个基准数据集,使用现有的人工神经网络(ANN),随机森林(RF)和支持向量机(SVM)方法测试了KELM-CPPpred模型的预测准确性。经验测试表明,KELM-CPPpred优于基于SVM,RF和ANN的现有预测方法。我们开发了一个名为KELM-CPPpred的Web界面,该界面可从http://sairam.people.iitgn.ac.in/KELM-CPPpred.html免费获得。假氨基酸组成,以及基于基序的杂种特征。我们进一步使用了独立的数据集来验证所提出的模型。此外,我们还使用现有的人工神经网络(ANN),随机森林(RF)和支持向量机(SVM)方法对先前研究中使用的各个基准数据集测试了KELM-CPPpred模型的预测准确性。经验测试表明,KELM-CPPpred优于基于SVM,RF和ANN的现有预测方法。我们开发了一个名为KELM-CPPpred的Web界面,该界面可从http://sairam.people.iitgn.ac.in/KELM-CPPpred.html免费获得。假氨基酸组成,以及基于基序的杂种特征。我们进一步使用了独立的数据集来验证所提出的模型。此外,我们还针对先前研究中使用的各个基准数据集,使用现有的人工神经网络(ANN),随机森林(RF)和支持向量机(SVM)方法测试了KELM-CPPpred模型的预测准确性。经验测试表明,KELM-CPPpred优于基于SVM,RF和ANN的现有预测方法。我们开发了一个名为KELM-CPPpred的Web界面,该界面可从http://sairam.people.iitgn.ac.in/KELM-CPPpred.html免费获得。我们还使用现有的人工神经网络(ANN),随机森林(RF)和支持向量机(SVM)方法对先前研究中使用的各个基准数据集测试了KELM-CPPpred模型的预测准确性。经验测试表明,KELM-CPPpred优于基于SVM,RF和ANN的现有预测方法。我们开发了一个名为KELM-CPPpred的Web界面,该界面可从http://sairam.people.iitgn.ac.in/KELM-CPPpred.html免费获得。我们还使用现有的人工神经网络(ANN),随机森林(RF)和支持向量机(SVM)方法对先前研究中使用的各个基准数据集测试了KELM-CPPpred模型的预测准确性。经验测试表明,KELM-CPPpred优于基于SVM,RF和ANN的现有预测方法。我们开发了一个名为KELM-CPPpred的Web界面,该界面可从http://sairam.people.iitgn.ac.in/KELM-CPPpred.html免费获得。
更新日期:2018-08-13
中文翻译:

KELM-CPPpred:基于核极限学习机的细胞穿透肽预测模型
细胞穿透肽(CPPs)促进了药理活性分子(如质粒DNA,短干扰RNA,纳米颗粒和小肽)的运输。准确识别新的和独特的CPP是迈进CPP活动的第一步。实验可以深入了解CPP的细胞穿透特性。但是,通过湿实验室实验合成和鉴定CPP既耗费资源又耗时。因此,开发有效的预测工具对于在实验前识别独特的CPP至关重要。为此,我们开发了一种基于内核极限学习机(KELM)的CPP预测模型,称为KELM-CPPpred。本研究中使用的主要数据集由408个CPP和相等数量的非CPP组成。输入功能 用于训练提出的预测模型的方法,包括氨基酸组成,二肽氨基酸组成,假氨基酸组成和基于基序的杂种特征。我们进一步使用了独立的数据集来验证所提出的模型。此外,我们还针对先前研究中使用的各个基准数据集,使用现有的人工神经网络(ANN),随机森林(RF)和支持向量机(SVM)方法测试了KELM-CPPpred模型的预测准确性。经验测试表明,KELM-CPPpred优于基于SVM,RF和ANN的现有预测方法。我们开发了一个名为KELM-CPPpred的Web界面,该界面可从http://sairam.people.iitgn.ac.in/KELM-CPPpred.html免费获得。假氨基酸组成,以及基于基序的杂种特征。我们进一步使用了独立的数据集来验证所提出的模型。此外,我们还使用现有的人工神经网络(ANN),随机森林(RF)和支持向量机(SVM)方法对先前研究中使用的各个基准数据集测试了KELM-CPPpred模型的预测准确性。经验测试表明,KELM-CPPpred优于基于SVM,RF和ANN的现有预测方法。我们开发了一个名为KELM-CPPpred的Web界面,该界面可从http://sairam.people.iitgn.ac.in/KELM-CPPpred.html免费获得。假氨基酸组成,以及基于基序的杂种特征。我们进一步使用了独立的数据集来验证所提出的模型。此外,我们还针对先前研究中使用的各个基准数据集,使用现有的人工神经网络(ANN),随机森林(RF)和支持向量机(SVM)方法测试了KELM-CPPpred模型的预测准确性。经验测试表明,KELM-CPPpred优于基于SVM,RF和ANN的现有预测方法。我们开发了一个名为KELM-CPPpred的Web界面,该界面可从http://sairam.people.iitgn.ac.in/KELM-CPPpred.html免费获得。我们还使用现有的人工神经网络(ANN),随机森林(RF)和支持向量机(SVM)方法对先前研究中使用的各个基准数据集测试了KELM-CPPpred模型的预测准确性。经验测试表明,KELM-CPPpred优于基于SVM,RF和ANN的现有预测方法。我们开发了一个名为KELM-CPPpred的Web界面,该界面可从http://sairam.people.iitgn.ac.in/KELM-CPPpred.html免费获得。我们还使用现有的人工神经网络(ANN),随机森林(RF)和支持向量机(SVM)方法对先前研究中使用的各个基准数据集测试了KELM-CPPpred模型的预测准确性。经验测试表明,KELM-CPPpred优于基于SVM,RF和ANN的现有预测方法。我们开发了一个名为KELM-CPPpred的Web界面,该界面可从http://sairam.people.iitgn.ac.in/KELM-CPPpred.html免费获得。

























 京公网安备 11010802027423号
京公网安备 11010802027423号