当前位置:
X-MOL 学术
›
Small Methods
›
论文详情
Our official English website, www.x-mol.net, welcomes your feedback! (Note: you will need to create a separate account there.)
Mechanical Polyprotein Assembly Using Sfp and Sortase‐Mediated Domain Oligomerization for Single‐Molecule Studies
Small Methods ( IF 12.4 ) Pub Date : 2018-05-03 , DOI: 10.1002/smtd.201800039 Haipei Liu 1, 2 , Duy Tien Ta 1, 2 , Michael A. Nash 1, 2
Small Methods ( IF 12.4 ) Pub Date : 2018-05-03 , DOI: 10.1002/smtd.201800039 Haipei Liu 1, 2 , Duy Tien Ta 1, 2 , Michael A. Nash 1, 2
Affiliation
|
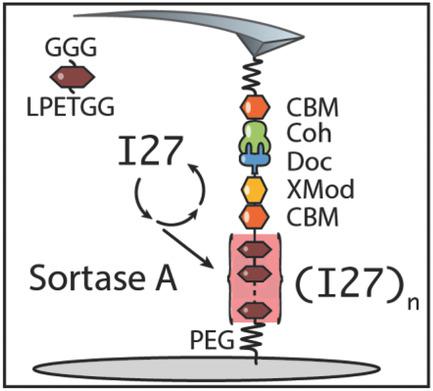
Single‐molecule force spectroscopy (SMFS) with the atomic force microscope (AFM) provides remarkable details on the energy landscapes governing protein (un)folding and intermolecular complex dissociation. In such experiments, multidomain polyproteins consisting of multiple copies of independently foldable domains provide internal controls identifiable by characteristic contour length increments, unfolding forces, and/or unfolding substeps. Here, a new approach to polyprotein synthesis is presented relying on posttranslational enzyme‐mediated oligomerization of domains. Mutant variants of immunoglobulin 27 (I27) and a bacterial cellulose binding module (CBM) fused to an Ig‐like X‐module (XMod), and a mechanostable receptor called Dockerin (Doc) are produced with complementary peptide tags. By utilizing 4′‐phosphopantetheinyl transferase and Sortase A, the system enables I27‐domain oligomerization into polyproteins of varying lengths followed by C‐terminal capping with mechanostable Doc. The number of oligomerized domains per molecule, the unfolding forces, and the complex rupture forces of posttranslationally assembled polyproteins are characterized using >40 h automated AFM–SMFS with a Cohesin (Coh)‐modified cantilever. Use of the Coh–Doc interaction to unfold polyproteins provides a high yield of ≈ 3800 specific single‐molecule interaction curves. This approach is advantageous for assembly of polyproteins from domains that lack proper folding or are insoluble in a polyprotein format.
中文翻译:

机械多蛋白组装使用Sfp和分选酶介导的域寡聚进行单分子研究
带有原子力显微镜(AFM)的单分子力谱(SMFS)提供了有关控制蛋白质(非)折叠和分子间复合物解离的能量分布的显着细节。在这样的实验中,由独立可折叠结构域的多个拷贝组成的多结构域多蛋白提供了通过特征轮廓长度增量,展开力和/或展开子步骤可识别的内部对照。在此,提出了一种新的多蛋白合成方法,该方法依赖于翻译后酶介导的域寡聚化。免疫球蛋白27(I27)和与Ig样X-module(XMod)融合的细菌纤维素结合模块(CBM)的突变体,以及称为Dockerin(Doc)的可机械接收的受体,都带有互补的肽标签。通过利用4'-磷酸泛锡基转移酶和Sortase A,该系统能够将I27域寡聚化为不同长度的多蛋白,然后使用可机械转换的Doc进行C端封端。每个分子的寡聚化结构域数量,解折叠后的多蛋白的解折叠力和复杂的断裂力可通过> 40 h带有黏着素(Coh)修饰的悬臂的自动AFM–SMFS进行表征。使用Coh-Doc相互作用来展开多蛋白可提供约3800个特定单分子相互作用曲线的高产率。这种方法对于从缺乏适当折叠或不溶于多蛋白形式的结构域组装多蛋白是有利的。每个分子的寡聚化结构域数量,解折叠后的多蛋白的解折叠力和复杂的断裂力可通过> 40 h带有黏着素(Coh)修饰的悬臂的自动AFM–SMFS进行表征。使用Coh-Doc相互作用来展开多蛋白可提供约3800个特定单分子相互作用曲线的高产率。这种方法对于从缺乏适当折叠或不溶于多蛋白形式的结构域组装多蛋白是有利的。每个分子的寡聚化结构域数量,解折叠后的多蛋白的解折叠力和复杂的断裂力可通过> 40 h带有黏着素(Coh)修饰的悬臂的自动AFM–SMFS进行表征。使用Coh-Doc相互作用来展开多蛋白可提供约3800个特定单分子相互作用曲线的高产率。该方法对于从缺乏适当折叠或不溶于多蛋白形式的结构域组装多蛋白是有利的。使用Coh-Doc相互作用来展开多蛋白可提供约3800个特定单分子相互作用曲线的高产率。该方法对于从缺乏适当折叠或不溶于多蛋白形式的结构域组装多蛋白是有利的。使用Coh-Doc相互作用来展开多蛋白可提供约3800个特定单分子相互作用曲线的高产率。这种方法对于从缺乏适当折叠或不溶于多蛋白形式的结构域组装多蛋白是有利的。
更新日期:2018-05-03
中文翻译:

机械多蛋白组装使用Sfp和分选酶介导的域寡聚进行单分子研究
带有原子力显微镜(AFM)的单分子力谱(SMFS)提供了有关控制蛋白质(非)折叠和分子间复合物解离的能量分布的显着细节。在这样的实验中,由独立可折叠结构域的多个拷贝组成的多结构域多蛋白提供了通过特征轮廓长度增量,展开力和/或展开子步骤可识别的内部对照。在此,提出了一种新的多蛋白合成方法,该方法依赖于翻译后酶介导的域寡聚化。免疫球蛋白27(I27)和与Ig样X-module(XMod)融合的细菌纤维素结合模块(CBM)的突变体,以及称为Dockerin(Doc)的可机械接收的受体,都带有互补的肽标签。通过利用4'-磷酸泛锡基转移酶和Sortase A,该系统能够将I27域寡聚化为不同长度的多蛋白,然后使用可机械转换的Doc进行C端封端。每个分子的寡聚化结构域数量,解折叠后的多蛋白的解折叠力和复杂的断裂力可通过> 40 h带有黏着素(Coh)修饰的悬臂的自动AFM–SMFS进行表征。使用Coh-Doc相互作用来展开多蛋白可提供约3800个特定单分子相互作用曲线的高产率。这种方法对于从缺乏适当折叠或不溶于多蛋白形式的结构域组装多蛋白是有利的。每个分子的寡聚化结构域数量,解折叠后的多蛋白的解折叠力和复杂的断裂力可通过> 40 h带有黏着素(Coh)修饰的悬臂的自动AFM–SMFS进行表征。使用Coh-Doc相互作用来展开多蛋白可提供约3800个特定单分子相互作用曲线的高产率。这种方法对于从缺乏适当折叠或不溶于多蛋白形式的结构域组装多蛋白是有利的。每个分子的寡聚化结构域数量,解折叠后的多蛋白的解折叠力和复杂的断裂力可通过> 40 h带有黏着素(Coh)修饰的悬臂的自动AFM–SMFS进行表征。使用Coh-Doc相互作用来展开多蛋白可提供约3800个特定单分子相互作用曲线的高产率。该方法对于从缺乏适当折叠或不溶于多蛋白形式的结构域组装多蛋白是有利的。使用Coh-Doc相互作用来展开多蛋白可提供约3800个特定单分子相互作用曲线的高产率。该方法对于从缺乏适当折叠或不溶于多蛋白形式的结构域组装多蛋白是有利的。使用Coh-Doc相互作用来展开多蛋白可提供约3800个特定单分子相互作用曲线的高产率。这种方法对于从缺乏适当折叠或不溶于多蛋白形式的结构域组装多蛋白是有利的。



























 京公网安备 11010802027423号
京公网安备 11010802027423号