当前位置:
X-MOL 学术
›
J. Chem. Inf. Model.
›
论文详情
Our official English website, www.x-mol.net, welcomes your feedback! (Note: you will need to create a separate account there.)
Development of a Reverse Phase HPLC Retention Index Model for Nontargeted Metabolomics Using Synthetic Compounds
Journal of Chemical Information and Modeling ( IF 5.6 ) Pub Date : 2018-02-28 00:00:00 , DOI: 10.1021/acs.jcim.7b00496 L Mark Hall 1 , Dennis W Hill 2 , Kelly Bugden 3 , Shannon Cawley 4 , Lowell H Hall 5 , Ming-Hui Chen 6 , David F Grant 2
Journal of Chemical Information and Modeling ( IF 5.6 ) Pub Date : 2018-02-28 00:00:00 , DOI: 10.1021/acs.jcim.7b00496 L Mark Hall 1 , Dennis W Hill 2 , Kelly Bugden 3 , Shannon Cawley 4 , Lowell H Hall 5 , Ming-Hui Chen 6 , David F Grant 2
Affiliation
|
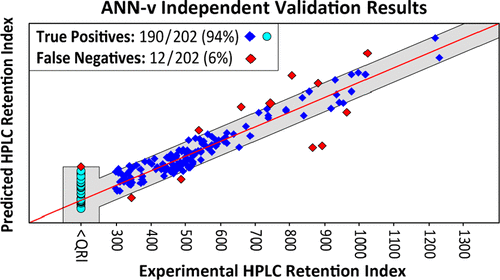
The MolFind application has been developed as a nontargeted metabolomics chemometric tool to facilitate structure identification when HPLC biofluids analysis reveals a feature of interest. Here synthetic compounds are selected and measured to form the basis of a new, more accurate, HPLC retention index model for use with MolFind. We show that relatively inexpensive synthetic screening compounds with simple structures can be used to develop an artificial neural network model that is successful in making quality predictions for human metabolites. A total of 1955 compounds were obtained and measured for the model. A separate set of 202 human metabolites was used for independent validation. The new ANN model showed improved accuracy over previous models. The model, based on relatively simple compounds, was able to make quality predictions for complex compounds not similar to training data. Independent validation metabolites with feature combinations found in three or more training compounds were predicted with 97% sensitivity while metabolites with feature combinations found in less than three training compounds were predicted with >90% sensitivity. The study describes the method used to select synthetic compounds and new descriptors developed to encode the relationship between lipophilic molecular subgraphs and HPLC retention. Finally, we introduce the QRI (qualitative range of interest) modification of neural network backpropagation learning to generate models simultaneously based on quantitative and qualitative data.
中文翻译:

使用合成化合物开发非靶向代谢组学的反相 HPLC 保留指数模型
MolFind 应用程序已开发为一种非靶向代谢组学化学计量学工具,可在 HPLC 生物流体分析揭示感兴趣的特征时促进结构鉴定。在这里选择和测量合成化合物,以形成与 MolFind 一起使用的新的、更准确的 HPLC 保留指数模型的基础。我们表明,具有简单结构的相对廉价的合成筛选化合物可用于开发人工神经网络模型,该模型成功地对人类代谢物进行质量预测。该模型共获得并测量了 1955 种化合物。一组单独的 202 种人类代谢物用于独立验证。新的 ANN 模型比以前的模型显示出更高的准确性。该模型基于相对简单的化合物,能够对与训练数据不相似的复杂化合物进行质量预测。在三个或更多训练化合物中发现的具有特征组合的独立验证代谢物的预测灵敏度为 97%,而在少于三个训练化合物中发现的具有特征组合的代谢物的预测灵敏度大于 90%。该研究描述了用于选择合成化合物的方法以及为编码亲脂性分子子图和 HPLC 保留之间的关系而开发的新描述符。最后,我们介绍了神经网络反向传播学习的 QRI(感兴趣的定性范围)修改,以基于定量和定性数据同时生成模型。在三个或更多训练化合物中发现的具有特征组合的独立验证代谢物的预测灵敏度为 97%,而在少于三个训练化合物中发现的具有特征组合的代谢物的预测灵敏度大于 90%。该研究描述了用于选择合成化合物的方法以及为编码亲脂性分子子图与 HPLC 保留之间的关系而开发的新描述符。最后,我们介绍了神经网络反向传播学习的 QRI(感兴趣的定性范围)修改,以基于定量和定性数据同时生成模型。在三个或更多训练化合物中发现的具有特征组合的独立验证代谢物的预测灵敏度为 97%,而在少于三个训练化合物中发现的具有特征组合的代谢物的预测灵敏度大于 90%。该研究描述了用于选择合成化合物的方法以及为编码亲脂性分子子图和 HPLC 保留之间的关系而开发的新描述符。最后,我们介绍了神经网络反向传播学习的 QRI(感兴趣的定性范围)修改,以基于定量和定性数据同时生成模型。该研究描述了用于选择合成化合物的方法以及为编码亲脂性分子子图与 HPLC 保留之间的关系而开发的新描述符。最后,我们介绍了神经网络反向传播学习的 QRI(感兴趣的定性范围)修改,以基于定量和定性数据同时生成模型。该研究描述了用于选择合成化合物的方法以及为编码亲脂性分子子图与 HPLC 保留之间的关系而开发的新描述符。最后,我们介绍了神经网络反向传播学习的 QRI(感兴趣的定性范围)修改,以基于定量和定性数据同时生成模型。
更新日期:2018-02-28
中文翻译:

使用合成化合物开发非靶向代谢组学的反相 HPLC 保留指数模型
MolFind 应用程序已开发为一种非靶向代谢组学化学计量学工具,可在 HPLC 生物流体分析揭示感兴趣的特征时促进结构鉴定。在这里选择和测量合成化合物,以形成与 MolFind 一起使用的新的、更准确的 HPLC 保留指数模型的基础。我们表明,具有简单结构的相对廉价的合成筛选化合物可用于开发人工神经网络模型,该模型成功地对人类代谢物进行质量预测。该模型共获得并测量了 1955 种化合物。一组单独的 202 种人类代谢物用于独立验证。新的 ANN 模型比以前的模型显示出更高的准确性。该模型基于相对简单的化合物,能够对与训练数据不相似的复杂化合物进行质量预测。在三个或更多训练化合物中发现的具有特征组合的独立验证代谢物的预测灵敏度为 97%,而在少于三个训练化合物中发现的具有特征组合的代谢物的预测灵敏度大于 90%。该研究描述了用于选择合成化合物的方法以及为编码亲脂性分子子图和 HPLC 保留之间的关系而开发的新描述符。最后,我们介绍了神经网络反向传播学习的 QRI(感兴趣的定性范围)修改,以基于定量和定性数据同时生成模型。在三个或更多训练化合物中发现的具有特征组合的独立验证代谢物的预测灵敏度为 97%,而在少于三个训练化合物中发现的具有特征组合的代谢物的预测灵敏度大于 90%。该研究描述了用于选择合成化合物的方法以及为编码亲脂性分子子图与 HPLC 保留之间的关系而开发的新描述符。最后,我们介绍了神经网络反向传播学习的 QRI(感兴趣的定性范围)修改,以基于定量和定性数据同时生成模型。在三个或更多训练化合物中发现的具有特征组合的独立验证代谢物的预测灵敏度为 97%,而在少于三个训练化合物中发现的具有特征组合的代谢物的预测灵敏度大于 90%。该研究描述了用于选择合成化合物的方法以及为编码亲脂性分子子图和 HPLC 保留之间的关系而开发的新描述符。最后,我们介绍了神经网络反向传播学习的 QRI(感兴趣的定性范围)修改,以基于定量和定性数据同时生成模型。该研究描述了用于选择合成化合物的方法以及为编码亲脂性分子子图与 HPLC 保留之间的关系而开发的新描述符。最后,我们介绍了神经网络反向传播学习的 QRI(感兴趣的定性范围)修改,以基于定量和定性数据同时生成模型。该研究描述了用于选择合成化合物的方法以及为编码亲脂性分子子图与 HPLC 保留之间的关系而开发的新描述符。最后,我们介绍了神经网络反向传播学习的 QRI(感兴趣的定性范围)修改,以基于定量和定性数据同时生成模型。



























 京公网安备 11010802027423号
京公网安备 11010802027423号