当前位置:
X-MOL 学术
›
ACS Catal.
›
论文详情
Our official English website, www.x-mol.net, welcomes your feedback! (Note: you will need to create a separate account there.)
Origin of Enzymatic Kinetic Isotope Effects in Human Purine Nucleoside Phosphorylase
ACS Catalysis ( IF 12.9 ) Pub Date : 2017-12-29 00:00:00 , DOI: 10.1021/acscatal.7b04199 M. Roca 1, 2 , V. Moliner 1 , I. Tuñón 2
ACS Catalysis ( IF 12.9 ) Pub Date : 2017-12-29 00:00:00 , DOI: 10.1021/acscatal.7b04199 M. Roca 1, 2 , V. Moliner 1 , I. Tuñón 2
Affiliation
|
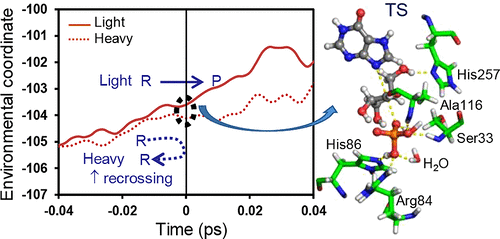
Here we report a study of the effect of heavy isotope labeling on the reaction catalyzed by human purine nucleoside phosphorylase (hPNP) to elucidate the origin of its catalytic effect and of the enzymatic kinetic isotope effect (EKIE). Using quantum mechanical and molecular mechanical (QM/MM) molecular dynamics (MD) simulations, we study the mechanism of the hPNP enzyme and the dynamic effects by means of the calculation of the recrossing transmission coefficient. A free energy surface (FES), as a function of both a chemical and an environmental coordinate, is obtained to show the role of the environment on the chemical reaction. Analysis of reactive and nonreactive trajectories allows us to study the geometric, dynamic, and electronic changes of the chemical system. Special attention is paid to the electrostatic potential created by the environment on those atoms involved in the chemical reaction. Some amino acid residues and solvent molecules that interact with the chemical system provide a specific configuration that electrostatically favor the reaction progress, producing a reactive trajectory. The EKIE is calculated within the framework of the variational transition state theory (VTST), giving very good agreement with the experimental data. According to our simulations the chemical reaction is slightly slower in the heavy enzyme than in its light counterpart enzyme because protein motions coupled to the reaction coordinate are slower. Thus, protein dynamics have a small but measurable effect on the chemical reaction rate.
中文翻译:

人嘌呤核苷磷酸化酶的酶动力学同位素效应的起源。
在这里,我们报告了重同位素标记对人嘌呤核苷磷酸化酶(hPNP)催化反应的影响的研究,以阐明其催化作用和酶动力学同位素作用(EKIE)的起源。使用量子力学和分子力学(QM / MM)分子动力学(MD)模拟,我们通过计算回交传输系数来研究hPNP酶的机理和动力学效应。获得了自由能表面(FES),它是化学和环境坐标的函数,以显示环境在化学反应中的作用。反应性和非反应性轨迹的分析使我们能够研究化学系统的几何,动态和电子变化。要特别注意环境在化学反应中所涉及的那些原子上产生的静电势。与化学系统相互作用的一些氨基酸残基和溶剂分子提供了一种特定的构型,该构型在静电上有利于反应进程,从而产生了反应性轨迹。EKIE是在变分过渡状态理论(VTST)的框架内计算的,与实验数据非常吻合。根据我们的模拟,由于与反应坐标相关的蛋白质运动较慢,重酶的化学反应比轻酶的化学反应稍慢。因此,蛋白质动力学对化学反应速率的影响很小但可测量。与化学系统相互作用的一些氨基酸残基和溶剂分子提供了一种特定的构型,该构型在静电上有利于反应进程,从而产生了反应性轨迹。EKIE是在变分过渡状态理论(VTST)的框架内计算的,与实验数据非常吻合。根据我们的模拟,由于与反应坐标相关的蛋白质运动较慢,重酶的化学反应比轻酶的化学反应稍慢。因此,蛋白质动力学对化学反应速率的影响很小但可测量。与化学系统相互作用的一些氨基酸残基和溶剂分子提供了一种特定的构型,该构型在静电上有利于反应进程,从而产生了反应性轨迹。EKIE是在变分过渡状态理论(VTST)的框架内计算的,与实验数据非常吻合。根据我们的模拟,由于与反应坐标相关的蛋白质运动较慢,重酶的化学反应比轻酶的化学反应稍慢。因此,蛋白质动力学对化学反应速率的影响很小但可测量。EKIE是在变分过渡状态理论(VTST)的框架内计算的,与实验数据非常吻合。根据我们的模拟,由于与反应坐标相关的蛋白质运动较慢,重酶的化学反应比轻酶的化学反应稍慢。因此,蛋白质动力学对化学反应速率的影响很小但可测量。EKIE是在变分过渡状态理论(VTST)的框架内计算的,与实验数据非常吻合。根据我们的模拟,由于与反应坐标相关的蛋白质运动较慢,重酶的化学反应比轻酶的化学反应稍慢。因此,蛋白质动力学对化学反应速率的影响很小但可测量。
更新日期:2017-12-29
中文翻译:

人嘌呤核苷磷酸化酶的酶动力学同位素效应的起源。
在这里,我们报告了重同位素标记对人嘌呤核苷磷酸化酶(hPNP)催化反应的影响的研究,以阐明其催化作用和酶动力学同位素作用(EKIE)的起源。使用量子力学和分子力学(QM / MM)分子动力学(MD)模拟,我们通过计算回交传输系数来研究hPNP酶的机理和动力学效应。获得了自由能表面(FES),它是化学和环境坐标的函数,以显示环境在化学反应中的作用。反应性和非反应性轨迹的分析使我们能够研究化学系统的几何,动态和电子变化。要特别注意环境在化学反应中所涉及的那些原子上产生的静电势。与化学系统相互作用的一些氨基酸残基和溶剂分子提供了一种特定的构型,该构型在静电上有利于反应进程,从而产生了反应性轨迹。EKIE是在变分过渡状态理论(VTST)的框架内计算的,与实验数据非常吻合。根据我们的模拟,由于与反应坐标相关的蛋白质运动较慢,重酶的化学反应比轻酶的化学反应稍慢。因此,蛋白质动力学对化学反应速率的影响很小但可测量。与化学系统相互作用的一些氨基酸残基和溶剂分子提供了一种特定的构型,该构型在静电上有利于反应进程,从而产生了反应性轨迹。EKIE是在变分过渡状态理论(VTST)的框架内计算的,与实验数据非常吻合。根据我们的模拟,由于与反应坐标相关的蛋白质运动较慢,重酶的化学反应比轻酶的化学反应稍慢。因此,蛋白质动力学对化学反应速率的影响很小但可测量。与化学系统相互作用的一些氨基酸残基和溶剂分子提供了一种特定的构型,该构型在静电上有利于反应进程,从而产生了反应性轨迹。EKIE是在变分过渡状态理论(VTST)的框架内计算的,与实验数据非常吻合。根据我们的模拟,由于与反应坐标相关的蛋白质运动较慢,重酶的化学反应比轻酶的化学反应稍慢。因此,蛋白质动力学对化学反应速率的影响很小但可测量。EKIE是在变分过渡状态理论(VTST)的框架内计算的,与实验数据非常吻合。根据我们的模拟,由于与反应坐标相关的蛋白质运动较慢,重酶的化学反应比轻酶的化学反应稍慢。因此,蛋白质动力学对化学反应速率的影响很小但可测量。EKIE是在变分过渡状态理论(VTST)的框架内计算的,与实验数据非常吻合。根据我们的模拟,由于与反应坐标相关的蛋白质运动较慢,重酶的化学反应比轻酶的化学反应稍慢。因此,蛋白质动力学对化学反应速率的影响很小但可测量。



























 京公网安备 11010802027423号
京公网安备 11010802027423号