中科院青岛能源所微生物分析方法进展二篇
(一)微生物组内部代谢互作示踪新技术
微生物组(Microbiome)是微生物在自然界中的存在形式,它们无处不在、无所不能,与我们每个人乃至海洋、土壤、大气的健康都息息相关。在微生物组的内部,不同种类的微生物之间存在着复杂、精妙的相互作用与影响,这一跨物种的细胞间代谢互作网络是群落功能和进化的基础。然而由于自然界中绝大部分微生物尚难以培养,而且细胞在纯培养状态往往无法体现其“原位”功能,因此“如何原位考察细胞间代谢互作”一直是微生物组方法学研究的重点与难点之一。近日,中科院青岛能源所与牛津大学合作发明了“反向拉曼标记技术”,可在单细胞水平上示踪微生物群落中细胞代谢互作,为微生物组原位功能研究提供了新思路与新平台。该工作近期发表于Analytical Chemistry。
单细胞拉曼成像与活细胞稳定同位素标记相结合的技术,能分辨群落内代谢特定底物的细胞,从而重建“食物链”的时空特征。然而自然界中的底物种类几近无限,许多人们感兴趣的底物无法或难以用稳定同位素标记,这大大限制了该技术的应用。针对上述瓶颈,中科院青岛能源所单细胞中心王允带领的研究小组打破了传统拉曼标记策略的思维定势,先使用13C标记的广谱碳源(如葡萄糖等)把群落中所有细胞都“喂饱”,再将碳源切换为未经标记的目标底物,然后通过观测单细胞拉曼光谱中目标底物代谢并替换细胞中13C标记化合物的过程,也就是测量13C拉曼峰反向漂移到12C位置的动态特征,从而分辨群落中特异性地代谢目标底物的细胞。同时,通过重水饲喂,观测2180 cm-1附近C-D峰的出现,以同时监测细胞的综合代谢活性。结合上述两种特性,可成像与重建细胞间的相互作用及底物传递过程(图1)。
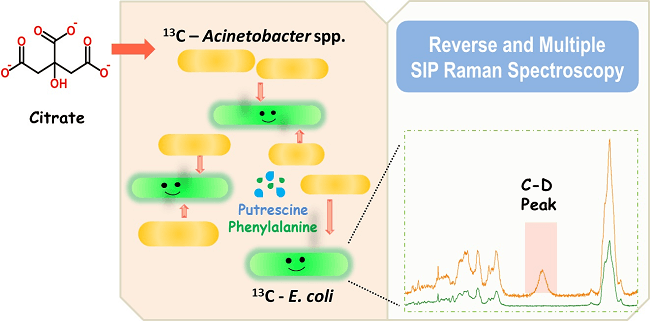
在此基础上,研究人员构建了由不动杆菌(Acinetobacter spp.)与大肠杆菌(E. coli)组成的模拟菌群,它们是人体、医院和自然界中均常见的机会致病菌,且其耐药性日益严重,已引起临床和微生物学者的关注。反向拉曼标记技术结合“碳源-重水”双标记实验发现,在柠檬酸作为唯一碳源的共培养体系中,不动杆菌可快速利用柠檬酸,并展现了较高的综合代谢活性。大肠杆菌“单身”时完全不能利用柠檬酸,但在与不动杆菌“同居”时却表现出了一定的综合代谢活性。代谢物组分析表明,在双方“同居”时,不动杆菌代谢柠檬酸的产物苯丙氨酸及腐胺可被大肠杆菌利用,含量虽不足以支撑后者的细胞分裂,但能保持其基础水平的综合代谢活性。因此该方法可在单细胞水平上解析与示踪群落中细胞之间的代谢互作与共生关系。
当前微生物组功能分析的主要工具,如元基因组、元转录组、元代谢组、稳定性同位素核酸探针(DNA/RNA-SIP)技术以及高通量培养技术等,通常难以揭示原位状态下群落中的各个成员在代谢功能上的互作关系。因此这一“反向拉曼标记技术”和双标记策略具备独特优势,有望作为一种共性手段与新工具,服务于人体健康、海洋生态、环境修复、生物发酵等广泛领域的微生物组研究。
上述工作由英国牛津大学工程系黄巍(点击查看介绍)和中科院青岛能源所单细胞中心徐健(点击查看介绍)联合主持,获得了基金委、中科院土壤微生物先导专项和中科院生物高通量检测分析服务网络(STS)等项目的支持。
该论文作者为:Yun Wang, Yizhi Song, Yifan Tao, Howbeer Muhamadali, Royston Goodacre, Ning-Yi Zhou, Gail M. Preston, Jian Xu, Wei E. Huang
原文(扫描或长按二维码,识别后直达原文页面):

http://pubs.acs.org/doi/abs/10.1021/acs.analchem.6b01602
Reverse and Multiple Stable Isotope Probing to Study Bacterial Metabolism and Interactions at the Single Cell Level
Anal. Chem., 2016, 88, 443-9450, DOI: 10.1021/acs.analchem.6b01602
(二)基于拉曼组的细菌耐药性快检技术
抗生素的滥用导致了耐药性的广泛传播。对抗耐药性不仅需要研发新型抗生素,还需要发展耐药性快检技术和监测体系,以提高现有抗生素使用的针对性和有效性,从而推迟与遏制耐药性的传播。因此,2016年08月26日国家卫生计生委等14部门联合印发的《遏制细菌耐药国家行动计划(2016-2020)》明确提出要“加强抗菌药物应用和耐药控制体系建设”和“完善抗菌药物应用和细菌耐药监测体系”。
自细菌发现至今,培养法仍是病原菌药敏试验的主流通用标准,但对于临床常见致病菌,培养法耗时长达24~48小时,难以揭示耐药机制,且对于难培养或生长缓慢的细菌无能为力。PCR和基因组测序技术可通过鉴定耐药基因来间接推测耐药的可能性,但它依赖于已知的耐药基因参照序列,故无法分析未知的耐药性,且通常无法定量检测,因此只能作为一种辅助手段。临床实践上为了指导“精准用药”,急需细菌耐药性及其耐药机制的直接、快速测量技术。临床需求与技术现状的矛盾如此紧迫和突出,以至于2016年9月8日美国NIH悬赏二千万美元,专门激励细菌耐药性快检技术的研发。
针对这些瓶颈,中科院青岛能源所单细胞中心提出了基于“拉曼组”的耐药性快检技术,证明通过高通量单细胞拉曼成像,能够不经培养、快速、定性、定量地表征细菌的药物应激性并区分其应激机制。该工作于10月19日在线发表于Scientific Reports。
首先,单细胞中心功能基因组团队徐健研究员及其博士生滕琳等提出了“拉曼组”的概念(图1)。“拉曼组”(Ramanome)是特定条件和时间点下,一个细菌细胞群体之单细胞拉曼光谱的集合。每个单细胞拉曼光谱由分别对应于一类化学键的上千个拉曼峰组成,反映的是特定细胞内化学物质的成分及含量的多维信息,而且其测量无需破坏细胞、不需要标记,通常仅需毫秒乃至数秒钟。因此,对于任一细菌群体,一个拉曼组相当于单细胞精度、可快速、低成本测定与监控的“代谢状态”或“代谢物组”,其变化可直接反映和表征其针对特定抗生素的敏感性和耐受性。而不同的抑菌机制会引起细胞内代谢物组的不同变化,因此拉曼组的变化还具有区分乃至识别各种药物应激机制的潜力。
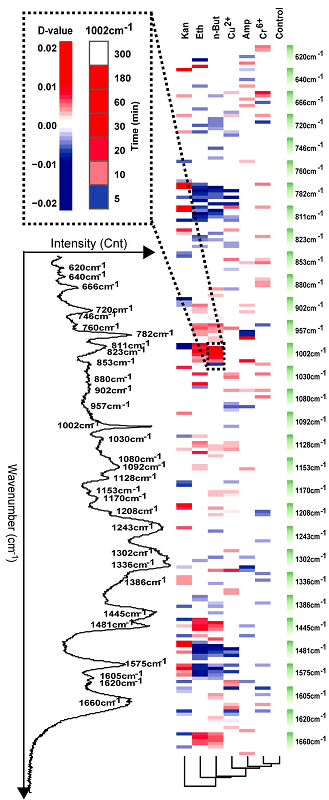
其次,研究人员以大肠杆菌为模式,通过单细胞拉曼光谱的高通量采集,结合多变量分析方法的创新,定量考察了抗生素、醇类、重金属等三类共六种不同类型化学药物在多个剂量、给药时间、细胞抗性条件下的拉曼组变化。研究发现,每一特定的药物及其剂量都对应着一种特定的拉曼组动态变化规律,而由31个特定拉曼峰组合而成的“耐药性拉曼识别码”可高度灵敏、可靠地识别细菌针对这些化学药品的应激反应,并区分与揭示细胞的耐药机制。此外,拉曼组还能准确测量给药过程中药敏性在细菌群体中的异质性变化,从而为研究细菌耐药性的起源和进化提供了有力工具。
最后,研究人员还证明拉曼组能够快速区分抗性细菌与非抗性细菌,因此它在抑菌药物筛选或耐药细菌筛选这两方面均具备成为一种新式平台技术的潜力。
拉曼组基于单细胞成像,不依赖于细菌的繁殖,因此通常能够在一个小时内完成细菌耐药性测量和机制区分,因此它在临床耐药性快检方面具有重要优势。通过系统构建各种主要病原菌和常用抗生素的拉曼组参照数据库,将能建立一个新型细菌耐药性表型组学技术平台,以服务耐药性快检,支撑临床精准用药。
上述工作由单细胞中心徐健实验室和英国牛津大学黄巍等合作完成,获得了科技部、基金委、中科院生物高通量检测分析服务网络(STS)项目的支持。
该论文作者为:Lin Teng, Xian Wang, Xiaojun Wang, Honglei Gou, Lihui Ren, Tingting Wang, Yun Wang, Yuetong Ji, Wei E. Huang, Jian Xu
原文(扫描或长按二维码,识别后直达原文页面):

http://www.nature.com/articles/srep34359
Label-free, rapid and quantitative phenotyping of stress response in E. coli via ramanome.
Sci. Rep., 2016, 6, 34359, DOI: 10.1038/srep34359
导师介绍
黄巍教授:http://www.x-mol.com/university/faculty/26785
徐健研究员:http://www.x-mol.com/university/faculty/26786
如果篇首注明了授权来源,任何转载需获得来源方的许可!如果篇首未特别注明出处,本文版权属于 X-MOL ( x-mol.com ), 未经许可,谢绝转载!




























 京公网安备 11010802027423号
京公网安备 11010802027423号