当前位置:
X-MOL 学术
›
J. Proteome Res.
›
论文详情
Our official English website, www.x-mol.net, welcomes your feedback! (Note: you will need to create a separate account there.)
Ultradeep Lysine Crotonylome Reveals the Crotonylation Enhancement on Both Histones and Nonhistone Proteins by SAHA Treatment
Journal of Proteome Research ( IF 4.4 ) Pub Date : 2017-09-19 00:00:00 , DOI: 10.1021/acs.jproteome.7b00380 Quan Wu 1 , Wenting Li 2 , Chi Wang 1 , Pingsheng Fan 3 , Lejie Cao 4 , Zhiwei Wu 1 , Fengsong Wang 5
Journal of Proteome Research ( IF 4.4 ) Pub Date : 2017-09-19 00:00:00 , DOI: 10.1021/acs.jproteome.7b00380 Quan Wu 1 , Wenting Li 2 , Chi Wang 1 , Pingsheng Fan 3 , Lejie Cao 4 , Zhiwei Wu 1 , Fengsong Wang 5
Affiliation
|
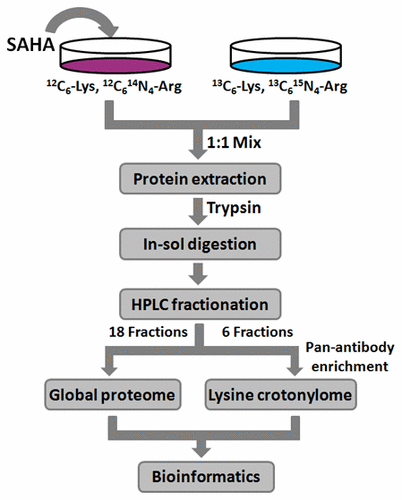
Lysine crotonylation is a newly discovered protein post-translational modification and was reported to share transferases and deacylases with lysine acetylation. The acetyltransferase p300 was reported to also contain crotonyltransferase activity, and class I histone deacetylases were demonstrated to be the major histone decrotonylases. However, the decrotonylases for nonhistone proteins are unclear. Moreover, because of the lack of high-quality pan-antibodies, large-scale analysis of crotonylome still remains a challenge. In this work, we comprehensively studied lysine crotonylome on both histones and nonhistone proteins upon SAHA treatment and dramatically identified 10 163 lysine crotonylation sites in A549 cells. This is the first identification of tens of thousands of lysine crotonylation sites and also the largest lysine crotonylome data set up to now. Moreover, a parallel-reaction-monitoring-based experiment was performed for validation, which presented highly consistent results with the SILAC experiments. By intensive bioinformatic analysis, it was found that lysine crotonylation participates in a wide range of biological functions and processes. More importantly, it was revealed that both the crotonylation and acetylation levels of most core histones sites and a number of nonhistone proteins as well as some known substrates of class IIa and IIb HDACs were up-regulated after SAHA treatment. These results suggest that SAHA may have decrotonylation inhibitory activities on both histones and nonhistone proteins by inhibiting HDACs.
中文翻译:

超深的赖氨酸巴豆基因组通过SAHA处理揭示了组蛋白和非组蛋白的巴豆酰化增强
赖氨酸巴豆酰化是一种新发现的蛋白翻译后修饰,据报道与赖氨酸乙酰化共享转移酶和脱酰基酶。据报道,乙酰基转移酶p300也具有巴豆酰基转移酶活性,并且I类组蛋白脱乙酰基酶被证明是主要的组蛋白巴豆酰基化酶。但是,尚不清楚非组蛋白蛋白的巴豆酰化酶。而且,由于缺乏高质量的泛抗体,对巴豆组的大规模分析仍然是一个挑战。在这项工作中,我们对SAHA处理后的组蛋白和非组蛋白的赖氨酸巴豆基蛋白质组进行了全面研究,并显着鉴定了A549细胞中的10 163个赖氨酸巴豆基化位点。这是对数万个赖氨酸巴豆酰化位点的首次鉴定,也是迄今为止建立的最大的赖氨酸巴豆酰化酶数据。此外,进行了基于平行反应监测的实验以进行验证,该实验与SILAC实验呈现出高度一致的结果。通过深入的生物信息学分析,发现赖氨酸巴豆酰化参与了广泛的生物学功能和过程。更重要的是,发现在SAHA处理后,大多数核心组蛋白位点和许多非组蛋白蛋白以及IIa和IIb类HDAC的某些已知底物的巴豆酰化和乙酰化水平均被上调。这些结果表明,SAHA可能通过抑制HDAC而对组蛋白和非组蛋白具有去甲酰基化抑制活性。进行了基于平行反应监测的实验以进行验证,该实验与SILAC实验呈现出高度一致的结果。通过深入的生物信息学分析,发现赖氨酸巴豆酰化参与了广泛的生物学功能和过程。更重要的是,发现在SAHA处理后,大多数核心组蛋白位点和许多非组蛋白蛋白以及IIa和IIb类HDAC的某些已知底物的巴豆酰化和乙酰化水平均被上调。这些结果表明,SAHA可能通过抑制HDAC而对组蛋白和非组蛋白具有去甲酰基化抑制活性。进行了基于平行反应监测的实验以进行验证,该实验与SILAC实验呈现出高度一致的结果。通过深入的生物信息学分析,发现赖氨酸巴豆酰化参与了广泛的生物学功能和过程。更重要的是,发现在SAHA处理后,大多数核心组蛋白位点和许多非组蛋白蛋白以及IIa和IIb类HDAC的某些已知底物的巴豆酰化和乙酰化水平均被上调。这些结果表明,SAHA可能通过抑制HDAC而对组蛋白和非组蛋白具有去甲酰基化抑制活性。通过深入的生物信息学分析,发现赖氨酸巴豆酰化参与了广泛的生物学功能和过程。更重要的是,发现在SAHA处理后,大多数核心组蛋白位点和许多非组蛋白蛋白以及IIa和IIb类HDAC的某些已知底物的巴豆酰化和乙酰化水平均被上调。这些结果表明,SAHA可能通过抑制HDAC而对组蛋白和非组蛋白具有去甲酰基化抑制活性。通过深入的生物信息学分析,发现赖氨酸巴豆酰化参与了广泛的生物学功能和过程。更重要的是,发现在SAHA处理后,大多数核心组蛋白位点和许多非组蛋白蛋白以及IIa和IIb类HDAC的某些已知底物的巴豆酰化和乙酰化水平均被上调。这些结果表明,SAHA可能通过抑制HDAC而对组蛋白和非组蛋白具有去甲酰基化抑制活性。结果表明,SAHA处理后,大多数核心组蛋白位点的巴豆酰化和乙酰化水平以及许多非组蛋白和某些已知的IIa和IIb类HDAC底物均被上调。这些结果表明,SAHA可能通过抑制HDAC而对组蛋白和非组蛋白具有去甲酰基化抑制活性。结果表明,SAHA处理后,大多数核心组蛋白位点的巴豆酰化和乙酰化水平以及许多非组蛋白和某些已知的IIa和IIb类HDAC底物均被上调。这些结果表明,SAHA可能通过抑制HDAC而对组蛋白和非组蛋白具有去甲酰基化抑制活性。
更新日期:2017-09-19
中文翻译:

超深的赖氨酸巴豆基因组通过SAHA处理揭示了组蛋白和非组蛋白的巴豆酰化增强
赖氨酸巴豆酰化是一种新发现的蛋白翻译后修饰,据报道与赖氨酸乙酰化共享转移酶和脱酰基酶。据报道,乙酰基转移酶p300也具有巴豆酰基转移酶活性,并且I类组蛋白脱乙酰基酶被证明是主要的组蛋白巴豆酰基化酶。但是,尚不清楚非组蛋白蛋白的巴豆酰化酶。而且,由于缺乏高质量的泛抗体,对巴豆组的大规模分析仍然是一个挑战。在这项工作中,我们对SAHA处理后的组蛋白和非组蛋白的赖氨酸巴豆基蛋白质组进行了全面研究,并显着鉴定了A549细胞中的10 163个赖氨酸巴豆基化位点。这是对数万个赖氨酸巴豆酰化位点的首次鉴定,也是迄今为止建立的最大的赖氨酸巴豆酰化酶数据。此外,进行了基于平行反应监测的实验以进行验证,该实验与SILAC实验呈现出高度一致的结果。通过深入的生物信息学分析,发现赖氨酸巴豆酰化参与了广泛的生物学功能和过程。更重要的是,发现在SAHA处理后,大多数核心组蛋白位点和许多非组蛋白蛋白以及IIa和IIb类HDAC的某些已知底物的巴豆酰化和乙酰化水平均被上调。这些结果表明,SAHA可能通过抑制HDAC而对组蛋白和非组蛋白具有去甲酰基化抑制活性。进行了基于平行反应监测的实验以进行验证,该实验与SILAC实验呈现出高度一致的结果。通过深入的生物信息学分析,发现赖氨酸巴豆酰化参与了广泛的生物学功能和过程。更重要的是,发现在SAHA处理后,大多数核心组蛋白位点和许多非组蛋白蛋白以及IIa和IIb类HDAC的某些已知底物的巴豆酰化和乙酰化水平均被上调。这些结果表明,SAHA可能通过抑制HDAC而对组蛋白和非组蛋白具有去甲酰基化抑制活性。进行了基于平行反应监测的实验以进行验证,该实验与SILAC实验呈现出高度一致的结果。通过深入的生物信息学分析,发现赖氨酸巴豆酰化参与了广泛的生物学功能和过程。更重要的是,发现在SAHA处理后,大多数核心组蛋白位点和许多非组蛋白蛋白以及IIa和IIb类HDAC的某些已知底物的巴豆酰化和乙酰化水平均被上调。这些结果表明,SAHA可能通过抑制HDAC而对组蛋白和非组蛋白具有去甲酰基化抑制活性。通过深入的生物信息学分析,发现赖氨酸巴豆酰化参与了广泛的生物学功能和过程。更重要的是,发现在SAHA处理后,大多数核心组蛋白位点和许多非组蛋白蛋白以及IIa和IIb类HDAC的某些已知底物的巴豆酰化和乙酰化水平均被上调。这些结果表明,SAHA可能通过抑制HDAC而对组蛋白和非组蛋白具有去甲酰基化抑制活性。通过深入的生物信息学分析,发现赖氨酸巴豆酰化参与了广泛的生物学功能和过程。更重要的是,发现在SAHA处理后,大多数核心组蛋白位点和许多非组蛋白蛋白以及IIa和IIb类HDAC的某些已知底物的巴豆酰化和乙酰化水平均被上调。这些结果表明,SAHA可能通过抑制HDAC而对组蛋白和非组蛋白具有去甲酰基化抑制活性。结果表明,SAHA处理后,大多数核心组蛋白位点的巴豆酰化和乙酰化水平以及许多非组蛋白和某些已知的IIa和IIb类HDAC底物均被上调。这些结果表明,SAHA可能通过抑制HDAC而对组蛋白和非组蛋白具有去甲酰基化抑制活性。结果表明,SAHA处理后,大多数核心组蛋白位点的巴豆酰化和乙酰化水平以及许多非组蛋白和某些已知的IIa和IIb类HDAC底物均被上调。这些结果表明,SAHA可能通过抑制HDAC而对组蛋白和非组蛋白具有去甲酰基化抑制活性。



























 京公网安备 11010802027423号
京公网安备 11010802027423号